| 登録情報 | データベース: PDB / ID: 1o1v
|
|---|
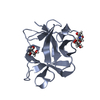
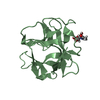
| タイトル | Human Ileal Lipid-Binding Protein (ILBP) in Complex with Cholyltaurine |
|---|
 要素 要素 | Gastrotropin |
|---|
 キーワード キーワード | LIPID BINDING PROTEIN / BETA CLAM STRUCTURE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / Triglyceride catabolism / fatty acid transport / Recycling of bile acids and salts / fatty acid binding / lipid metabolic process / negative regulation of cell population proliferation / lipid binding / nucleus / membrane ...NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / Triglyceride catabolism / fatty acid transport / Recycling of bile acids and salts / fatty acid binding / lipid metabolic process / negative regulation of cell population proliferation / lipid binding / nucleus / membrane / cytoplasm / cytosol類似検索 - 分子機能 Lipocalin / cytosolic fatty-acid binding protein family / Cytosolic fatty-acid binding proteins signature. / Intracellular lipid binding protein / Cytosolic fatty-acid binding / Calycin beta-barrel core domain / Calycin / Lipocalin / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / distance geometry (DIANA), molecular dynamics (SYBYL), energy minimization (SYBYL) |
|---|
 データ登録者 データ登録者 | Kurz, M. / Brachvogel, V. / Matter, H. / Stengelin, S. / Thuering, H. / Kramer, W. |
|---|
 引用 引用 |  ジャーナル: Proteins / 年: 2003 ジャーナル: Proteins / 年: 2003
タイトル: Insights into the bile acid transportation system: the human ileal lipid-binding protein-cholyltaurine complex and its comparison with homologous structures.
著者: Kurz, M. / Brachvogel, V. / Matter, H. / Stengelin, S. / Thuering, H. / Kramer, W. |
|---|
| 履歴 | | 登録 | 2003年2月10日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2003年2月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月26日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年2月23日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.4 | 2023年12月27日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Proteins / 年: 2003
ジャーナル: Proteins / 年: 2003 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1o1v.cif.gz
1o1v.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1o1v.ent.gz
pdb1o1v.ent.gz PDB形式
PDB形式 1o1v.json.gz
1o1v.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1o1v_validation.pdf.gz
1o1v_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1o1v_full_validation.pdf.gz
1o1v_full_validation.pdf.gz 1o1v_validation.xml.gz
1o1v_validation.xml.gz 1o1v_validation.cif.gz
1o1v_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/o1/1o1v
https://data.pdbj.org/pub/pdb/validation_reports/o1/1o1v ftp://data.pdbj.org/pub/pdb/validation_reports/o1/1o1v
ftp://data.pdbj.org/pub/pdb/validation_reports/o1/1o1v リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: FABP6 OR ILLBP OR ILBP / プラスミド: pHUGA1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: FABP6 OR ILLBP OR ILBP / プラスミド: pHUGA1 / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj






 HSQC
HSQC