+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1m8a | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Human MIP-3alpha/CCL20 | ||||||
 要素 要素 | Small inducible cytokine A20 | ||||||
 キーワード キーワード | CYTOKINE / CC-chemokine / IL-8 type dimer | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thymocyte migration / CCR6 chemokine receptor binding / chemokine activity / Chemokine receptors bind chemokines / Interleukin-10 signaling / positive regulation of T cell migration / T cell migration / cell chemotaxis / calcium-mediated signaling / chemotaxis ...thymocyte migration / CCR6 chemokine receptor binding / chemokine activity / Chemokine receptors bind chemokines / Interleukin-10 signaling / positive regulation of T cell migration / T cell migration / cell chemotaxis / calcium-mediated signaling / chemotaxis / cell-cell signaling / G alpha (i) signalling events / defense response to bacterium / immune response / inflammatory response / signal transduction / extracellular space / extracellular region 類似検索 - 分子機能 | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å | ||||||
 データ登録者 データ登録者 | Hoover, D.M. / Boulegue, C. / Yang, D. / Oppenheim, J.J. / Tucker, K. / Lu, W. / Lubkowski, J. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2002 ジャーナル: J.Biol.Chem. / 年: 2002タイトル: The structure of human macrophage inflammatory protein-3alpha /CCL20. Linking antimicrobial and CC chemokine receptor-6-binding activities with human beta-defensins 著者: Hoover, D.M. / Boulegue, C. / Yang, D. / Oppenheim, J.J. / Tucker, K. / Lu, W. / Lubkowski, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1m8a.cif.gz 1m8a.cif.gz | 43.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1m8a.ent.gz pdb1m8a.ent.gz | 30.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1m8a.json.gz 1m8a.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1m8a_validation.pdf.gz 1m8a_validation.pdf.gz | 453.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1m8a_full_validation.pdf.gz 1m8a_full_validation.pdf.gz | 456.6 KB | 表示 | |
| XML形式データ |  1m8a_validation.xml.gz 1m8a_validation.xml.gz | 10 KB | 表示 | |
| CIF形式データ |  1m8a_validation.cif.gz 1m8a_validation.cif.gz | 13.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/m8/1m8a https://data.pdbj.org/pub/pdb/validation_reports/m8/1m8a ftp://data.pdbj.org/pub/pdb/validation_reports/m8/1m8a ftp://data.pdbj.org/pub/pdb/validation_reports/m8/1m8a | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1eqtS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 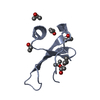
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | The molecule is likely a monomer |
- 要素
要素
| #1: タンパク質 | 分子量: 8043.568 Da / 分子数: 2 / 由来タイプ: 合成 詳細: THE PROTEIN WAS SYNTHESIZED USING Machine-assisted stepwise solid phase peptide synthesis on an Applied Biosystems 433A synthesizer using a custom-designed program tailored from the published ...詳細: THE PROTEIN WAS SYNTHESIZED USING Machine-assisted stepwise solid phase peptide synthesis on an Applied Biosystems 433A synthesizer using a custom-designed program tailored from the published in situ neutralization/HBTU activation protocol for Boc chemistry (Schnolzer, M. et al. (1992) J.Pept.Protein Res. 40, 180-193). IT IS NATURALLY FOUND IN HOMO SAPIENS (HUMAN). 参照: UniProt: P78556 #2: 化合物 | ChemComp-IPA / #3: 水 | ChemComp-HOH / | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.69 Å3/Da / 溶媒含有率: 54.3 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 285 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: isopropanol, sodium citrate, N-(2-hydroxyethyl)piperazine-N'-(2-ethanesulfonic acid), pH 7.5, VAPOR DIFFUSION, HANGING DROP, temperature 285K | ||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 12 ℃ | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS NSLS  / ビームライン: X9B / 波長: 0.98 Å / ビームライン: X9B / 波長: 0.98 Å |
| 検出器 | タイプ: ADSC QUANTUM 4 / 検出器: CCD / 日付: 2002年1月25日 / 詳細: mirrors |
| 放射 | モノクロメーター: si crystal / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.98 Å / 相対比: 1 |
| 反射 | 解像度: 1.7→20 Å / Num. all: 21953 / Num. obs: 21953 / % possible obs: 97.5 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 4.5 % / Rmerge(I) obs: 0.067 / Net I/σ(I): 23.8 |
| 反射 シェル | 解像度: 1.7→1.76 Å / Rmerge(I) obs: 0.623 / Mean I/σ(I) obs: 1.9 / % possible all: 96.8 |
| 反射 | *PLUS 最高解像度: 1.7 Å / 最低解像度: 20 Å / Num. measured all: 99101 |
| 反射 シェル | *PLUS 最高解像度: 1.7 Å / % possible obs: 96.8 % |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1EQT 解像度: 1.7→15 Å / Num. parameters: 4979 / Num. restraintsaints: 4435 Isotropic thermal model: anisotropic for sulfur, isotropic otherwise 交差検証法: FREE R / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber 詳細: ANISOTROPIC SCALING APPLIED BY THE METHOD OF PARKIN, MOEZZI & HOPE, J.APPL.CRYST.28(1995)53-56
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER, J.MOL.BIOL.91(1973)201-228 | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 27.7 Å2 | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 5 / Occupancy sum hydrogen: 0 / Occupancy sum non hydrogen: 1175 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→15 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL / バージョン: 97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 15 Å / % reflection Rfree: 4 % / Rfactor all: 0.19 / Rfactor Rfree: 0.238 / Rfactor Rwork: 0.19 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS タイプ: s_plane_restr / Dev ideal: 0.028 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





