+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ltx | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of Rab Escort Protein-1 in complex with Rab geranylgeranyl transferase and isoprenoid | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | Transferase/Protein binding / RAB PRENYLATION / PRENYLTRANSFERASE / LUCINE-RICH REPEATS / POST-TRANSLATIONAL MODIFICATION / Transferase-Protein binding COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RAB GEFs exchange GTP for GDP on RABs / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / protein geranylgeranyltransferase type II / RAB geranylgeranylation / Rab-protein geranylgeranyltransferase complex / isoprenoid binding / Rab geranylgeranyltransferase activity / protein geranylgeranylation / protein targeting to membrane / GDP-dissociation inhibitor activity ...RAB GEFs exchange GTP for GDP on RABs / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / protein geranylgeranyltransferase type II / RAB geranylgeranylation / Rab-protein geranylgeranyltransferase complex / isoprenoid binding / Rab geranylgeranyltransferase activity / protein geranylgeranylation / protein targeting to membrane / GDP-dissociation inhibitor activity / small GTPase-mediated signal transduction / blood vessel development / endoplasmic reticulum to Golgi vesicle-mediated transport / vesicle-mediated transport / GTPase activator activity / intracellular protein transport / small GTPase binding / protein-containing complex binding / zinc ion binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å | ||||||
 データ登録者 データ登録者 | Pylypenko, O. / Rak, A. / Reents, R. / Niculae, A. / Thoma, N.H. / Waldmann, H. / Schlichting, I. / Goody, R.S. / Alexandrov, K. | ||||||
 引用 引用 |  ジャーナル: Mol.Cell / 年: 2003 ジャーナル: Mol.Cell / 年: 2003タイトル: Structure of Rab Escort Protein-1 in Complex with Rab Geranylgeranyltransferase 著者: Pylypenko, O. / Rak, A. / Reents, R. / Niculae, A. / Sidorovitch, V. / Cioaca, M.D. / Bessolitsyna, E. / Thoma, N.H. / Waldmann, H. / Schlichting, I. / Goody, R.S. / Alexandrov, K. #1:  ジャーナル: J.STRUCT.BIOL. / 年: 2001 ジャーナル: J.STRUCT.BIOL. / 年: 2001タイトル: Crystallization and Preliminary X-ray Diffraction Analysis of the Rab Escort Protein-1 in Complex with Rab Geranylgeranyltransferase 著者: Rak, A. / Reents, R. / Pylypenko, O. / Niculae, A. / Sidorovitch, V. / Thoma, N.H. / Waldmann, H. / Schlichting, I. / Goody, R.S. / Alexandrov, K. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ltx.cif.gz 1ltx.cif.gz | 281.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ltx.ent.gz pdb1ltx.ent.gz | 222.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ltx.json.gz 1ltx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1ltx_validation.pdf.gz 1ltx_validation.pdf.gz | 478.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1ltx_full_validation.pdf.gz 1ltx_full_validation.pdf.gz | 540.9 KB | 表示 | |
| XML形式データ |  1ltx_validation.xml.gz 1ltx_validation.xml.gz | 54.3 KB | 表示 | |
| CIF形式データ |  1ltx_validation.cif.gz 1ltx_validation.cif.gz | 73.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lt/1ltx https://data.pdbj.org/pub/pdb/validation_reports/lt/1ltx ftp://data.pdbj.org/pub/pdb/validation_reports/lt/1ltx ftp://data.pdbj.org/pub/pdb/validation_reports/lt/1ltx | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1dceS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-RAB GERANYLGERANYLTRANSFERASE ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 64981.156 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   参照: UniProt: Q08602, 転移酵素; メチル基以外のアルキル基またはアリル基を移すもの; - |
|---|---|
| #2: タンパク質 | 分子量: 36892.160 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   参照: UniProt: Q08603, 転移酵素; メチル基以外のアルキル基またはアリル基を移すもの; - |
-タンパク質 / タンパク質・ペプチド , 2種, 2分子 RP
| #3: タンパク質 | 分子量: 72594.219 Da / 分子数: 1 / Mutation: A473T, G483A, Q231K / 由来タイプ: 組換発現 / 由来: (組換発現)  発現宿主:  参照: UniProt: P37727 |
|---|---|
| #4: タンパク質・ペプチド | 分子量: 302.326 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: PUTATIVE PEPTIDE. |
-非ポリマー , 4種, 106分子 

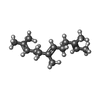




| #5: 化合物 | ChemComp-ZN / |
|---|---|
| #6: 化合物 | ChemComp-CL / |
| #7: 化合物 | ChemComp-FAR / |
| #8: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.05 Å3/Da / 溶媒含有率: 59.65 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.2 詳細: 20% PEG 3350, 100mM KSCN, 100mM Namalonate, pH 7.2, VAPOR DIFFUSION, HANGING DROP, temperature 298.0K | |||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法詳細: used microseeding, Rak, A., (2001) J.STRUCT.BIOL., 136, 158. | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID14-1 / 波長: 0.9393 Å / ビームライン: ID14-1 / 波長: 0.9393 Å |
| 検出器 | タイプ: ADSC QUANTUM 4 / 検出器: CCD |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9393 Å / 相対比: 1 |
| 反射 | 解像度: 2.7→19.7 Å / Num. all: 54362 / Num. obs: 54362 / % possible obs: 94.5 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 3.6 % / Biso Wilson estimate: 53.8 Å2 / Rsym value: 0.094 / Net I/σ(I): 9.5 |
| 反射 シェル | 解像度: 2.7→2.8 Å / 冗長度: 2.7 % / Mean I/σ(I) obs: 3.25 / Num. unique all: 3716 / Rsym value: 0.364 / % possible all: 62.5 |
| 反射 | *PLUS Rmerge(I) obs: 0.094 |
| 反射 シェル | *PLUS % possible obs: 62.5 % / Num. unique obs: 3716 / Rmerge(I) obs: 0.364 / Mean I/σ(I) obs: 3.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1DCE 解像度: 2.7→19.74 Å / Rfactor Rfree error: 0.005 / Data cutoff high absF: 2824477.81 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 31.0783 Å2 / ksol: 0.342551 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 52.4 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.7→19.74 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.7→2.87 Å / Rfactor Rfree error: 0.022 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.8 Å / % reflection Rfree: 5 % / Rfactor Rfree: 0.274 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS 最高解像度: 2.7 Å / 最低解像度: 2.8 Å |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




