+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1llp | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | LIGNIN PEROXIDASE (ISOZYME H2) PI 4.15 | |||||||||
 要素 要素 | LIGNIN PEROXIDASE | |||||||||
 キーワード キーワード | OXIDOREDUCTASE / HEME PROTEIN / GLYCO PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lignin peroxidase / diarylpropane peroxidase activity / lignin catabolic process / response to reactive oxygen species / hydrogen peroxide catabolic process / cellular response to oxidative stress / heme binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Phanerochaete chrysosporium (菌類) Phanerochaete chrysosporium (菌類) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.7 Å シンクロトロン / 解像度: 1.7 Å | |||||||||
 データ登録者 データ登録者 | Choinowski, T.H. / Piontek, K. / Glumoff, T. | |||||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1999 ジャーナル: J.Mol.Biol. / 年: 1999タイトル: The crystal structure of lignin peroxidase at 1.70 A resolution reveals a hydroxy group on the cbeta of tryptophan 171: a novel radical site formed during the redox cycle. 著者: Choinowski, T. / Blodig, W. / Winterhalter, K.H. / Piontek, K. #1:  ジャーナル: Bioorg.Med.Chem. / 年: 1994 ジャーナル: Bioorg.Med.Chem. / 年: 1994タイトル: Do Carbohydrates Play a Role in the Lignin Peroxidase Cycle? Redox Catalysis in the Endergonic Region of the Driving Force 著者: Schoemaker, H.E. / Lundell, T.K. / Floris, R. / Glumoff, T. / Winterhalter, K.H. / Piontek, K. #2:  ジャーナル: Fems Microbiol.Rev. / 年: 1994 ジャーナル: Fems Microbiol.Rev. / 年: 1994タイトル: The Oxidation of Veratryl Alcohol, Dimeric Lignin Models and Lignin by Lignin Peroxidase: The Redox Cycle Revisited 著者: Schoemaker, H.E. / Lundell, T.K. / Hatakka, A.I. / Piontek, K. #3:  ジャーナル: FEBS Lett. / 年: 1993 ジャーナル: FEBS Lett. / 年: 1993タイトル: Low Ph Crystal Structure of Glycosylated Lignin Peroxidase from Phanerochaete Chrysosporium at 2.5 Angstrom Resolution 著者: Piontek, K. / Glumoff, T. / Winterhalter, K. | |||||||||
| 履歴 |
| |||||||||
| Remark 700 | SHEET SHEET SHEET_ID: A; DETERMINATION METHOD: DSSP; SHORT ANTIPARALLEL BETA-SHEET. SHEET_ID: B; ...SHEET SHEET SHEET_ID: A; DETERMINATION METHOD: DSSP; SHORT ANTIPARALLEL BETA-SHEET. SHEET_ID: B; SHORT ANTIPARALLEL BETA-SHEET. SHEET_ID: C; SHORT ANTIPARALLEL BETA-SHEET. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1llp.cif.gz 1llp.cif.gz | 91.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1llp.ent.gz pdb1llp.ent.gz | 67 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1llp.json.gz 1llp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1llp_validation.pdf.gz 1llp_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1llp_full_validation.pdf.gz 1llp_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  1llp_validation.xml.gz 1llp_validation.xml.gz | 22.9 KB | 表示 | |
| CIF形式データ |  1llp_validation.cif.gz 1llp_validation.cif.gz | 32.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ll/1llp https://data.pdbj.org/pub/pdb/validation_reports/ll/1llp ftp://data.pdbj.org/pub/pdb/validation_reports/ll/1llp ftp://data.pdbj.org/pub/pdb/validation_reports/ll/1llp | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 36411.648 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Phanerochaete chrysosporium (菌類) / 株: BKM-F1267 Phanerochaete chrysosporium (菌類) / 株: BKM-F1267参照: UniProt: P49012, 酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ |
|---|
-糖 , 3種, 3分子 


| #2: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |
|---|---|
| #3: 糖 | ChemComp-MAN / |
| #4: 糖 | ChemComp-A2G / |
-非ポリマー , 4種, 374分子 
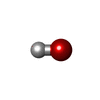





| #5: 化合物 | | #6: 化合物 | ChemComp-OH / | #7: 化合物 | ChemComp-HEM / | #8: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.31 Å3/Da / 溶媒含有率: 62.83 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 4 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 277 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: X31 / 波長: 0.92 Å / ビームライン: X31 / 波長: 0.92 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1993年11月17日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.92 Å / 相対比: 1 |
| 反射 | 解像度: 1.7→10 Å / Num. obs: 52732 / % possible obs: 98.3 % / Observed criterion σ(I): 0 / 冗長度: 5 % / Rmerge(I) obs: 0.083 |
| 反射 | *PLUS Num. measured all: 243426 / Rmerge(I) obs: 0.083 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.7→10 Å / σ(F): 2 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 16.62 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / 分類: refinement X-PLOR / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Num. reflection obs: 52504 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS Biso mean: 13.6 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj












