+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1k1a | ||||||
|---|---|---|---|---|---|---|---|
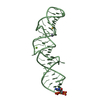
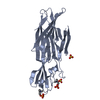
| タイトル | Crystal structure of the ankyrin repeat domain of Bcl-3: a unique member of the IkappaB protein family | ||||||
 要素 要素 | B-cell lymphoma 3-encoded protein | ||||||
 キーワード キーワード | TRANSCRIPTION / Bcl-3 / NF-kappaB transcription factors / IkappaB proteins | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Bcl3-Bcl10 complex / follicular dendritic cell differentiation / Bcl3/NF-kappaB2 complex / regulation of DNA binding / marginal zone B cell differentiation / regulation of non-canonical NF-kappaB signal transduction / humoral immune response mediated by circulating immunoglobulin / negative regulation of interleukin-8 production / T cell apoptotic process / negative regulation of receptor signaling pathway via JAK-STAT ...Bcl3-Bcl10 complex / follicular dendritic cell differentiation / Bcl3/NF-kappaB2 complex / regulation of DNA binding / marginal zone B cell differentiation / regulation of non-canonical NF-kappaB signal transduction / humoral immune response mediated by circulating immunoglobulin / negative regulation of interleukin-8 production / T cell apoptotic process / negative regulation of receptor signaling pathway via JAK-STAT / negative regulation of T cell apoptotic process / antimicrobial humoral response / germinal center formation / response to UV-C / T-helper 2 cell differentiation / T-helper 1 type immune response / negative regulation of NF-kappaB transcription factor activity / defense response to protozoan / positive regulation of interleukin-10 production / negative regulation of tumor necrosis factor production / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / canonical NF-kappaB signal transduction / spleen development / extracellular matrix organization / positive regulation of translation / DNA damage response, signal transduction by p53 class mediator / response to virus / histone deacetylase binding / positive regulation of type II interferon production / protein import into nucleus / transcription corepressor activity / midbody / protein-macromolecule adaptor activity / regulation of apoptotic process / DNA-binding transcription factor binding / transcription coactivator activity / defense response to bacterium / ciliary basal body / negative regulation of DNA-templated transcription / intracellular membrane-bounded organelle / DNA damage response / negative regulation of apoptotic process / positive regulation of DNA-templated transcription / perinuclear region of cytoplasm / positive regulation of transcription by RNA polymerase II / protein-containing complex / nucleoplasm / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.86 Å 分子置換 / 解像度: 1.86 Å | ||||||
 データ登録者 データ登録者 | Michel, F. / Soler-Lopez, M. / Petosa, C. / Cramer, P. / Siebenlist, U. / Mueller, C.W. | ||||||
 引用 引用 |  ジャーナル: EMBO J. / 年: 2001 ジャーナル: EMBO J. / 年: 2001タイトル: Crystal structure of the ankyrin repeat domain of Bcl-3: a unique member of the IkappaB protein family. 著者: Michel, F. / Soler-Lopez, M. / Petosa, C. / Cramer, P. / Siebenlist, U. / Muller, C.W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1k1a.cif.gz 1k1a.cif.gz | 59.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1k1a.ent.gz pdb1k1a.ent.gz | 43.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1k1a.json.gz 1k1a.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1k1a_validation.pdf.gz 1k1a_validation.pdf.gz | 373.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1k1a_full_validation.pdf.gz 1k1a_full_validation.pdf.gz | 379.2 KB | 表示 | |
| XML形式データ |  1k1a_validation.xml.gz 1k1a_validation.xml.gz | 6.9 KB | 表示 | |
| CIF形式データ |  1k1a_validation.cif.gz 1k1a_validation.cif.gz | 11 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/k1/1k1a https://data.pdbj.org/pub/pdb/validation_reports/k1/1k1a ftp://data.pdbj.org/pub/pdb/validation_reports/k1/1k1a ftp://data.pdbj.org/pub/pdb/validation_reports/k1/1k1a | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 25829.451 Da / 分子数: 1 / 断片: ankyrin repeat domain / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:  |
|---|---|
| #2: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.99 Å3/Da / 溶媒含有率: 38.12 % | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6 詳細: PEG 6000, MES, pH 6.0, VAPOR DIFFUSION, HANGING DROP, temperature 293K | ||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 20 ℃ / pH: 5.8 | ||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID14-1 / 波長: 0.934 Å / ビームライン: ID14-1 / 波長: 0.934 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 2000年10月28日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.934 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→40 Å / Num. all: 17021 / Num. obs: 17021 / % possible obs: 89.8 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 2.7 % / Rsym value: 0.072 / Net I/σ(I): 10.5 |
| 反射 シェル | 解像度: 1.8→1.86 Å / 冗長度: 1.73 % / Mean I/σ(I) obs: 1.65 / Num. unique all: 1479 / Rsym value: 0.697 / % possible all: 77.8 |
| 反射 | *PLUS 最低解像度: 40 Å / 冗長度: 2.71 % / Num. measured all: 46088 / Rmerge(I) obs: 0.072 |
| 反射 シェル | *PLUS % possible obs: 77.8 % / Rmerge(I) obs: 0.697 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1ikn 解像度: 1.86→20 Å / SU B: 3.68115 / SU ML: 0.11055 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.18488 / ESU R Free: 0.15154 / 立体化学のターゲット値: Engh & Huber 詳細: RESIDUES SER178,VAL179,LEU182,GLN251,ARG269,SER307 PRESENT DOUBLE CONFORMATIONS.
| ||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 21.105 Å2
| ||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.86→20 Å
| ||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: REFMAC / 分類: refinement | ||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 20 Å / σ(F): 0 / % reflection Rfree: 5 % / Rfactor obs: 0.199 | ||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


