+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1jkp | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Testing the Water-Mediated HIN Recombinase DNA Recognition by Systematic Mutations | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA / WATER-MEDIATED RECOGNITION / PROTEIN-DNA COMPLEX / HIN RECOMBINASE / T11G MUTANT / DNA BINDING PROTEIN-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Chiu, T.K. / Sohn, C. / Johnson, R.C. / Dickerson, R.E. | ||||||
 引用 引用 |  ジャーナル: EMBO J. / 年: 2002 ジャーナル: EMBO J. / 年: 2002タイトル: Testing water-mediated DNA recognition by the Hin recombinase. 著者: Chiu, T.K. / Sohn, C. / Dickerson, R.E. / Johnson, R.C. #1:  ジャーナル: Thesis / 年: 2001 ジャーナル: Thesis / 年: 2001タイトル: How Hin Recombinase, FIS and Cations Bind DNA. Chapter 4. Water-Mediated Sequence-Specific Recognition by Hin Recombinase 著者: Chiu, T.K. #2:  ジャーナル: Science / 年: 1994 ジャーナル: Science / 年: 1994タイトル: Hin Recombinase Bound to DNA: The Origin of Specificity in Major and Minor Groove Interactions 著者: Feng, J.A. / Johnson, R.C. / Dickerson, R.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1jkp.cif.gz 1jkp.cif.gz | 38.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1jkp.ent.gz pdb1jkp.ent.gz | 24.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1jkp.json.gz 1jkp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1jkp_validation.pdf.gz 1jkp_validation.pdf.gz | 440.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1jkp_full_validation.pdf.gz 1jkp_full_validation.pdf.gz | 443.6 KB | 表示 | |
| XML形式データ |  1jkp_validation.xml.gz 1jkp_validation.xml.gz | 5.2 KB | 表示 | |
| CIF形式データ |  1jkp_validation.cif.gz 1jkp_validation.cif.gz | 6.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jk/1jkp https://data.pdbj.org/pub/pdb/validation_reports/jk/1jkp ftp://data.pdbj.org/pub/pdb/validation_reports/jk/1jkp ftp://data.pdbj.org/pub/pdb/validation_reports/jk/1jkp | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 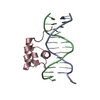
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 4349.850 Da / 分子数: 1 / 由来タイプ: 合成 |
|---|---|
| #2: DNA鎖 | 分子量: 4207.781 Da / 分子数: 1 / 由来タイプ: 合成 |
| #3: タンパク質 | 分子量: 6047.051 Da / 分子数: 1 / Fragment: RESIDUES 139 TO 190 / 由来タイプ: 合成 / 詳細: SYNTHETIC PEPTIDE / 参照: UniProt: P03013 |
| #4: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.72 Å3/Da / 溶媒含有率: 53.11 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 詳細: HANGING DROP VAPOR DIFFUSION AT 4C, WITH INITIAL CONCENTRATION IN DROP OF 0.10 MM DNA, 0.06 MM HIN, 10 MM TRIS (PH 8.5), 5 MM CACL2, 23 MM NACL, 2.5% V/V PEG400, AND 1.56 MM NA CACODYLATE. ...詳細: HANGING DROP VAPOR DIFFUSION AT 4C, WITH INITIAL CONCENTRATION IN DROP OF 0.10 MM DNA, 0.06 MM HIN, 10 MM TRIS (PH 8.5), 5 MM CACL2, 23 MM NACL, 2.5% V/V PEG400, AND 1.56 MM NA CACODYLATE. RESERVOIR SOLUTION CONTAINS 100 MM TRIS (PH 8.5), 50 MM CACL2, 100 MM NACL, AND 25% PEG400. CONCENTRATION OF PEG400 IN RESERVOIR SOLUTION WAS INCREASED IN 5% INCREMENTS TO 35%., pH 8.50, VAPOR DIFFUSION, HANGING DROP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / pH: 8.5 / 手法: 蒸気拡散法, シッティングドロップ法 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS NSLS  / ビームライン: X25 / 波長: 1.1 / ビームライン: X25 / 波長: 1.1 |
| 検出器 | タイプ: MAR scanner 300 mm plate / 検出器: IMAGE PLATE / 日付: 1997年9月1日 |
| 放射 | プロトコル: MOLECULAR REPLACEMENT WITH 1IJW HAVING THE PROPER DNA SUBSTITUTIONS AS THE STARTING MODEL. 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.1 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→42.7 Å / Num. obs: 3774 / % possible obs: 91.23 % / Observed criterion σ(I): 0 / 冗長度: 13 % / Biso Wilson estimate: 43 Å2 / Rsym value: 0.077 / Net I/σ(I): 16.9 |
| 反射 シェル | 解像度: 2.8→2.93 Å / 冗長度: 3 % / Mean I/σ(I) obs: 4.06 / Rsym value: 0.218 / % possible all: 76.37 |
| 反射 | *PLUS 最高解像度: 2.83 Å / % possible obs: 91.2 % / 冗長度: 13 % / Rmerge(I) obs: 0.077 |
| 反射 シェル | *PLUS % possible obs: 76.4 % / Rmerge(I) obs: 0.218 / Mean I/σ(I) obs: 4.1 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1IJW 解像度: 2.8→42.7 Å / Isotropic thermal model: ANISOTROPIC_FIXED_ISOTROPIC / σ(F): 0 / 立体化学のターゲット値: MLF
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | Bsol: 50 Å2 / ksol: 0.2 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 54.1 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→42.7 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.93 Å / Total num. of bins used: 8
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS % reflection Rfree: 10 % / 最高解像度: 2.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor obs: 0.416 |
 ムービー
ムービー コントローラー
コントローラー











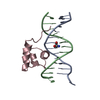
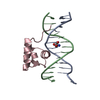




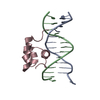


 PDBj
PDBj







































