+ データを開く
データを開く
- 基本情報
基本情報
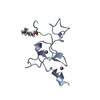
| 登録情報 | データベース: PDB / ID: 1irs | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | IRS-1 PTB DOMAIN COMPLEXED WITH A IL-4 RECEPTOR PHOSPHOPEPTIDE, NMR, MINIMIZED AVERAGE STRUCTURE | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | COMPLEX (SIGNAL TRANSDUCTION/PEPTIDE) / PHOSPHOTYROSINE BINDING DOMAIN (PTB) / COMPLEX / SIGNAL TRANSDUCTION / COMPLEX (SIGNAL TRANSDUCTION-PEPTIDE) / COMPLEX (SIGNAL TRANSDUCTION-PEPTIDE) complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報interleukin-4 receptor activity / negative regulation of T-helper 1 cell differentiation / T-helper 1 cell differentiation / production of molecular mediator involved in inflammatory response / positive regulation of T-helper 2 cell differentiation / IRS-related events triggered by IGF1R / positive regulation of glucose metabolic process / positive regulation of fatty acid beta-oxidation / interleukin-4-mediated signaling pathway / positive regulation of mast cell degranulation ...interleukin-4 receptor activity / negative regulation of T-helper 1 cell differentiation / T-helper 1 cell differentiation / production of molecular mediator involved in inflammatory response / positive regulation of T-helper 2 cell differentiation / IRS-related events triggered by IGF1R / positive regulation of glucose metabolic process / positive regulation of fatty acid beta-oxidation / interleukin-4-mediated signaling pathway / positive regulation of mast cell degranulation / IRS-mediated signalling / insulin receptor complex / Activated NTRK3 signals through PI3K / transmembrane receptor protein tyrosine kinase adaptor activity / positive regulation of macrophage activation / Signaling by Leptin / cellular response to fatty acid / cytokine receptor activity / Signaling by LTK / PI3K/AKT activation / T-helper 2 cell differentiation / positive regulation of immunoglobulin production / positive regulation of myoblast fusion / Signaling by ALK / IRS activation / defense response to protozoan / immunoglobulin mediated immune response / PI3K Cascade / SOS-mediated signalling / positive regulation of insulin receptor signaling pathway / positive regulation of glycogen biosynthetic process / Signal attenuation / Growth hormone receptor signaling / phosphatidylinositol 3-kinase binding / positive regulation of chemokine production / insulin-like growth factor receptor binding / signaling adaptor activity / phosphotyrosine residue binding / Interleukin-7 signaling / SH2 domain binding / negative regulation of insulin receptor signaling pathway / insulin-like growth factor receptor signaling pathway / protein kinase C binding / positive regulation of D-glucose import / insulin receptor binding / phosphatidylinositol 3-kinase/protein kinase B signal transduction / negative regulation of insulin secretion / response to insulin / cytokine-mediated signaling pathway / caveola / centriolar satellite / cellular response to insulin stimulus / Constitutive Signaling by Aberrant PI3K in Cancer / Signaling by ALK fusions and activated point mutants / insulin receptor signaling pathway / glucose homeostasis / PIP3 activates AKT signaling / signaling receptor complex adaptor activity / positive regulation of cold-induced thermogenesis / RAF/MAP kinase cascade / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / Interleukin-4 and Interleukin-13 signaling / receptor complex / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / immune response / external side of plasma membrane / intracellular membrane-bounded organelle / positive regulation of cell population proliferation / signal transduction / extracellular region / nucleoplasm / nucleus / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR | ||||||
 データ登録者 データ登録者 | Zhou, M.-M. / Huang, B. / Olejniczak, E.T. / Meadows, R.P. / Shuker, S.B. / Miyazaki, M. / Trub, T. / Shoelson, S.E. / Feisk, S.W. | ||||||
 引用 引用 |  ジャーナル: Nat.Struct.Biol. / 年: 1996 ジャーナル: Nat.Struct.Biol. / 年: 1996タイトル: Structural basis for IL-4 receptor phosphopeptide recognition by the IRS-1 PTB domain. 著者: Zhou, M.M. / Huang, B. / Olejniczak, E.T. / Meadows, R.P. / Shuker, S.B. / Miyazaki, M. / Trub, T. / Shoelson, S.E. / Fesik, S.W. #1:  ジャーナル: J.Biol.Chem. / 年: 1995 ジャーナル: J.Biol.Chem. / 年: 1995タイトル: Ptb Domains of Irs-1 and Shc Have Distinct But Overlapping Binding Specificities 著者: Wolf, G. / Trub, T. / Ottinger, E. / Groninga, L. / Lynch, A. / White, M.F. / Miyazaki, M. / Lee, J. / Shoelson, S.E. #2:  ジャーナル: Nature / 年: 1995 ジャーナル: Nature / 年: 1995タイトル: Structure and Ligand Recognition of the Phosphotyrosine Binding Domain of Shc 著者: Zhou, M.M. / Ravichandran, K.S. / Olejniczak, E.F. / Petros, A.M. / Meadows, R.P. / Sattler, M. / Harlan, J.E. / Wade, W.S. / Burakoff, S.J. / Fesik, S.W. #3:  ジャーナル: Mol.Cell.Biol. / 年: 1994 ジャーナル: Mol.Cell.Biol. / 年: 1994タイトル: Characterization of an Interaction between Insulin Receptor Substrate 1 and the Insulin Receptor by Using the Two-Hybrid System 著者: O'Neill, T.J. / Craparo, A. / Gustafson, T.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1irs.cif.gz 1irs.cif.gz | 56.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1irs.ent.gz pdb1irs.ent.gz | 39.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1irs.json.gz 1irs.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1irs_validation.pdf.gz 1irs_validation.pdf.gz | 258.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1irs_full_validation.pdf.gz 1irs_full_validation.pdf.gz | 258.4 KB | 表示 | |
| XML形式データ |  1irs_validation.xml.gz 1irs_validation.xml.gz | 4.5 KB | 表示 | |
| CIF形式データ |  1irs_validation.cif.gz 1irs_validation.cif.gz | 5.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ir/1irs https://data.pdbj.org/pub/pdb/validation_reports/ir/1irs ftp://data.pdbj.org/pub/pdb/validation_reports/ir/1irs ftp://data.pdbj.org/pub/pdb/validation_reports/ir/1irs | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 12648.737 Da / 分子数: 1 / 断片: PTB DOMAIN / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 株: BL21 (DE2) PLYSS / 細胞株: BL21 / 遺伝子: PTB DOMAIN OF IRS-1 / 器官: SKELETAL / プラスミド: PET30B / 遺伝子 (発現宿主): PTB DOMAIN OF IRS-1 / 発現宿主: Homo sapiens (ヒト) / 株: BL21 (DE2) PLYSS / 細胞株: BL21 / 遺伝子: PTB DOMAIN OF IRS-1 / 器官: SKELETAL / プラスミド: PET30B / 遺伝子 (発現宿主): PTB DOMAIN OF IRS-1 / 発現宿主:  |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 1241.311 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:  |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|
- 試料調製
試料調製
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
|---|
- 解析
解析
| ソフトウェア |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| NMR software | 名称:  X-PLOR / 開発者: BRUNGER / 分類: 精密化 X-PLOR / 開発者: BRUNGER / 分類: 精密化 | ||||||||
| 精密化 | ソフトェア番号: 1 詳細: SET OF IDEAL BOND LENGTHS AND ANGLES USED DURING REFINEMENT: PARALLHDG.PRO IN X-PLOR. | ||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj












