+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1i8g | ||||||
|---|---|---|---|---|---|---|---|
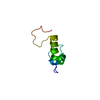
| タイトル | SOLUTION STRUCTURE OF PIN1 WW DOMAIN COMPLEXED WITH CDC25 PHOSPHOTHREONINE PEPTIDE | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE/ISOMERASE / CELL DIVISION / NUCLEAR PROTEIN / HYDROLASE-ISOMERASE COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of G2/MI transition of meiotic cell cycle / cis-trans isomerase activity / phosphothreonine residue binding / negative regulation of cell motility / ubiquitin ligase activator activity / regulation of protein localization to nucleus / GTPase activating protein binding / mitogen-activated protein kinase kinase binding / protein targeting to mitochondrion / protein peptidyl-prolyl isomerization ...positive regulation of G2/MI transition of meiotic cell cycle / cis-trans isomerase activity / phosphothreonine residue binding / negative regulation of cell motility / ubiquitin ligase activator activity / regulation of protein localization to nucleus / GTPase activating protein binding / mitogen-activated protein kinase kinase binding / protein targeting to mitochondrion / protein peptidyl-prolyl isomerization / regulation of mitotic nuclear division / negative regulation of SMAD protein signal transduction / PI5P Regulates TP53 Acetylation / negative regulation of amyloid-beta formation / cytoskeletal motor activity / RHO GTPases Activate NADPH Oxidases / phosphoserine residue binding / positive regulation of G2/M transition of mitotic cell cycle / postsynaptic cytosol / negative regulation of protein binding / Rho protein signal transduction / protein-tyrosine-phosphatase / protein tyrosine phosphatase activity / Negative regulators of DDX58/IFIH1 signaling / regulation of cytokinesis / peptidylprolyl isomerase / phosphoprotein binding / peptidyl-prolyl cis-trans isomerase activity / negative regulation of transforming growth factor beta receptor signaling pathway / ISG15 antiviral mechanism / synapse organization / negative regulation of protein catabolic process / beta-catenin binding / regulation of protein stability / negative regulation of ERK1 and ERK2 cascade / tau protein binding / G2/M transition of mitotic cell cycle / positive regulation of protein phosphorylation / neuron differentiation / positive regulation of canonical Wnt signaling pathway / regulation of gene expression / midbody / Regulation of TP53 Activity through Phosphorylation / cellular response to hypoxia / response to hypoxia / protein stabilization / nuclear speck / ciliary basal body / cell division / glutamatergic synapse / positive regulation of transcription by RNA polymerase II / nucleoplasm / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 手法 | 溶液NMR / distance geometry simulated annealing | ||||||
 データ登録者 データ登録者 | Wintjens, R. / Wieruszeski, J.-M. / Drobecq, H. / Lippens, G. / Landrieu, I. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2001 ジャーナル: J.Biol.Chem. / 年: 2001タイトル: 1H NMR study on the binding of Pin1 Trp-Trp domain with phosphothreonine peptides. 著者: Wintjens, R. / Wieruszeski, J.M. / Drobecq, H. / Rousselot-Pailley, P. / Buee, L. / Lippens, G. / Landrieu, I. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1i8g.cif.gz 1i8g.cif.gz | 162.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1i8g.ent.gz pdb1i8g.ent.gz | 133.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1i8g.json.gz 1i8g.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1i8g_validation.pdf.gz 1i8g_validation.pdf.gz | 366.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1i8g_full_validation.pdf.gz 1i8g_full_validation.pdf.gz | 460.2 KB | 表示 | |
| XML形式データ |  1i8g_validation.xml.gz 1i8g_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  1i8g_validation.cif.gz 1i8g_validation.cif.gz | 18.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i8/1i8g https://data.pdbj.org/pub/pdb/validation_reports/i8/1i8g ftp://data.pdbj.org/pub/pdb/validation_reports/i8/1i8g ftp://data.pdbj.org/pub/pdb/validation_reports/i8/1i8g | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 1192.209 Da / 分子数: 1 / 断片: RESIDUES 63-72 / 由来タイプ: 合成 詳細: The ligand phosphopeptide was synthesized from Rink amide resin using the Fmoc strategy and activation by HBTU and HOBT in a 431A peptide synthesizer. The sequence of the peptide is naturally ...詳細: The ligand phosphopeptide was synthesized from Rink amide resin using the Fmoc strategy and activation by HBTU and HOBT in a 431A peptide synthesizer. The sequence of the peptide is naturally found in Xenopus laevis (African clawed frog). 参照: UniProt: P30311, protein-tyrosine-phosphatase |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 4462.899 Da / 分子数: 1 / 断片: WW DOMAIN (RESIDUES 6-44) / 由来タイプ: 合成 詳細: The Pin1 WW domain was obtained by peptide synthesis using the BOC-benzyl strategy and the HBTU in situ activation protocol on an Applied 430A peptide synthesizer. The sequence of the peptide ...詳細: The Pin1 WW domain was obtained by peptide synthesis using the BOC-benzyl strategy and the HBTU in situ activation protocol on an Applied 430A peptide synthesizer. The sequence of the peptide is naturally found in Homo sapiens (Human). 参照: UniProt: Q13526, peptidylprolyl isomerase |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験 | タイプ: 2D NOESY |
- 試料調製
試料調製
| 詳細 | 内容: sample of 1mM WW domain / 4.5 mM Cdc25 ligand buffer of 50 mM deutered Tris-D2O, pH 6.4, 100 mM NaCl 溶媒系: 90% H2O/10% D2O |
|---|---|
| 試料状態 | イオン強度: 100 mM NaCl / pH: 6.4 / 圧: ambient / 温度: 285 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker DMX / 製造業者: Bruker / モデル: DMX / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: distance geometry simulated annealing / ソフトェア番号: 1 詳細: hybrid of distance geometry / simulated annealing protocol Minimization procedure using CVFF as force field | ||||||||||||
| 代表構造 | 選択基準: closest to the average | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj







 X-PLOR
X-PLOR