+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1i31 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | MU2 ADAPTIN SUBUNIT (AP50) OF AP2 CLATHRIN ADAPTOR, COMPLEXED WITH EGFR INTERNALIZATION PEPTIDE FYRALM AT 2.5 A RESOLUTION | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | ENDOCYTOSIS/EXOCYTOSIS / beta-sandwich / peptide-binding site / protein-peptide complex / clathrin adaptor / ENDOCYTOSIS-EXOCYTOSIS COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報diterpenoid metabolic process / positive regulation of prolactin secretion / response to hydroxyisoflavone / ovulation cycle / positive regulation of mucus secretion / Gap junction degradation / Formation of annular gap junctions / WNT5A-dependent internalization of FZD2, FZD5 and ROR2 / LDL clearance / tongue development ...diterpenoid metabolic process / positive regulation of prolactin secretion / response to hydroxyisoflavone / ovulation cycle / positive regulation of mucus secretion / Gap junction degradation / Formation of annular gap junctions / WNT5A-dependent internalization of FZD2, FZD5 and ROR2 / LDL clearance / tongue development / Retrograde neurotrophin signalling / VLDLR internalisation and degradation / WNT5A-dependent internalization of FZD4 / midgut development / extrinsic component of presynaptic endocytic zone membrane / MHC class II antigen presentation / hydrogen peroxide metabolic process / regulation of vesicle size / AP-2 adaptor complex / postsynaptic neurotransmitter receptor internalization / Recycling pathway of L1 / response to cobalamin / positive regulation of synaptic vesicle endocytosis / Cargo recognition for clathrin-mediated endocytosis / clathrin adaptor activity / Clathrin-mediated endocytosis / vesicle budding from membrane / clathrin-dependent endocytosis / signal sequence binding / positive regulation of bone resorption / negative regulation of mitotic cell cycle / multivesicular body, internal vesicle lumen / negative regulation of cardiocyte differentiation / epidermal growth factor receptor activity / epidermal growth factor binding / response to UV-A / low-density lipoprotein particle receptor binding / ERBB2-EGFR signaling pathway / morphogenesis of an epithelial fold / response to lipid / digestive tract morphogenesis / positive regulation of vasoconstriction / intracellular vesicle / negative regulation of epidermal growth factor receptor signaling pathway / eyelid development in camera-type eye / cerebral cortex cell migration / Trafficking of GluR2-containing AMPA receptors / protein tyrosine kinase activator activity / positive regulation of receptor internalization / positive regulation of glial cell proliferation / positive regulation of phosphorylation / endocytic vesicle / synaptic vesicle endocytosis / epidermis development / hair follicle development / cellular response to dexamethasone stimulus / positive regulation of G1/S transition of mitotic cell cycle / embryonic placenta development / negative regulation of protein localization to plasma membrane / positive regulation of synaptic transmission, glutamatergic / salivary gland morphogenesis / positive regulation of superoxide anion generation / neuron projection morphogenesis / clathrin-coated pit / transmembrane receptor protein tyrosine kinase activity / liver regeneration / astrocyte activation / positive regulation of smooth muscle cell proliferation / receptor-mediated endocytosis / lung development / positive regulation of DNA repair / basal plasma membrane / synaptic membrane / positive regulation of DNA replication / epithelial cell proliferation / positive regulation of epithelial cell proliferation / positive regulation of protein localization to plasma membrane / cellular response to amino acid stimulus / intracellular protein transport / phosphatidylinositol 3-kinase/protein kinase B signal transduction / cellular response to estradiol stimulus / liver development / cellular response to mechanical stimulus / cell-cell adhesion / circadian rhythm / receptor protein-tyrosine kinase / response to calcium ion / negative regulation of protein catabolic process / cellular response to growth factor stimulus / receptor internalization / positive regulation of miRNA transcription / kinase binding / vasodilation / ruffle membrane / integrin binding / cellular response to xenobiotic stimulus / epidermal growth factor receptor signaling pathway / cell morphogenesis / terminal bouton / neuron differentiation 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å | ||||||
 データ登録者 データ登録者 | Modis, Y. / Boll, W. / Rapoport, I. / Kirchhausen, T. | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: MU2 ADAPTIN SUBUNIT (AP50) OF AP2 CLATHRIN ADAPTOR, COMPLEXED WITH EGFR INTERNALIZATION PEPTIDE FYRALM AT 2.5 A RESOLUTION 著者: Modis, Y. / Boll, W. / Rapoport, I. / Kirchhausen, T. #1:  ジャーナル: Science / 年: 1998 ジャーナル: Science / 年: 1998タイトル: A Structural Explanation for the Recognition of Tyrosine-Based Endocytic Signals 著者: Owen, D.J. / Evans, P.R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1i31.cif.gz 1i31.cif.gz | 70.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1i31.ent.gz pdb1i31.ent.gz | 50.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1i31.json.gz 1i31.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1i31_validation.pdf.gz 1i31_validation.pdf.gz | 438.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1i31_full_validation.pdf.gz 1i31_full_validation.pdf.gz | 459.7 KB | 表示 | |
| XML形式データ |  1i31_validation.xml.gz 1i31_validation.xml.gz | 15.1 KB | 表示 | |
| CIF形式データ |  1i31_validation.cif.gz 1i31_validation.cif.gz | 19.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i3/1i31 https://data.pdbj.org/pub/pdb/validation_reports/i3/1i31 ftp://data.pdbj.org/pub/pdb/validation_reports/i3/1i31 ftp://data.pdbj.org/pub/pdb/validation_reports/i3/1i31 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1bw8S S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 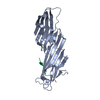
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 35746.543 Da / 分子数: 1 / 断片: RESIDUES 122-435 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 800.988 Da / 分子数: 1 / 断片: RESIDUES 998-1003 / 由来タイプ: 合成 詳細: The hexapeptide of epidermal growth factor receptor was chemically synthesized 参照: GenBank: 6478868, UniProt: A8IP97*PLUS |
| #3: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.66 Å3/Da / 溶媒含有率: 73.59 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.5 詳細: SODIUM FORMATE, SODIUM ACETATE, pH 4.5, VAPOR DIFFUSION, HANGING DROP, temperature 291K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 14-BM-D / 波長: 1.006 Å / ビームライン: 14-BM-D / 波長: 1.006 Å |
| 検出器 | タイプ: ADSC QUANTUM 4 / 検出器: CCD / 日付: 2000年6月14日 / 詳細: BENT CYLINDRICAL SI-MIRROR (RH COATING) |
| 放射 | モノクロメーター: SI(111) DOUBLE CRYSTAL MONOCHROMATOR プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.006 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→20 Å / Num. all: 21919 / Num. obs: 21919 / % possible obs: 96.4 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 50.8 % / Biso Wilson estimate: 74.6 Å2 / Rmerge(I) obs: 0.077 / Net I/σ(I): 25.8 |
| 反射 シェル | 解像度: 2.52→2.56 Å / 冗長度: 6 % / Rmerge(I) obs: 0.599 / Mean I/σ(I) obs: 1.6 / % possible all: 76.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1BW8 解像度: 2.5→20 Å Isotropic thermal model: ISOTROPIC ATOMIC TEMPERATURE FACTORS 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: ENGH & HUBER 詳細: MINIMIZATION OF MAXIMUM LIKELIHOOD RESIDUAL BY CONJUGATE DIRECTION METHOD
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: -0.01087 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj





