+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1gpx | ||||||
|---|---|---|---|---|---|---|---|
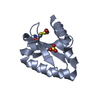
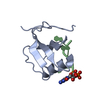
| タイトル | C85S GAPDX, NMR, 20 STRUCTURES | ||||||
 要素 要素 | PUTIDAREDOXIN | ||||||
 キーワード キーワード | ELECTRON TRANSPORT / GAPDX C85S / 20 STRUCTURES ALIGNED AND SA | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報P450-containing electron transport chain / 2 iron, 2 sulfur cluster binding / electron transfer activity / metal ion binding / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Pseudomonas putida (バクテリア) Pseudomonas putida (バクテリア) | ||||||
| 手法 | 溶液NMR / DG, SIMULATED ANNEALING | ||||||
 データ登録者 データ登録者 | Pochapsky, T.C. / Kuti, M. / Kazanis, S. | ||||||
 引用 引用 |  ジャーナル: J.Biomol.NMR / 年: 1998 ジャーナル: J.Biomol.NMR / 年: 1998タイトル: The solution structure of a gallium-substituted putidaredoxin mutant: GaPdx C85S. 著者: Pochapsky, T.C. / Kuti, M. / Kazanis, S. #1:  ジャーナル: J.Biomol.NMR / 年: 1997 ジャーナル: J.Biomol.NMR / 年: 1997タイトル: Structural Features of the Metal Binding Site and Dynamics of Gallium Putidaredoxin, a Diamagnetic Derivative of a Cys4Fe2S2 Ferredoxin 著者: Kazanis, S. / Pochapsky, T.C. #2:  ジャーナル: J.Am.Chem.Soc. / 年: 1995 ジャーナル: J.Am.Chem.Soc. / 年: 1995タイトル: Conversion of a Fe2S2 Ferredoxin Into a Ga+3 Rubredoxin 著者: Kazanis, S. / Pochapsky, T.C. / Barnhart, T.M. / Penner-Hahn, J.E. / Mirza, U.A. / Chait, B.T. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1gpx.cif.gz 1gpx.cif.gz | 615.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1gpx.ent.gz pdb1gpx.ent.gz | 509.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1gpx.json.gz 1gpx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1gpx_validation.pdf.gz 1gpx_validation.pdf.gz | 357.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1gpx_full_validation.pdf.gz 1gpx_full_validation.pdf.gz | 628.7 KB | 表示 | |
| XML形式データ |  1gpx_validation.xml.gz 1gpx_validation.xml.gz | 110.6 KB | 表示 | |
| CIF形式データ |  1gpx_validation.cif.gz 1gpx_validation.cif.gz | 149.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gp/1gpx https://data.pdbj.org/pub/pdb/validation_reports/gp/1gpx ftp://data.pdbj.org/pub/pdb/validation_reports/gp/1gpx ftp://data.pdbj.org/pub/pdb/validation_reports/gp/1gpx | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 11412.846 Da / 分子数: 1 / 変異: C85S / 由来タイプ: 組換発現 詳細: COORDINATION OF GA+3 BY SG OF CYS 39, CYS 45, CYS 48 AND CYS 86 由来: (組換発現)  Pseudomonas putida (バクテリア) / 解説: FROM P. PUTIDA CAMPHOR HYDROXYLASE SYSTEM / 遺伝子: CAMB / プラスミド: PKM536 / 発現宿主: Pseudomonas putida (バクテリア) / 解説: FROM P. PUTIDA CAMPHOR HYDROXYLASE SYSTEM / 遺伝子: CAMB / プラスミド: PKM536 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-GA / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||
| NMR実験の詳細 | Text: TRIPLE-RESONANCE 13C,15N NMR SPECTROSCOPY |
- 試料調製
試料調製
| 詳細 | 内容: 90/10 H2O/D2O |
|---|---|
| 試料状態 | イオン強度: 0.01M / pH: 7.4 / 圧: 1 ATMOSPHERE / 温度: 298 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR software |
| ||||||||||||
| 精密化 | 手法: DG, SIMULATED ANNEALING / ソフトェア番号: 1 / 詳細: REFINEMENT DETAILS IN PRIMARY REFERENCE | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj








 X-PLOR
X-PLOR