| 登録情報 | データベース: PDB / ID: 1fsb
|
|---|
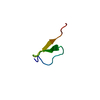
| タイトル | STRUCTURE OF THE EGF DOMAIN OF P-SELECTIN, NMR, 19 STRUCTURES |
|---|
 要素 要素 | P-SELECTIN |
|---|
 キーワード キーワード | CELL ADHESION PROTEIN / EGF-LIKE DOMAIN / TRANSMEMBRANE / GLYCOPROTEIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of integrin activation / fucose binding / glycosphingolipid binding / platelet dense granule membrane / positive regulation of leukocyte tethering or rolling / sialic acid binding / oligosaccharide binding / calcium-dependent cell-cell adhesion / platelet alpha granule membrane / leukocyte tethering or rolling ...regulation of integrin activation / fucose binding / glycosphingolipid binding / platelet dense granule membrane / positive regulation of leukocyte tethering or rolling / sialic acid binding / oligosaccharide binding / calcium-dependent cell-cell adhesion / platelet alpha granule membrane / leukocyte tethering or rolling / positive regulation of platelet activation / positive regulation of leukocyte migration / heterophilic cell-cell adhesion / leukocyte cell-cell adhesion / response to cytokine / Cell surface interactions at the vascular wall / lipopolysaccharide binding / cell-cell adhesion / integrin binding / calcium-dependent protein binding / Platelet degranulation / heparin binding / response to lipopolysaccharide / defense response to Gram-negative bacterium / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / cell adhesion / inflammatory response / external side of plasma membrane / calcium ion binding / extracellular space / plasma membrane類似検索 - 分子機能 Selectin superfamily / Selectin, C-type lectin-like domain / : / C-type lectin, conserved site / C-type lectin domain signature. / Sushi repeat (SCR repeat) / Domain abundant in complement control proteins; SUSHI repeat; short complement-like repeat (SCR) / Sushi/SCR/CCP domain / Sushi/CCP/SCR domain profile. / Sushi/SCR/CCP superfamily ...Selectin superfamily / Selectin, C-type lectin-like domain / : / C-type lectin, conserved site / C-type lectin domain signature. / Sushi repeat (SCR repeat) / Domain abundant in complement control proteins; SUSHI repeat; short complement-like repeat (SCR) / Sushi/SCR/CCP domain / Sushi/CCP/SCR domain profile. / Sushi/SCR/CCP superfamily / Lectin C-type domain / C-type lectin domain profile. / C-type lectin-like / C-type lectin (CTL) or carbohydrate-recognition domain (CRD) / Laminin / Laminin / C-type lectin-like/link domain superfamily / C-type lectin fold / EGF-like domain profile. / EGF-like domain signature 1. / EGF-like domain signature 2. / EGF-like domain / Ribbon / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR |
|---|
 データ登録者 データ登録者 | Freedman, S.J. / Sanford, D.G. / Bachovchin, W.W. / Furie, B.C. / Baleja, J.D. / Furie, B. |
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 1996 ジャーナル: Biochemistry / 年: 1996
タイトル: Structure and function of the epidermal growth factor domain of P-selectin.
著者: Freedman, S.J. / Sanford, D.G. / Bachovchin, W.W. / Furie, B.C. / Baleja, J.D. / Furie, B. |
|---|
| 履歴 | | 登録 | 1996年3月25日 | 処理サイト: BNL |
|---|
| 改定 1.0 | 1997年4月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年3月24日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2017年11月29日 | Group: Derived calculations / Other
カテゴリ: pdbx_database_status / pdbx_struct_assembly ...pdbx_database_status / pdbx_struct_assembly / pdbx_struct_oper_list / struct_conf
Item: _pdbx_database_status.process_site |
|---|
| 改定 1.4 | 2024年11月6日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 1996
ジャーナル: Biochemistry / 年: 1996 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1fsb.cif.gz
1fsb.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1fsb.ent.gz
pdb1fsb.ent.gz PDB形式
PDB形式 1fsb.json.gz
1fsb.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1fsb_validation.pdf.gz
1fsb_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1fsb_full_validation.pdf.gz
1fsb_full_validation.pdf.gz 1fsb_validation.xml.gz
1fsb_validation.xml.gz 1fsb_validation.cif.gz
1fsb_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/fs/1fsb
https://data.pdbj.org/pub/pdb/validation_reports/fs/1fsb ftp://data.pdbj.org/pub/pdb/validation_reports/fs/1fsb
ftp://data.pdbj.org/pub/pdb/validation_reports/fs/1fsb リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 参照: UniProt: P16109
Homo sapiens (ヒト) / 参照: UniProt: P16109 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj






