| 登録情報 | データベース: PDB / ID: 1f3v
|
|---|
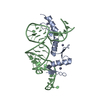
| タイトル | Crystal structure of the complex between the N-terminal domain of TRADD and the TRAF domain of TRAF2 |
|---|
 要素 要素 | - TUMOR NECROSIS FACTOR RECEPTOR TYPE 1 ASSOCIATED DEATH DOMAIN PROTEIN
- TUMOR NECROSIS FACTOR RECEPTOR-ASSOCIATED PROTEIN
|
|---|
 キーワード キーワード | APOPTOSIS / a-b sandwich |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
TORC2 complex disassembly / CD40 receptor binding / TORC1 complex assembly / tumor necrosis factor receptor superfamily complex / death domain binding / sphingolipid binding / TRAF2-GSTP1 complex / IRE1-TRAF2-ASK1 complex / CD27 signaling pathway / positive regulation of hair follicle development ...TORC2 complex disassembly / CD40 receptor binding / TORC1 complex assembly / tumor necrosis factor receptor superfamily complex / death domain binding / sphingolipid binding / TRAF2-GSTP1 complex / IRE1-TRAF2-ASK1 complex / CD27 signaling pathway / positive regulation of hair follicle development / Defective RIPK1-mediated regulated necrosis / TNF receptor superfamily (TNFSF) members mediating non-canonical NF-kB pathway / CD40 signaling pathway / tumor necrosis factor binding / TRAIL-activated apoptotic signaling pathway / Regulation by c-FLIP / CASP8 activity is inhibited / Dimerization of procaspase-8 / interleukin-17-mediated signaling pathway / negative regulation of glial cell apoptotic process / TNF signaling / CD40 receptor complex / programmed necrotic cell death / Caspase activation via Death Receptors in the presence of ligand / death-inducing signaling complex / transmembrane receptor protein tyrosine kinase adaptor activity / thioesterase binding / positive regulation of tumor necrosis factor-mediated signaling pathway / tumor necrosis factor receptor binding / mRNA stabilization / regulation of immunoglobulin production / non-canonical NF-kappaB signal transduction / vesicle membrane / regulation of JNK cascade / positive regulation of extrinsic apoptotic signaling pathway / mitogen-activated protein kinase kinase kinase binding / signal transduction involved in regulation of gene expression / TNFR1-induced proapoptotic signaling / TRAF6 mediated IRF7 activation / RIPK1-mediated regulated necrosis / positive regulation of JUN kinase activity / TRAF6 mediated NF-kB activation / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / extrinsic apoptotic signaling pathway via death domain receptors / protein K63-linked ubiquitination / canonical NF-kappaB signal transduction / ubiquitin ligase complex / regulation of protein-containing complex assembly / protein autoubiquitination / extrinsic apoptotic signaling pathway / signaling adaptor activity / positive regulation of interleukin-2 production / cellular response to nitric oxide / response to endoplasmic reticulum stress / T cell activation / tumor necrosis factor-mediated signaling pathway / TNFR1-induced NF-kappa-B signaling pathway / Regulation of NF-kappa B signaling / TNFR2 non-canonical NF-kB pathway / Regulation of TNFR1 signaling / protein catabolic process / positive regulation of NF-kappaB transcription factor activity / positive regulation of T cell cytokine production / RING-type E3 ubiquitin transferase / Regulation of necroptotic cell death / kinase binding / cytoplasmic side of plasma membrane / positive regulation of inflammatory response / ubiquitin-protein transferase activity / cellular response to tumor necrosis factor / ubiquitin protein ligase activity / protein-containing complex assembly / cell cortex / protein phosphatase binding / regulation of apoptotic process / protein-macromolecule adaptor activity / cytoskeleton / positive regulation of canonical NF-kappaB signal transduction / receptor complex / Ub-specific processing proteases / positive regulation of cell migration / positive regulation of apoptotic process / membrane raft / innate immune response / apoptotic process / ubiquitin protein ligase binding / protein kinase binding / protein-containing complex binding / enzyme binding / signal transduction / zinc ion binding / nucleoplasm / identical protein binding / nucleus / plasma membrane / cytoplasm / cytosol類似検索 - 分子機能 TRADD, N-terminal domain / TRADD, N-terminal / TRADD / TRADD, N-terminal domain superfamily / TRADD, N-terminal domain / TNF receptor-associated factor 2 / TNF receptor-associated factor 2, MATH domain / : / TRAF2, second zinc finger / TNF receptor-associated factor, BIRC3 binding domain ...TRADD, N-terminal domain / TRADD, N-terminal / TRADD / TRADD, N-terminal domain superfamily / TRADD, N-terminal domain / TNF receptor-associated factor 2 / TNF receptor-associated factor 2, MATH domain / : / TRAF2, second zinc finger / TNF receptor-associated factor, BIRC3 binding domain / TNF receptor-associated factor BIRC3 binding domain / TRAF-type zinc finger / TNF receptor-associated factor TRAF, metazoa / : / TRAF/meprin, MATH domain / Zinc finger, TRAF-type / Zinc finger TRAF-type profile. / Apoptosis, Tumor Necrosis Factor Receptor Associated Protein 2; Chain A / Apoptosis, Tumor Necrosis Factor Receptor Associated Protein 2; Chain A / MATH/TRAF domain / MATH/TRAF domain profile. / meprin and TRAF homology / TRAF-like / Death domain profile. / DEATH domain, found in proteins involved in cell death (apoptosis). / Death domain / Death domain / Zinc finger, C3HC4 RING-type / Zinc finger, C3HC4 type (RING finger) / Zinc finger, RING-type, conserved site / Zinc finger RING-type signature. / Death-like domain superfamily / Ring finger / Zinc finger RING-type profile. / Zinc finger, RING-type / Zinc finger, RING/FYVE/PHD-type / Alpha-Beta Plaits / Sandwich / 2-Layer Sandwich / Mainly Beta / Alpha Beta類似検索 - ドメイン・相同性 TNF receptor-associated factor 2 / Tumor necrosis factor receptor type 1-associated DEATH domain protein類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2 Å シンクロトロン / 解像度: 2 Å |
|---|
 データ登録者 データ登録者 | Park, Y.C. / Ye, H. / Hsia, C. / Segal, D. / Rich, R. / Liou, H.-C. / Myszka, D. / Wu, H. |
|---|
 引用 引用 |  ジャーナル: Cell(Cambridge,Mass.) / 年: 2000 ジャーナル: Cell(Cambridge,Mass.) / 年: 2000
タイトル: A novel mechanism of TRAF signaling revealed by structural and functional analyses of the TRADD-TRAF2 interaction.
著者: Park, Y.C. / Ye, H. / Hsia, C. / Segal, D. / Rich, R.L. / Liou, H.C. / Myszka, D.G. / Wu, H. |
|---|
| 履歴 | | 登録 | 2000年6月6日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2000年9月6日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2012年5月16日 | Group: Other |
|---|
| 改定 1.4 | 2017年10月4日 | Group: Refinement description / カテゴリ: software |
|---|
| 改定 1.5 | 2024年11月13日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / struct_conn / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2 Å
シンクロトロン / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell(Cambridge,Mass.) / 年: 2000
ジャーナル: Cell(Cambridge,Mass.) / 年: 2000 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1f3v.cif.gz
1f3v.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1f3v.ent.gz
pdb1f3v.ent.gz PDB形式
PDB形式 1f3v.json.gz
1f3v.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1f3v_validation.pdf.gz
1f3v_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1f3v_full_validation.pdf.gz
1f3v_full_validation.pdf.gz 1f3v_validation.xml.gz
1f3v_validation.xml.gz 1f3v_validation.cif.gz
1f3v_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/f3/1f3v
https://data.pdbj.org/pub/pdb/validation_reports/f3/1f3v ftp://data.pdbj.org/pub/pdb/validation_reports/f3/1f3v
ftp://data.pdbj.org/pub/pdb/validation_reports/f3/1f3v リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / プラスミド: PET / 発現宿主:
Homo sapiens (ヒト) / プラスミド: PET / 発現宿主: 
 Homo sapiens (ヒト) / プラスミド: PET / 発現宿主:
Homo sapiens (ヒト) / プラスミド: PET / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X4A / 波長: 0.9
/ ビームライン: X4A / 波長: 0.9  解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj














