+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1dct | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | DNA (CYTOSINE-5) METHYLASE FROM HAEIII COVALENTLY BOUND TO DNA | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSFERASE/DNA / ENZYME / CYTOSINE METHYLASE / TRANSFERASE-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA (cytosine-5-)-methyltransferase / DNA (cytosine-5-)-methyltransferase activity / DNA restriction-modification system / methylation / DNA binding / ATP binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Haemophilus influenzae biotype aegyptius (インフルエンザ菌) Haemophilus influenzae biotype aegyptius (インフルエンザ菌) | ||||||
| 手法 |  X線回折 / 解像度: 2.8 Å X線回折 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Reinisch, K.M. / Chen, L. / Verdine, G.L. / Lipscomb, W.N. | ||||||
 引用 引用 |  ジャーナル: Cell(Cambridge,Mass.) / 年: 1995 ジャーナル: Cell(Cambridge,Mass.) / 年: 1995タイトル: The crystal structure of HaeIII methyltransferase convalently complexed to DNA: an extrahelical cytosine and rearranged base pairing. 著者: Reinisch, K.M. / Chen, L. / Verdine, G.L. / Lipscomb, W.N. #1:  ジャーナル: J.Mol.Biol. / 年: 1994 ジャーナル: J.Mol.Biol. / 年: 1994タイトル: Crystallization and Prelimanary Crystallographic Analysis of a DNA (Cytosine-5) -Methyltransferase from Haemophilus Aegyptius Bound Covalently to DNA 著者: Reinisch, K.M. / Chen, L. / Verdine, G.L. / Lipscomb, W.N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1dct.cif.gz 1dct.cif.gz | 178.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1dct.ent.gz pdb1dct.ent.gz | 145.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1dct.json.gz 1dct.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1dct_validation.pdf.gz 1dct_validation.pdf.gz | 406.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1dct_full_validation.pdf.gz 1dct_full_validation.pdf.gz | 460.5 KB | 表示 | |
| XML形式データ |  1dct_validation.xml.gz 1dct_validation.xml.gz | 22 KB | 表示 | |
| CIF形式データ |  1dct_validation.cif.gz 1dct_validation.cif.gz | 32.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dc/1dct https://data.pdbj.org/pub/pdb/validation_reports/dc/1dct ftp://data.pdbj.org/pub/pdb/validation_reports/dc/1dct ftp://data.pdbj.org/pub/pdb/validation_reports/dc/1dct | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 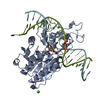
| ||||||||||
| 2 | 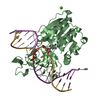
| ||||||||||
| 単位格子 |
| ||||||||||
| 詳細 | THIS FILE CONTAINS 2 PROTEIN-DNA COMPLEXES IN THE ASYMMETRIC UNIT. PROTEIN MONOMER A IS COVALENTLY BOUND TO A DNA DUPLEX CONSISTING OF CHAINS F AND M, AND PROTEIN MONOMER B IS COVALENTLY LINKED TO A DUPLEX CONSISTING OF CHAINS G AND N. THERE ARE TWO SPECIAL NUCLEOTIDE BASES INCORPORATED INTO EACH DNA DUPLEX - ONE IRREGULAR BASE IN EACH CHAIN. ONE OF THESE BASES IS A CYTOSINE METHYLATED AT THE 5-POSITION, AND THE OTHER (DESCRIBED IN MORE DETAIL BELOW) IS USED TO COVALENTLY LINK THE DNA TO THE PROTEIN. THE DNA AT THE RECOGNITION SITE IS VERY DISTORTED: THERE IS AN EXTRAHELICAL CYTOSINE (THE MODIFIED ONE COVALENTLY LINKED TO THE PROTEIN VIA CYS 71: +C F 10 AND +C G 10) AND BASE PAIRING IN THE DNA RECOGNITION SEQUENCE IS REORGANIZED. THERE ARE ALSO TWO CA+2 IONS IN THE ASYMMETRIC UNIT. |
- 要素
要素
| #1: DNA鎖 | 分子量: 5559.653 Da / 分子数: 2 / 由来タイプ: 合成 #2: DNA鎖 | 分子量: 5553.590 Da / 分子数: 2 / 由来タイプ: 合成 #3: タンパク質 | 分子量: 37093.559 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Haemophilus influenzae biotype aegyptius (インフルエンザ菌) Haemophilus influenzae biotype aegyptius (インフルエンザ菌)生物種: Haemophilus influenzae / 株: biotype aegyptius / 参照: UniProt: P20589, EC: 2.1.1.73 #4: 化合物 | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.48 Å3/Da / 溶媒含有率: 45 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.5 詳細: pH 6.50, VAPOR DIFFUSION, HANGING DROP, temperature 277.00K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS pH: 6.5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 113 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU / 波長: 1.5418 回転陽極 / タイプ: RIGAKU / 波長: 1.5418 |
| 検出器 | タイプ: RIGAKU RAXIS II / 検出器: IMAGE PLATE |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 最高解像度: 2.7 Å / Num. all: 129524 / Num. obs: 26866 / % possible obs: 95 % / Rmerge(I) obs: 0.076 |
| 反射 | *PLUS 最高解像度: 2.7 Å / % possible obs: 95 % / Observed criterion σ(I): 1 / Num. measured all: 129524 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.8→10 Å / 交差検証法: THROUGHOUT / σ(F): 2 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati sigma a obs: 0.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / 分類: refinement X-PLOR / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.8 Å / 最低解像度: 10 Å / σ(F): 2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj








































