+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1d2u | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | 1.15 A CRYSTAL STRUCTURE OF NITROPHORIN 4 FROM RHODNIUS PROLIXUS | ||||||
 要素 要素 | NITROPHORIN 4 | ||||||
 キーワード キーワード | TRANSPORT PROTEIN / NITRIC OXIDE TRANSPORT / FERRIC HEME / ANTIHISTAMINE / VASODILATOR / LIPOCALIN / BILAN BINDING PROTEIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nitrite dismutase / histamine binding / nitric oxide binding / vasodilation / oxidoreductase activity / extracellular region / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.15 Å シンクロトロン / 解像度: 1.15 Å | ||||||
 データ登録者 データ登録者 | Weichsel, A. / Andersen, J.F. / Roberts, S.A. / Montfort, W.R. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2001 ジャーナル: Biochemistry / 年: 2001タイトル: Ligand-induced heme ruffling and bent no geometry in ultra-high-resolution structures of nitrophorin 4. 著者: Roberts, S.A. / Weichsel, A. / Qiu, Y. / Shelnutt, J.A. / Walker, F.A. / Montfort, W.R. | ||||||
| 履歴 |
|
- 構造の表示
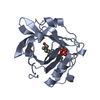
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1d2u.cif.gz 1d2u.cif.gz | 93.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1d2u.ent.gz pdb1d2u.ent.gz | 70.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1d2u.json.gz 1d2u.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1d2u_validation.pdf.gz 1d2u_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1d2u_full_validation.pdf.gz 1d2u_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  1d2u_validation.xml.gz 1d2u_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  1d2u_validation.cif.gz 1d2u_validation.cif.gz | 19.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/d2/1d2u https://data.pdbj.org/pub/pdb/validation_reports/d2/1d2u ftp://data.pdbj.org/pub/pdb/validation_reports/d2/1d2u ftp://data.pdbj.org/pub/pdb/validation_reports/d2/1d2u | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||||||||
| 単位格子 |
| |||||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 20292.664 Da / 分子数: 1 / 断片: RESIDUES 22-205 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
| #2: 化合物 | ChemComp-HEM / |
| #3: 化合物 | ChemComp-NH3 / |
| #4: 水 | ChemComp-HOH / |
| Has protein modification | Y |
| 非ポリマーの詳細 | Ammonia (NH3) occupies the Fe sixth coordination position The heme is disordered by a rotation of ...Ammonia (NH3) occupies the Fe sixth coordination position The heme is disordered by a rotation of 180 degrees around the CHA-Fe-CHC axis. The only evidence of this disorder is the appearence of methyl groups CMB and CMC as vinyls. These extra atoms are called CBBB and CBCB. THE LOOP CONTAINING |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.94 Å3/Da / 溶媒含有率: 36.44 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 3.0 M AMMONIUM PHOSPHATE 10 MM TRIS-HCL, pH 7.5, VAPOR DIFFUSION, HANGING DROP, temperature 298K | |||||||||||||||||||||||||
| 結晶化 | *PLUS pH: 5.6 / 詳細: Weichsel, A., (2000) Nat. Struct. Biol., 7, 551. | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 105 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: BM14 / 波長: 0.8727 / ビームライン: BM14 / 波長: 0.8727 |
| 検出器 | タイプ: MARRESEARCH / 検出器: AREA DETECTOR / 日付: 1998年12月11日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.8727 Å / 相対比: 1 |
| 反射 | 解像度: 1.15→52 Å / Num. all: 49629 / Num. obs: 49629 / % possible obs: 90 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 2.4 % / Biso Wilson estimate: 10.5 Å2 / Rmerge(I) obs: 0.04 / Net I/σ(I): 22.4 |
| 反射 シェル | 解像度: 1.15→1.19 Å / 冗長度: 1.7 % / Rmerge(I) obs: 0.26 / % possible all: 74 |
| 反射 | *PLUS % possible obs: 90 % / Num. measured all: 118648 |
| 反射 シェル | *PLUS % possible obs: 74 % |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.15→52 Å / Num. parameters: 15661 / Num. restraintsaints: 18721 / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: ENGH & HUBER 詳細: SHELXL97 REFINEMENT. FE-HEME AND FE-LIGAND DISTANCES AND ANGLES WERE UNRESTRAINED.
| |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 6 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.15→52 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL-97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 52 Å / σ(F): 0 / % reflection Rfree: 5 % / Rfactor all: 0.13 / Rfactor Rfree: 0.19 / Rfactor Rwork: 0.13 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj