| 登録情報 | データベース: PDB / ID: 1co1
|
|---|
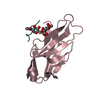
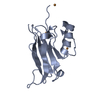
| タイトル | FOLD OF THE CBFA |
|---|
 要素 要素 | CORE BINDING FACTOR ALPHA |
|---|
 キーワード キーワード | GENE REGULATION / CBFA RUNT AML-1 RUNT DOMAIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of connective tissue replacement / peripheral nervous system neuron development / positive regulation of granulocyte differentiation / RUNX1 regulates transcription of genes involved in BCR signaling / SLC-mediated transport of organic cations / RUNX1 regulates transcription of genes involved in interleukin signaling / core-binding factor complex / RUNX1 regulates expression of components of tight junctions / positive regulation of CD8-positive, alpha-beta T cell differentiation / myeloid leukocyte differentiation ...regulation of connective tissue replacement / peripheral nervous system neuron development / positive regulation of granulocyte differentiation / RUNX1 regulates transcription of genes involved in BCR signaling / SLC-mediated transport of organic cations / RUNX1 regulates transcription of genes involved in interleukin signaling / core-binding factor complex / RUNX1 regulates expression of components of tight junctions / positive regulation of CD8-positive, alpha-beta T cell differentiation / myeloid leukocyte differentiation / regulation of cardiac muscle cell proliferation / cardiac muscle tissue regeneration / negative regulation of CD4-positive, alpha-beta T cell differentiation / negative regulation of granulocyte differentiation / RUNX1 and FOXP3 control the development of regulatory T lymphocytes (Tregs) / positive regulation of extracellular matrix organization / RUNX2 regulates genes involved in differentiation of myeloid cells / regulation of plasminogen activation / RUNX1 regulates transcription of genes involved in differentiation of keratinocytes / myeloid cell differentiation / Regulation of RUNX1 Expression and Activity / RUNX1 regulates transcription of genes involved in differentiation of myeloid cells / RUNX1 regulates transcription of genes involved in WNT signaling / RUNX1 regulates estrogen receptor mediated transcription / hematopoietic stem cell proliferation / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / regulation of cell differentiation / positive regulation of collagen biosynthetic process / hemopoiesis / RUNX3 regulates p14-ARF / chondrocyte differentiation / positive regulation of interleukin-2 production / ossification / transcription corepressor binding / RNA polymerase II transcription regulatory region sequence-specific DNA binding / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Pre-NOTCH Transcription and Translation / transcription coactivator binding / Transcriptional regulation of granulopoiesis / neuron differentiation / positive regulation of angiogenesis / SARS-CoV-1 activates/modulates innate immune responses / RUNX1 regulates transcription of genes involved in differentiation of HSCs / DNA-binding transcription activator activity, RNA polymerase II-specific / Estrogen-dependent gene expression / DNA-binding transcription factor binding / DNA-binding transcription factor activity, RNA polymerase II-specific / transcription cis-regulatory region binding / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein heterodimerization activity / intracellular membrane-bounded organelle / calcium ion binding / regulation of transcription by RNA polymerase II / chromatin / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / DNA binding / nucleoplasm / ATP binding / nucleus類似検索 - 分子機能 Runx, central domain superfamily / Acute myeloid leukemia 1 protein (AML1)/Runt / Runt domain / Runx, C-terminal domain / Runt-related transcription factor RUNX / Runt domain / Runx inhibition domain / Runt domain profile. / Immunoglobulin-like - #720 / p53/RUNT-type transcription factor, DNA-binding domain superfamily ...Runx, central domain superfamily / Acute myeloid leukemia 1 protein (AML1)/Runt / Runt domain / Runx, C-terminal domain / Runt-related transcription factor RUNX / Runt domain / Runx inhibition domain / Runt domain profile. / Immunoglobulin-like - #720 / p53/RUNT-type transcription factor, DNA-binding domain superfamily / p53-like transcription factor, DNA-binding / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR |
|---|
 データ登録者 データ登録者 | Berardi, M.J. / Bushweller, J.H. |
|---|
 引用 引用 |  ジャーナル: Structure Fold.Des. / 年: 1999 ジャーナル: Structure Fold.Des. / 年: 1999
タイトル: The Ig fold of the core binding factor alpha Runt domain is a member of a family of structurally and functionally related Ig-fold DNA-binding domains.
著者: Berardi, M.J. / Sun, C. / Zehr, M. / Abildgaard, F. / Peng, J. / Speck, N.A. / Bushweller, J.H. |
|---|
| 履歴 | | 登録 | 1999年5月31日 | 登録サイト: BNL / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2000年6月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月26日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年2月16日 | Group: Database references / Derived calculations
カテゴリ: database_2 / pdbx_struct_assembly / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 1.4 | 2023年12月27日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Structure Fold.Des. / 年: 1999
ジャーナル: Structure Fold.Des. / 年: 1999 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1co1.cif.gz
1co1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1co1.ent.gz
pdb1co1.ent.gz PDB形式
PDB形式 1co1.json.gz
1co1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1co1_validation.pdf.gz
1co1_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1co1_full_validation.pdf.gz
1co1_full_validation.pdf.gz 1co1_validation.xml.gz
1co1_validation.xml.gz 1co1_validation.cif.gz
1co1_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/co/1co1
https://data.pdbj.org/pub/pdb/validation_reports/co/1co1 ftp://data.pdbj.org/pub/pdb/validation_reports/co/1co1
ftp://data.pdbj.org/pub/pdb/validation_reports/co/1co1 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 発現宿主:
Homo sapiens (ヒト) / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj











