+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1bor | ||||||
|---|---|---|---|---|---|---|---|
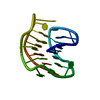
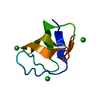
| タイトル | TRANSCRIPTION FACTOR PML, A PROTO-ONCOPROTEIN, NMR, 1 REPRESENTATIVE STRUCTURE AT PH 7.5, 30 C, IN THE PRESENCE OF ZINC | ||||||
 要素 要素 | TRANSCRIPTION FACTOR PML | ||||||
 キーワード キーワード | TRANSCRIPTION REGULATION / PROTO-ONCOGENE / NUCLEAR BODIES (PODS) / LEUKEMIA | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of calcium ion transport into cytosol / ubiquitin-like protein ligase activity / negative regulation of translation in response to oxidative stress / positive regulation of protein localization to chromosome, telomeric region / suppression of viral release by host / PML body organization / SUMO binding / fibroblast migration / positive regulation of apoptotic process involved in mammary gland involution / SMAD protein signal transduction ...regulation of calcium ion transport into cytosol / ubiquitin-like protein ligase activity / negative regulation of translation in response to oxidative stress / positive regulation of protein localization to chromosome, telomeric region / suppression of viral release by host / PML body organization / SUMO binding / fibroblast migration / positive regulation of apoptotic process involved in mammary gland involution / SMAD protein signal transduction / myeloid cell differentiation / maintenance of protein location in nucleus / protein-containing complex localization / regulation of double-strand break repair / endoplasmic reticulum calcium ion homeostasis / oncogene-induced cell senescence / branching involved in mammary gland duct morphogenesis / positive regulation of extrinsic apoptotic signaling pathway / Regulation of RUNX1 Expression and Activity / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / SUMO transferase activity / cobalt ion binding / negative regulation of interleukin-1 beta production / intrinsic apoptotic signaling pathway in response to oxidative stress / SUMOylation of ubiquitinylation proteins / entrainment of circadian clock by photoperiod / SMAD binding / negative regulation of telomere maintenance via telomerase / positive regulation of telomere maintenance / protein sumoylation / positive regulation of signal transduction by p53 class mediator / negative regulation of mitotic cell cycle / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / negative regulation of ubiquitin-dependent protein catabolic process / cell fate commitment / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / protein targeting / regulation of cell adhesion / SUMOylation of DNA damage response and repair proteins / retinoic acid receptor signaling pathway / response to UV / extrinsic apoptotic signaling pathway / Regulation of TP53 Activity through Acetylation / positive regulation of defense response to virus by host / response to cytokine / transforming growth factor beta receptor signaling pathway / cellular response to interleukin-4 / cellular response to leukemia inhibitory factor / Regulation of PTEN localization / DNA damage response, signal transduction by p53 class mediator / negative regulation of angiogenesis / response to gamma radiation / circadian regulation of gene expression / regulation of circadian rhythm / negative regulation of cell growth / PML body / nuclear matrix / Transcriptional regulation of granulopoiesis / HCMV Early Events / positive regulation of fibroblast proliferation / intrinsic apoptotic signaling pathway in response to DNA damage / protein import into nucleus / Interferon gamma signaling / cellular senescence / protein-containing complex assembly / early endosome membrane / molecular adaptor activity / nuclear membrane / proteasome-mediated ubiquitin-dependent protein catabolic process / transcription coactivator activity / chromosome, telomeric region / response to hypoxia / regulation of cell cycle / protein stabilization / chromatin remodeling / protein heterodimerization activity / negative regulation of cell population proliferation / innate immune response / negative regulation of DNA-templated transcription / apoptotic process / ubiquitin protein ligase binding / endoplasmic reticulum membrane / regulation of DNA-templated transcription / nucleolus / protein homodimerization activity / DNA binding / zinc ion binding / nucleoplasm / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR | ||||||
 データ登録者 データ登録者 | Borden, K.L.B. / Freemont, P.S. | ||||||
 引用 引用 |  ジャーナル: EMBO J. / 年: 1995 ジャーナル: EMBO J. / 年: 1995タイトル: The solution structure of the RING finger domain from the acute promyelocytic leukaemia proto-oncoprotein PML. 著者: Borden, K.L. / Boddy, M.N. / Lally, J. / O'Reilly, N.J. / Martin, S. / Howe, K. / Solomon, E. / Freemont, P.S. #1:  ジャーナル: Ann.N.Y.Acad.Sci. / 年: 1993 ジャーナル: Ann.N.Y.Acad.Sci. / 年: 1993タイトル: The Ring Finger. A Novel Protein Sequence Motif Related to the Zinc Finger 著者: Freemont, P.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1bor.cif.gz 1bor.cif.gz | 20 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1bor.ent.gz pdb1bor.ent.gz | 10.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1bor.json.gz 1bor.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bo/1bor https://data.pdbj.org/pub/pdb/validation_reports/bo/1bor ftp://data.pdbj.org/pub/pdb/validation_reports/bo/1bor ftp://data.pdbj.org/pub/pdb/validation_reports/bo/1bor | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6113.134 Da / 分子数: 1 / 断片: RING FINGER DOMAIN, RESIDUES 49 - 104 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 参照: UniProt: P29590 Homo sapiens (ヒト) / 参照: UniProt: P29590 |
|---|---|
| #2: 化合物 |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|
- 試料調製
試料調製
| 試料状態 | pH: 7.5 / 温度: 303 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
- 解析
解析
| NMRアンサンブル | コンフォーマー選択の基準: REPRESENTATIVE STRUCTURE 登録したコンフォーマーの数: 1 |
|---|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj















