+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1b11 | ||||||
|---|---|---|---|---|---|---|---|
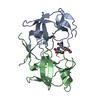
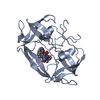
| タイトル | STRUCTURE OF FELINE IMMUNODEFICIENCY VIRUS PROTEASE COMPLEXED WITH TL-3-093 | ||||||
 要素 要素 | PROTEIN (Feline Immunodeficiency Virus PROTEASE) | ||||||
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / FIV PROTEASE / HYDROLASE-HYDROLASE INHIBITOR COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dUTP catabolic process / dUMP biosynthetic process / dUTP diphosphatase / dUTP diphosphatase activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / DNA integration / viral genome integration into host DNA ...dUTP catabolic process / dUMP biosynthetic process / dUTP diphosphatase / dUTP diphosphatase activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / DNA recombination / aspartic-type endopeptidase activity / 加水分解酵素; エステル加水分解酵素 / symbiont entry into host cell / magnesium ion binding / proteolysis / DNA binding / zinc ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Feline immunodeficiency virus (ネコ免疫不全ウイルス) Feline immunodeficiency virus (ネコ免疫不全ウイルス) | ||||||
| 手法 |  X線回折 / OTHER / 解像度: 1.9 Å X線回折 / OTHER / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Gustchina, A. / Li, M. / Wlodawer, A. | ||||||
 引用 引用 |  ジャーナル: Proteins / 年: 2000 ジャーナル: Proteins / 年: 2000タイトル: Structural studies of FIV and HIV-1 proteases complexed with an efficient inhibitor of FIV protease 著者: Li, M. / Morris, G.M. / Lee, T. / Laco, G.S. / Wong, C.H. / Olson, A.J. / Elder, J.H. / Wlodawer, A. / Gustchina, A. #1:  ジャーナル: Nat.Struct.Biol. / 年: 1995 ジャーナル: Nat.Struct.Biol. / 年: 1995タイトル: Structural Studies of HIV and Fiv Proteases Complexed with an Efficient Inhibitor of Fiv Pr 著者: Wlodawer, A. / Gustchina, A. / Reshetnikova, L. / Lubkowski, J. / Zdanov, A. / Hui, K. / Angleton, E. / Farmerie, W. / Goodenow, M. / Bhatt, D. / Zhang, L. / Dunn, B. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1b11.cif.gz 1b11.cif.gz | 39.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1b11.ent.gz pdb1b11.ent.gz | 26.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1b11.json.gz 1b11.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1b11_validation.pdf.gz 1b11_validation.pdf.gz | 708.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1b11_full_validation.pdf.gz 1b11_full_validation.pdf.gz | 710.8 KB | 表示 | |
| XML形式データ |  1b11_validation.xml.gz 1b11_validation.xml.gz | 8.1 KB | 表示 | |
| CIF形式データ |  1b11_validation.cif.gz 1b11_validation.cif.gz | 10.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b1/1b11 https://data.pdbj.org/pub/pdb/validation_reports/b1/1b11 ftp://data.pdbj.org/pub/pdb/validation_reports/b1/1b11 ftp://data.pdbj.org/pub/pdb/validation_reports/b1/1b11 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 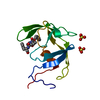
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 12842.825 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Feline immunodeficiency virus (ネコ免疫不全ウイルス) Feline immunodeficiency virus (ネコ免疫不全ウイルス)属: Lentivirus / 参照: UniProt: P16088, HIV-1 retropepsin | ||||||
|---|---|---|---|---|---|---|---|
| #2: 化合物 | | #3: 化合物 | ChemComp-3TL / | #4: 水 | ChemComp-HOH / | 非ポリマーの詳細 | THE INHIBITOR IS A C2 SYMMETRIC HIV PROTEASE | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.2 Å3/Da / 溶媒含有率: 44.4 % | |||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 5 詳細: SAMPLE: 3.4MG/ML FIVPR IN 25MM IMIDAZOL BUFFER AT PH 7.0 WITH 1MM EDTA AND 10MM DTT. WELL SOLUTION: 1.6M AMMONIUM SULFATE IN SODIUM ACETATE BUFFER AT PH 4.5 MIXING SAMPLE AND WELL SOLLUTION 1:1, pH 5.0 PH範囲: 4.5-7.0 | |||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / pH: 7 / 手法: 蒸気拡散法, ハンギングドロップ法 | |||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 297 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: ENRAF-NONIUS FR591 / 波長: 1.5418 回転陽極 / タイプ: ENRAF-NONIUS FR591 / 波長: 1.5418 |
| 検出器 | タイプ: MAC Science DIP-2020 / 検出器: IMAGE PLATE / 日付: 1997年9月1日 / 詳細: MIRRORS |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.9→20 Å / Num. obs: 9018 / % possible obs: 99.5 % / Observed criterion σ(I): -3 / 冗長度: 4.6 % / Rsym value: 0.083 / Net I/σ(I): 9.5 |
| 反射 シェル | 解像度: 1.9→1.93 Å / Mean I/σ(I) obs: 1.92 / Rsym value: 0.692 / % possible all: 99.5 |
| 反射 | *PLUS 最低解像度: 10 Å / Rmerge(I) obs: 0.083 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: OTHER / 解像度: 1.9→10 Å / Num. parameters: 4117 / Num. restraintsaints: 3974 / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER 詳細: ANISOTROPIC SCALING APPLIED BY METHOD PF PARKIN, MOEZZI & HOPE, J.APPL.CRYST.28(1995)53-56
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 5 / Occupancy sum hydrogen: 0 / Occupancy sum non hydrogen: 1002 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→10 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL / バージョン: 97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor all: 0.179 / Rfactor obs: 0.176 / Rfactor Rfree: 0.251 / Rfactor Rwork: 0.176 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj