+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1qd7 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
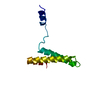
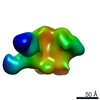
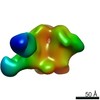
| タイトル | PARTIAL MODEL FOR 30S RIBOSOMAL SUBUNIT | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | RIBOSOME / 30S RIBOSOMAL SUBUNIT / LOW RESOLUTION MODEL | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribosomal small subunit biogenesis / small ribosomal subunit / cytosolic small ribosomal subunit / tRNA binding / rRNA binding / ribosome / structural constituent of ribosome / ribonucleoprotein complex / translation / response to antibiotic / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 5.5 Å 多波長異常分散 / 解像度: 5.5 Å | |||||||||
 データ登録者 データ登録者 | Clemons Jr., W.M. / May, J.L.C. / Wimberly, B.T. / McCutcheon, J.P. / Capel, M.S. / Ramakrishnan, V. | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 1999 ジャーナル: Nature / 年: 1999タイトル: Structure of a bacterial 30S ribosomal subunit at 5.5 A resolution. 著者: Clemons Jr., W.M. / May, J.L. / Wimberly, B.T. / McCutcheon, J.P. / Capel, M.S. / Ramakrishnan, V. #1:  ジャーナル: Embo J. / 年: 1998 ジャーナル: Embo J. / 年: 1998タイトル: The crystal structure of ribosomal protein S4 reveals a two-domain molecule with an extensive RNA-binding surface: one domain shows structural homology to the ETS DNA-binding motif 著者: Davies, C. / Gerstner, R.B. / Draper, D.E. / Ramakrishnan, V. / White, S.W. #2:  ジャーナル: Nature / 年: 1992 ジャーナル: Nature / 年: 1992タイトル: The structure of ribosomal protein S5 reveals sites of interaction with 16S rRNA 著者: Ramakrishnan, V. / White, S.W. #3:  ジャーナル: Embo J. / 年: 1994 ジャーナル: Embo J. / 年: 1994タイトル: Crystal structure of the ribosomal protein S6 from Thermus thermophilus 著者: Lindahl, M. / Svensson, L.A. / Liljas, A. / Sedelnikova, I.A. / Eliseikina, I.A. / Fomenkova, N.P. / Nevskaya, N. / Nikonov, S.V. / Garber, M.B. / Muranova, T.A. / Rykonova, A.I. / Amons, R. #4:  ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: The structure of ribosomal protein S7 at 1.9 A resolution reveals a beta- hairpin motif that binds double-stranded nucleic acids 著者: Wimberly, B.T. / White, S.W. / Ramakrishnan, V. #5:  ジャーナル: J.Mol.Biol. / 年: 1998 ジャーナル: J.Mol.Biol. / 年: 1998タイトル: Crystal structure of ribosomal protein S8 from Thermus thermophilus reveals a high degree of structural conservation of a specific RNA binding site 著者: Nevskaya, N. / Tischenko, S. / Nikulin, A. / Al-Karadaghi, S. / Liljas, A. / Ehresmann, B. / Ehresmann, C. / Garber, M. / Nikonov, S. #6:  ジャーナル: Structure / 年: 1998 ジャーナル: Structure / 年: 1998タイトル: Conformational variability of the N-terminal helix in the structure of ribosomal protein S15 著者: Clemons Jr., W.M. / Davies, C.R. / White, S.W. / Ramakrishnan, V. #7:  ジャーナル: Biochemistry / 年: 1996 ジャーナル: Biochemistry / 年: 1996タイトル: Solution structure of prokaryotic ribosomal protein S17 by high-resolution NMR spectroscopy 著者: Jaishree, T.N. / Ramakrishnan, V. / White, S.W. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1qd7.cif.gz 1qd7.cif.gz | 60.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1qd7.ent.gz pdb1qd7.ent.gz | 33.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1qd7.json.gz 1qd7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1qd7_validation.pdf.gz 1qd7_validation.pdf.gz | 360.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1qd7_full_validation.pdf.gz 1qd7_full_validation.pdf.gz | 360.4 KB | 表示 | |
| XML形式データ |  1qd7_validation.xml.gz 1qd7_validation.xml.gz | 1.2 KB | 表示 | |
| CIF形式データ |  1qd7_validation.cif.gz 1qd7_validation.cif.gz | 12.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qd/1qd7 https://data.pdbj.org/pub/pdb/validation_reports/qd/1qd7 ftp://data.pdbj.org/pub/pdb/validation_reports/qd/1qd7 ftp://data.pdbj.org/pub/pdb/validation_reports/qd/1qd7 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 単位格子 |
|
- 要素
要素
-RNA鎖 , 2種, 2分子 AB
| #1: RNA鎖 | 分子量: 53096.625 Da / 分子数: 1 / 断片: RESIDUES 563-912 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
|---|---|
| #2: RNA鎖 | 分子量: 17211.391 Da / 分子数: 1 / 断片: RESIDUES 1400-1500 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
-タンパク質 , 8種, 8分子 CDEFGHIJ
| #3: タンパク質 | 分子量: 18624.359 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) / 参照: UniProt: P81288 Thermus thermophilus (バクテリア) / 参照: UniProt: P81288 |
|---|---|
| #4: タンパク質 | 分子量: 15240.724 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) / 参照: UniProt: P02357 Thermus thermophilus (バクテリア) / 参照: UniProt: P02357 |
| #5: タンパク質 | 分子量: 11619.337 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) / 参照: UniProt: P23370, UniProt: Q5SLP8*PLUS Thermus thermophilus (バクテリア) / 参照: UniProt: P23370, UniProt: Q5SLP8*PLUS |
| #6: タンパク質 | 分子量: 15342.846 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) / 参照: UniProt: P17291 Thermus thermophilus (バクテリア) / 参照: UniProt: P17291 |
| #7: タンパク質 | 分子量: 15624.214 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) / 参照: UniProt: P24319, UniProt: P0DOY9*PLUS Thermus thermophilus (バクテリア) / 参照: UniProt: P24319, UniProt: P0DOY9*PLUS |
| #8: タンパク質 | 分子量: 10200.844 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) / 参照: UniProt: P05766 Thermus thermophilus (バクテリア) / 参照: UniProt: P05766 |
| #9: タンパク質 | 分子量: 10081.853 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) / 参照: UniProt: P23828 Thermus thermophilus (バクテリア) / 参照: UniProt: P23828 |
| #10: タンパク質 | 分子量: 8528.504 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 6 X線回折 / 使用した結晶の数: 6 |
|---|
- 試料調製
試料調製
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法 / 詳細: VAPOUR DIFFUSION AT 277 K, MPD, VAPOR DIFFUSION | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 溶液の組成 |
| ||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: Trakhanov, S.D., (1987) FEBS Lett., 220, 319. | ||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS NSLS  / ビームライン: X25 / 波長: 1.000, 1.700 / ビームライン: X25 / 波長: 1.000, 1.700 | |||||||||
| 検出器 | タイプ: BRANDEIS - B4 / 検出器: CCD / 日付: 1999年3月4日 | |||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||
| 放射波長 |
| |||||||||
| 反射 | 最高解像度: 5.5 Å / Num. all: 42000 / Num. obs: 42000 / % possible obs: 95 % / Observed criterion σ(I): 0 / 冗長度: 5 % | |||||||||
| 反射 シェル | 最高解像度: 5.5 Å / Mean I/σ(I) obs: 5.1 / % possible all: 83 | |||||||||
| 反射 | *PLUS |
- 解析
解析
| ソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 多波長異常分散開始モデル: PROTEINS WERE FIT INTO MAP AS RIGID BODIES FROM KNOWN CRYSTAL OR NMR STRUCTURES OF ISOLATED PROTEINS EXCEPT FOR S20, FOR WHICH NO HIGH RESOLUTION STRUCTURE EXISTS. S20 WAS FIT AS ...開始モデル: PROTEINS WERE FIT INTO MAP AS RIGID BODIES FROM KNOWN CRYSTAL OR NMR STRUCTURES OF ISOLATED PROTEINS EXCEPT FOR S20, FOR WHICH NO HIGH RESOLUTION STRUCTURE EXISTS. S20 WAS FIT AS THREE-HELIX BUNDLE.S4 SEE REFERENCE (1); S5 MODELED ACCORDING TO PDB CODE 1PKP;S6 MODELED ACCORDING TO PDB CODE 1RIS;S7 MODELED ACCORDING TO PDB CODE 1RSS;S8 MODELED ACCORDING TO PDB CODE 1AN7;S15 MODELED ACCORDING TO PDB CODE 1A23;S17 SEE REFERENCE (7); 最高解像度: 5.5 Å / Num. reflection all: 42000 / Num. reflection obs: 42000 詳細: NO REFINEMENT WAS DONE EXCEPT FOR VISUAL MAP FITTING. THIS MODEL WAS MADE FROM A 5.5 ANGSTROM X-RAY MAP BY FITTING A-FORM RNA HELICES INTO REGIONS OF DOUBLE- HELICAL DENSITY. NO ATTEMPT HAS ...詳細: NO REFINEMENT WAS DONE EXCEPT FOR VISUAL MAP FITTING. THIS MODEL WAS MADE FROM A 5.5 ANGSTROM X-RAY MAP BY FITTING A-FORM RNA HELICES INTO REGIONS OF DOUBLE- HELICAL DENSITY. NO ATTEMPT HAS BEEN MADE TO MAINTAIN PROPER STEREOCHEMISTRY OR EVEN PHOSPHATE-PHOSPHATE DISTANCES. IN PARTICULAR AT JUNCTIONS OF THE SHORT HELICES, THE CHAIN MAY BE UNREALISTIC | ||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 5.5 Å
| ||||||||||||
| 精密化 | *PLUS 最高解像度: 5.5 Å | ||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





























