+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9832 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tetrameric PepTSo2 incorporated in salipro nano particle | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Peptide transporter / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dipeptide transmembrane transport / tripeptide transmembrane transporter activity / peptide:proton symporter activity / dipeptide transmembrane transporter activity / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Shewanella oneidensis (バクテリア) / Shewanella oneidensis (バクテリア) /  Shewanella oneidensis MR-1 (バクテリア) Shewanella oneidensis MR-1 (バクテリア) | |||||||||
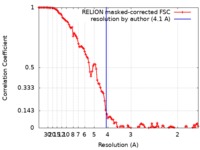
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Kawamoto A / Matoba K | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr F Struct Biol Commun / 年: 2019 ジャーナル: Acta Crystallogr F Struct Biol Commun / 年: 2019タイトル: Structural basis for oligomerization of the prokaryotic peptide transporter PepT. 著者: Reina Nagamura / Masahiro Fukuda / Akihiro Kawamoto / Kyoko Matoba / Naoshi Dohmae / Ryuichiro Ishitani / Junichi Takagi / Osamu Nureki /  要旨: Proton-dependent oligopeptide transporters (POTs) belong to the major facilitator superfamily (MFS) and transport dipeptides and tripeptides from the extracellular environment into the target cell. ...Proton-dependent oligopeptide transporters (POTs) belong to the major facilitator superfamily (MFS) and transport dipeptides and tripeptides from the extracellular environment into the target cell. The human POTs PepT1 and PepT2 are also involved in the absorption of various orally ingested drugs. Previously reported structures revealed that the bacterial POTs possess 14 helices, of which H1-H6 and H7-H12 constitute the typical MFS fold and the residual two helices are involved in the cytoplasmic linker. PepT from Shewanella oneidensis is a unique POT which reportedly assembles as a 200 kDa tetramer. Although the previously reported structures suggested the importance of H12 for tetramer formation, the structural basis for the PepT-specific oligomerization remains unclear owing to the lack of a high-resolution tetrameric structure. In this study, the expression and purification conditions for tetrameric PepT were optimized. A single-particle cryo-EM analysis revealed the tetrameric structure of PepT incorporated into Salipro nanoparticles at 4.1 Å resolution. Furthermore, a combination of lipidic cubic phase (LCP) crystallization and an automated data-processing system for multiple microcrystals enabled crystal structures of PepT to be determined at resolutions of 3.5 and 3.9 Å. The present structures in a lipid bilayer revealed the detailed mechanism for the tetrameric assembly of PepT, in which a characteristic extracellular loop (ECL) interacts with two asparagine residues on H12 which were reported to be important for tetramerization and plays an essential role in oligomeric assembly. This study provides valuable insights into the oligomerization mechanism of this MFS-type transporter, which will further pave the way for understanding other oligomeric membrane proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9832.map.gz emd_9832.map.gz | 9.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9832-v30.xml emd-9832-v30.xml emd-9832.xml emd-9832.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9832_fsc.xml emd_9832_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9832.png emd_9832.png | 112.8 KB | ||
| Filedesc metadata |  emd-9832.cif.gz emd-9832.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9832 http://ftp.pdbj.org/pub/emdb/structures/EMD-9832 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9832 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9832 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9832_validation.pdf.gz emd_9832_validation.pdf.gz | 415 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9832_full_validation.pdf.gz emd_9832_full_validation.pdf.gz | 414.6 KB | 表示 | |
| XML形式データ |  emd_9832_validation.xml.gz emd_9832_validation.xml.gz | 11.9 KB | 表示 | |
| CIF形式データ |  emd_9832_validation.cif.gz emd_9832_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9832 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9832 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9832 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9832 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9832.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9832.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tetrameric PepTSo2 with saposin A
| 全体 | 名称: Tetrameric PepTSo2 with saposin A |
|---|---|
| 要素 |
|
-超分子 #1: Tetrameric PepTSo2 with saposin A
| 超分子 | 名称: Tetrameric PepTSo2 with saposin A / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Shewanella oneidensis (バクテリア) Shewanella oneidensis (バクテリア) |
-分子 #1: Proton:oligopeptide symporter POT family
| 分子 | 名称: Proton:oligopeptide symporter POT family / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Shewanella oneidensis MR-1 (バクテリア) / 株: MR-1 Shewanella oneidensis MR-1 (バクテリア) / 株: MR-1 |
| 分子量 | 理論値: 57.626559 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTLGTNQVSK THSFMTVSLI ELWERFGYYG MQALIVYFMV QRLGFDDSRA NLVWSACAAL IYVSPAIGGW VGDKILGTKR TMLLGAGIL SVGYALMTVP TENTWFMFSA LGVIVVGNGL FKPNAGNLVR KIYEGDDSKI DSAFTIYYMA VNVGSTFSML L TPWIKDYV ...文字列: MTLGTNQVSK THSFMTVSLI ELWERFGYYG MQALIVYFMV QRLGFDDSRA NLVWSACAAL IYVSPAIGGW VGDKILGTKR TMLLGAGIL SVGYALMTVP TENTWFMFSA LGVIVVGNGL FKPNAGNLVR KIYEGDDSKI DSAFTIYYMA VNVGSTFSML L TPWIKDYV NAQYGNEFGW HAAFAVCCVG ILVGLGNYAL MHKSLANYGS EPDTRPVNKK SLAIVLALAA LSVVASAIIL EY EDVARVF VYAAGVAVLG IFFHLIRTSE PSERAGLIAA LILTVQTVFF FIFYQQMSTS LALFALRNVD WDFQVFGTHL WTW SPAQFQ ALNPIWIMVL SPVLAWSYSW AGRNNKDFSI AAKFALGFAV VAIGFFIYGF AGQFAVNGKT SSWVMIWGYA SYSL GELLV SGLGLAMIAR YVPARMGGFM MGAYFVASGI SQYLGGVVAN FASVPQDLVD PLQTLPVYTN LFNKLGVAAV VCTII ALAV LPLMRRLTES HHAHSSIENN AAASLRDVKA EQLESSGENL YFQ UniProtKB: Proton:oligopeptide symporter POT family |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blotted for 4.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 79.55 K / 最高: 79.55 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 実像数: 1609 / 平均電子線量: 82.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 96000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6ji1: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)