+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9654 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of type III-A Csm-NTR complex | |||||||||
 マップデータ マップデータ | Cryo-EM structure of type III-A Csm-NTR complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Csm complex / Type III-A / CRISPR-Cas system / RNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exonuclease activity / transferase activity / endonuclease activity / defense response to virus / RNA binding / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) /  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.16 Å | |||||||||
 データ登録者 データ登録者 | You L / Ma J | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2019 ジャーナル: Cell / 年: 2019タイトル: Structure Studies of the CRISPR-Csm Complex Reveal Mechanism of Co-transcriptional Interference. 著者: Lilan You / Jun Ma / Jiuyu Wang / Daria Artamonova / Min Wang / Liang Liu / Hua Xiang / Konstantin Severinov / Xinzheng Zhang / Yanli Wang /   要旨: Csm, a type III-A CRISPR-Cas interference complex, is a CRISPR RNA (crRNA)-guided RNase that also possesses target RNA-dependent DNase and cyclic oligoadenylate (cOA) synthetase activities. However, ...Csm, a type III-A CRISPR-Cas interference complex, is a CRISPR RNA (crRNA)-guided RNase that also possesses target RNA-dependent DNase and cyclic oligoadenylate (cOA) synthetase activities. However, the structural features allowing target RNA-binding-dependent activation of DNA cleavage and cOA generation remain unknown. Here, we report the structure of Csm in complex with crRNA together with structures of cognate or non-cognate target RNA bound Csm complexes. We show that depending on complementarity with the 5' tag of crRNA, the 3' anti-tag region of target RNA binds at two distinct sites of the Csm complex. Importantly, the interaction between the non-complementary anti-tag region of cognate target RNA and Csm1 induces a conformational change at the Csm1 subunit that allosterically activates DNA cleavage and cOA generation. Together, our structural studies provide crucial insights into the mechanistic processes required for crRNA-meditated sequence-specific RNA cleavage, RNA target-dependent non-specific DNA cleavage, and cOA generation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9654.map.gz emd_9654.map.gz | 28.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9654-v30.xml emd-9654-v30.xml emd-9654.xml emd-9654.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9654.png emd_9654.png | 64.1 KB | ||
| Filedesc metadata |  emd-9654.cif.gz emd-9654.cif.gz | 6.8 KB | ||
| その他 |  emd_9654_additional.map.gz emd_9654_additional.map.gz | 28.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9654 http://ftp.pdbj.org/pub/emdb/structures/EMD-9654 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9654 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9654 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9654_validation.pdf.gz emd_9654_validation.pdf.gz | 591.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9654_full_validation.pdf.gz emd_9654_full_validation.pdf.gz | 590.9 KB | 表示 | |
| XML形式データ |  emd_9654_validation.xml.gz emd_9654_validation.xml.gz | 5.7 KB | 表示 | |
| CIF形式データ |  emd_9654_validation.cif.gz emd_9654_validation.cif.gz | 6.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9654 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9654 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9654 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9654 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6iflMC  9653C  9655C  9656C  9657C  9658C  9659C  9660C  6ifkC  6ifnC  6ifrC  6ifuC  6ifyC  6ifzC  6ig0C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9654.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9654.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of type III-A Csm-NTR complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
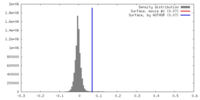
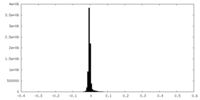
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Cryo-EM structure of type III-A Csm-NTR complex,subunit Csm1
| ファイル | emd_9654_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of type III-A Csm-NTR complex,subunit Csm1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
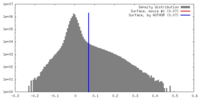
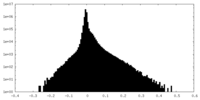
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Csm-CTR1 complex, AMPPNP bound
| 全体 | 名称: Csm-CTR1 complex, AMPPNP bound |
|---|---|
| 要素 |
|
-超分子 #1: Csm-CTR1 complex, AMPPNP bound
| 超分子 | 名称: Csm-CTR1 complex, AMPPNP bound / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: type III-A CRISPR-Cas interference complex |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 87 KDa |
-超分子 #2: Csm1
| 超分子 | 名称: Csm1 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 詳細: type III-A CRISPR-Cas interference complex subunit Csm1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
-分子 #1: Type III-A CRISPR-associated protein Csm1
| 分子 | 名称: Type III-A CRISPR-associated protein Csm1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 86.976734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKKEKIDLFY GALLHNIGKV IQRATGERKK HALVGADWFD EIADNQVISD QIRYHMANYQ SDKLGNDHLA YITYIADNIA SGVDRRQSN EESDEDTSAK IWDTYTNQAD IFNVFGAQTD KRYFKPTVLN LKSKPNFASA TYEPFSKGDY AAIATRIKNE L AEFEFNQV ...文字列: MKKEKIDLFY GALLHNIGKV IQRATGERKK HALVGADWFD EIADNQVISD QIRYHMANYQ SDKLGNDHLA YITYIADNIA SGVDRRQSN EESDEDTSAK IWDTYTNQAD IFNVFGAQTD KRYFKPTVLN LKSKPNFASA TYEPFSKGDY AAIATRIKNE L AEFEFNQV QIDSLLNLFE ATLSFVPSST NTKEIADISL ADHSRLTAAF ALAIYDYLED KGRHNYKEDL FTKVSAFYEE EA FLLASFD LSGIQDFIYN INIATNGAAK QLKARSLYLD FMSEYIADSL LDKLGLNRAN MLYVGGGHAY FVLANTEKTV ETL VQFEKD FNQFLLANFQ TRLYVAFGWG SFAAKDIMSE LNSPESYRQV YQKASRMISK KKISRYDYQT LMLLNRGGKS SERE CEICH SVENLVSYHD QKVCDICRGL YQFSKEIAHD HFIITENEGL PIGPNACLKG VAFEKLSQEA FSRVYVKNDY KAGTV KATH VFVGDYQCDE IYNYAALSKN ENGLGIKRLA VVRLDVDDLG AAFMAGFSQQ GNGQYSTLSR SATFSRSMSL FFKVYI NQF ASDKKLSIIY AGGDDVFAIG SWQDIIAFTV ELRENFIKWT NGKLTLSAGI GLFADKTPIS LMAHQTGELE EAAKGNE KD SISLFSSDYT FKFDRFITNV YDDKLEQIRY FFNHQDERGK NFIYKLIELL RNHDRMNMAR LAYYLTRLEE LTRETDRD K FKTFKNLFYS WYTNKNDKDR KEAELALLLY IYEIRKD UniProtKB: CRISPR system single-strand-specific deoxyribonuclease Cas10/Csm1 (subtype III-A) |
-分子 #2: Type III-A CRISPR-associated protein Csm2
| 分子 | 名称: Type III-A CRISPR-associated protein Csm2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 14.848145 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTILTDENYV DIAEKAILKL ERNTRNRKNP DAFFLTTSKL RNLLSLTSTL FDESKVKEYD ALLDRIAYLR VQFVYQAGRE IAVKDLIEK AQILEALKEI KDRETLQRFC RYMEALVAYF KFYGGKD UniProtKB: CRISPR system Cms protein Csm2 |
-分子 #3: Type III-A CRISPR-associated RAMP protein Csm3
| 分子 | 名称: Type III-A CRISPR-associated RAMP protein Csm3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 24.584855 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTFAKIKFSA QIRLETGLHI GGSDAFAAIG AINSPVIKDP ITNLPIIPGS SLKGKMRTLL AKVYNEKVAE KPSDDSDILS RLFGNSKDK RFKMGRLIFR DAFLSNADEL DSLGVRSYTE VKFENTIDRI TAEANPRQIE RAIRNSTFDF ELIYEITDEN E NQVEEDFK ...文字列: MTFAKIKFSA QIRLETGLHI GGSDAFAAIG AINSPVIKDP ITNLPIIPGS SLKGKMRTLL AKVYNEKVAE KPSDDSDILS RLFGNSKDK RFKMGRLIFR DAFLSNADEL DSLGVRSYTE VKFENTIDRI TAEANPRQIE RAIRNSTFDF ELIYEITDEN E NQVEEDFK VIRDGLKLLE LDYLGGSGSR GYGKVAFENL KATTVFGNYD VKTLNELLTA EV UniProtKB: CRISPR system Cms endoribonuclease Csm3 |
-分子 #4: Type III-A CRISPR-associated RAMP protein Csm4
| 分子 | 名称: Type III-A CRISPR-associated RAMP protein Csm4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 33.828984 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTYKLYIMTF QNAHFGSGTL DSSKLTFSAD RIFSALVLEA LKMGKLDAFL AEANQDKFTL TDAFPFQFGP FLPKPIGYPK HDQIDQSVD VKEVRRQAKL SKKLQFLALE NVDDYLNGEL FENEEHAVID TVTKNQPHKD DNLYQVATTR FSNDTSLYVI A NESDLLNE ...文字列: MTYKLYIMTF QNAHFGSGTL DSSKLTFSAD RIFSALVLEA LKMGKLDAFL AEANQDKFTL TDAFPFQFGP FLPKPIGYPK HDQIDQSVD VKEVRRQAKL SKKLQFLALE NVDDYLNGEL FENEEHAVID TVTKNQPHKD DNLYQVATTR FSNDTSLYVI A NESDLLNE LMSSLQYSGL GGKRSSGFGR FELDIQNIPL ELSDRLTKNH SDKVMSLTTA LPVDADLEEA MEDGHYLLTK SS GFAFSHA TNENYRKQDL YKFASGSTFS KTFEGQIVDV RPLDFPHAVL NYAKPLFFKL EV UniProtKB: CRISPR system Cms protein Csm4 |
-分子 #5: Type III-A CRISPR-associated RAMP protein Csm5
| 分子 | 名称: Type III-A CRISPR-associated RAMP protein Csm5 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 41.102113 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKNDYRTFKL SLLTLAPIHI GNGEKYTSRE FIYENKKFYF PDMGKFYNKM VEKRLAEKFE AFLIQTRPNA RNNRLISFLN DNRIAERSF GGYSISETGL ESDKNPNSAG AINEVNKFIR DAFGNPYIPG SSLKGAIRTI LMNTTPKWNN ENAVNDFGRF P KENKNLIP ...文字列: MKNDYRTFKL SLLTLAPIHI GNGEKYTSRE FIYENKKFYF PDMGKFYNKM VEKRLAEKFE AFLIQTRPNA RNNRLISFLN DNRIAERSF GGYSISETGL ESDKNPNSAG AINEVNKFIR DAFGNPYIPG SSLKGAIRTI LMNTTPKWNN ENAVNDFGRF P KENKNLIP WGPKKGKEYD DLFNAIRVSD SKPFDNKSLI LVQKWDYSAK TNKAKPLPLY RESISPLTKI EFEITTTTDE AG RLIEELG KRAQAFYKDY KAFFLSEFPD DKIQANLQYP IYLGAGSGAW TKTLFKQADG ILQRRYSRMK TKMVKKGVLK LTK APLKTV KIPSGNHSLV KNHESFYEMG KANFMIKEID K UniProtKB: CRISPR system Cms protein Csm5 |
-分子 #6: crRNA
| 分子 | 名称: crRNA / タイプ: rna / ID: 6 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 11.39277 KDa |
| 配列 | 文字列: ACGGAAACGC UUUCUAGCUC GCUAUAAUUA CCCAUU |
-分子 #7: NTR
| 分子 | 名称: NTR / タイプ: rna / ID: 7 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus ND03 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 13.937306 KDa |
| 配列 | 文字列: GGUAGGAAUG GGUAAUUAUA GCGAGCUAGA AAGCGUUUCC GUC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 60 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.16 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 133127 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)