+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8427 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Initial contact of HIV-1 Env with CD4: cryo-EM structure of BG505 DS-SOSIP trimer in complex with CD4 and antibody PGT145 | |||||||||
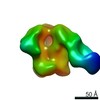
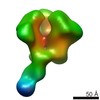
 マップデータ マップデータ | Initial contact of HIV-1 Env with CD4: cryo-EM structure of BG505 DS-SOSIP trimer in complex with CD4 and antibody PGT145 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報helper T cell enhancement of adaptive immune response / interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell / T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation ...helper T cell enhancement of adaptive immune response / interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell / T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation / Alpha-defensins / positive regulation of kinase activity / regulation of T cell activation / extracellular matrix structural constituent / T cell receptor complex / Other interleukin signaling / enzyme-linked receptor protein signaling pathway / Translocation of ZAP-70 to Immunological synapse / Phosphorylation of CD3 and TCR zeta chains / regulation of calcium ion transport / macrophage differentiation / Generation of second messenger molecules / T cell differentiation / PD-1 signaling / positive regulation of protein kinase activity / Binding and entry of HIV virion / coreceptor activity / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / cell surface receptor protein tyrosine kinase signaling pathway / positive regulation of establishment of T cell polarity / T cell activation / positive regulation of calcium-mediated signaling / positive regulation of interleukin-2 production / protein tyrosine kinase binding / host cell endosome membrane / Vpu mediated degradation of CD4 / calcium-mediated signaling / clathrin-coated endocytic vesicle membrane / transmembrane signaling receptor activity / positive regulation of peptidyl-tyrosine phosphorylation / positive regulation of T cell activation / Downstream TCR signaling / Cargo recognition for clathrin-mediated endocytosis / MHC class II protein complex binding / Clathrin-mediated endocytosis / virus receptor activity / signaling receptor activity / clathrin-dependent endocytosis of virus by host cell / positive regulation of canonical NF-kappaB signal transduction / defense response to Gram-negative bacterium / adaptive immune response / positive regulation of MAPK cascade / positive regulation of ERK1 and ERK2 cascade / positive regulation of viral entry into host cell / early endosome / cell surface receptor signaling pathway / viral protein processing / cell adhesion / immune response / positive regulation of protein phosphorylation / membrane raft / endoplasmic reticulum lumen / external side of plasma membrane / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / lipid binding / viral envelope / endoplasmic reticulum membrane / virion attachment to host cell / apoptotic process / protein kinase binding / host cell plasma membrane / structural molecule activity / positive regulation of DNA-templated transcription / virion membrane / enzyme binding / signal transduction / protein homodimerization activity / zinc ion binding / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
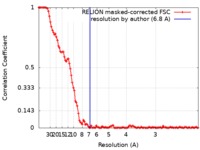
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.8 Å | |||||||||
 データ登録者 データ登録者 | Acharya P / Kwong PD / Potter CS / Carragher B | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2017 ジャーナル: Nat Struct Mol Biol / 年: 2017タイトル: Quaternary contact in the initial interaction of CD4 with the HIV-1 envelope trimer. 著者: Qingbo Liu / Priyamvada Acharya / Michael A Dolan / Peng Zhang / Christina Guzzo / Jacky Lu / Alice Kwon / Deepali Gururani / Huiyi Miao / Tatsiana Bylund / Gwo-Yu Chuang / Aliaksandr Druz / ...著者: Qingbo Liu / Priyamvada Acharya / Michael A Dolan / Peng Zhang / Christina Guzzo / Jacky Lu / Alice Kwon / Deepali Gururani / Huiyi Miao / Tatsiana Bylund / Gwo-Yu Chuang / Aliaksandr Druz / Tongqing Zhou / William J Rice / Christoph Wigge / Bridget Carragher / Clinton S Potter / Peter D Kwong / Paolo Lusso /  要旨: Binding of the gp120 envelope (Env) glycoprotein to the CD4 receptor is the first step in the HIV-1 infectious cycle. Although the CD4-binding site has been extensively characterized, the initial ...Binding of the gp120 envelope (Env) glycoprotein to the CD4 receptor is the first step in the HIV-1 infectious cycle. Although the CD4-binding site has been extensively characterized, the initial receptor interaction has been difficult to study because of major CD4-induced structural rearrangements. Here we used cryogenic electron microscopy (cryo-EM) to visualize the initial contact of CD4 with the HIV-1 Env trimer at 6.8-Å resolution. A single CD4 molecule is embraced by a quaternary HIV-1-Env surface formed by coalescence of the previously defined CD4-contact region with a second CD4-binding site (CD4-BS2) in the inner domain of a neighboring gp120 protomer. Disruption of CD4-BS2 destabilized CD4-trimer interaction and abrogated HIV-1 infectivity by preventing the acquisition of coreceptor-binding competence. A corresponding reduction in HIV-1 infectivity occurred after the mutation of CD4 residues that interact with CD4-BS2. Our results document the critical role of quaternary interactions in the initial HIV-Env-receptor contact, with implications for treatment and vaccine design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
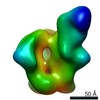
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8427.map.gz emd_8427.map.gz | 6.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8427-v30.xml emd-8427-v30.xml emd-8427.xml emd-8427.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8427_fsc.xml emd_8427_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8427.png emd_8427.png | 59.4 KB | ||
| その他 |  emd_8427_additional.map.gz emd_8427_additional.map.gz | 10.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8427 http://ftp.pdbj.org/pub/emdb/structures/EMD-8427 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8427 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8427 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8427.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8427.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Initial contact of HIV-1 Env with CD4: cryo-EM structure of BG505 DS-SOSIP trimer in complex with CD4 and antibody PGT145 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: HIV-1 Env with CD4: cryo-EM structure of BG505...
| ファイル | emd_8427_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HIV-1 Env with CD4: cryo-EM structure of BG505 DS-SOSIP trimer in complex with CD4 and antibody PGT145 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 Env trimer
| 全体 | 名称: HIV-1 Env trimer |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 Env trimer
| 超分子 | 名称: HIV-1 Env trimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 |
-超分子 #2: HIV-1 Env trimer in complex with antibody PGT145 and CD4
| 超分子 | 名称: HIV-1 Env trimer in complex with antibody PGT145 and CD4 タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: Antibody PGT145
| 超分子 | 名称: Antibody PGT145 / タイプ: complex / ID: 3 / 親要素: 2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 |
-超分子 #4: CD4
| 超分子 | 名称: CD4 / タイプ: complex / ID: 4 / 親要素: 2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 |
-超分子 #5: HIV-1 trimer
| 超分子 | 名称: HIV-1 trimer / タイプ: complex / ID: 5 / 親要素: 2 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 Homo sapiens (ヒト) / 組換プラスミド: pVRC8400 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
| ||||||
| グリッド | 材質: GOLD / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均露光時間: 10.0 sec. / 平均電子線量: 8.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 22000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation Coefficient |
|---|---|
| 得られたモデル |  PDB-5u1f: |
 ムービー
ムービー コントローラー
コントローラー




























 Z
Z Y
Y X
X