+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8403 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | sub-tomogram average of in vitro assembled HIV-1 Gag VLPs | |||||||||
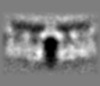
 マップデータ マップデータ | sub-Tomogram average of in vitro reconstituted HIV-1 virus-like particles imaged prior to protease treatement confirming immature conformation of the Gag lattice. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 18.0 Å | |||||||||
 データ登録者 データ登録者 | Himes BA / Zhang P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2016 ジャーナル: Nat Commun / 年: 2016タイトル: In vitro protease cleavage and computer simulations reveal the HIV-1 capsid maturation pathway. 著者: Jiying Ning / Gonca Erdemci-Tandogan / Ernest L Yufenyuy / Jef Wagner / Benjamin A Himes / Gongpu Zhao / Christopher Aiken / Roya Zandi / Peijun Zhang /   要旨: HIV-1 virions assemble as immature particles containing Gag polyproteins that are processed by the viral protease into individual components, resulting in the formation of mature infectious particles. ...HIV-1 virions assemble as immature particles containing Gag polyproteins that are processed by the viral protease into individual components, resulting in the formation of mature infectious particles. There are two competing models for the process of forming the mature HIV-1 core: the disassembly and de novo reassembly model and the non-diffusional displacive model. To study the maturation pathway, we simulate HIV-1 maturation in vitro by digesting immature particles and assembled virus-like particles with recombinant HIV-1 protease and monitor the process with biochemical assays and cryoEM structural analysis in parallel. Processing of Gag in vitro is accurate and efficient and results in both soluble capsid protein and conical or tubular capsid assemblies, seemingly converted from immature Gag particles. Computer simulations further reveal probable assembly pathways of HIV-1 capsid formation. Combining the experimental data and computer simulations, our results suggest a sequential combination of both displacive and disassembly/reassembly processes for HIV-1 maturation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8403.map.gz emd_8403.map.gz | 891.1 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8403-v30.xml emd-8403-v30.xml emd-8403.xml emd-8403.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8403.png emd_8403.png | 79.2 KB | ||
| その他 |  emd_8403_half_map_1.map.gz emd_8403_half_map_1.map.gz emd_8403_half_map_2.map.gz emd_8403_half_map_2.map.gz | 690.5 KB 711.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8403 http://ftp.pdbj.org/pub/emdb/structures/EMD-8403 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8403 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8403 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8403_validation.pdf.gz emd_8403_validation.pdf.gz | 386.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8403_full_validation.pdf.gz emd_8403_full_validation.pdf.gz | 386.4 KB | 表示 | |
| XML形式データ |  emd_8403_validation.xml.gz emd_8403_validation.xml.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8403 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8403 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8403 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8403 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8403.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8403.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sub-Tomogram average of in vitro reconstituted HIV-1 virus-like particles imaged prior to protease treatement confirming immature conformation of the Gag lattice. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.62 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: gold-standard half map used for FSC calculation, masked...
| ファイル | emd_8403_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
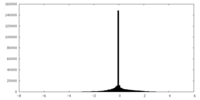
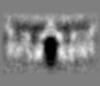
| 注釈 | gold-standard half map used for FSC calculation, masked by soft cylinder. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
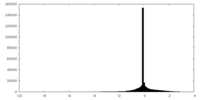
| 密度ヒストグラム |
-ハーフマップ: gold-standard half map used for FSC calculation, masked...
| ファイル | emd_8403_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
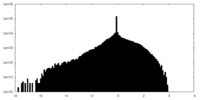
| 注釈 | gold-standard half map used for FSC calculation, masked by soft cylinder. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
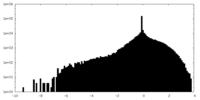
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : gag polyprotein, Pr55gag ([delta]-MA15-100[delta]p6)
| 全体 | 名称: gag polyprotein, Pr55gag ([delta]-MA15-100[delta]p6) |
|---|---|
| 要素 |
|
-超分子 #1: gag polyprotein, Pr55gag ([delta]-MA15-100[delta]p6)
| 超分子 | 名称: gag polyprotein, Pr55gag ([delta]-MA15-100[delta]p6) タイプ: complex / ID: 1 / 親要素: 0 詳細: PRR-55 construct, expressed recombinantly without protease. |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 39.9 KDa |
-分子 #1: HIV-1 Gag VLPs
| 分子 | 名称: HIV-1 Gag VLPs / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 配列 | 文字列: GARASVLSGG ELDRALDKIE EEQNKSKKKA QQAAADTGHS SQVSQNYPIV QNIQGQMVHQ AISPRTLNAW VKVVEEKAFS PEVIPMFSAL SEGATPQDLN TMLNTVGGHQ AAMQMLKETI NEEAAEWDRV HPVHAGPIAP GQMREPRGSD IAGTTSTLQE QIGWMTNNPP ...文字列: GARASVLSGG ELDRALDKIE EEQNKSKKKA QQAAADTGHS SQVSQNYPIV QNIQGQMVHQ AISPRTLNAW VKVVEEKAFS PEVIPMFSAL SEGATPQDLN TMLNTVGGHQ AAMQMLKETI NEEAAEWDRV HPVHAGPIAP GQMREPRGSD IAGTTSTLQE QIGWMTNNPP IPVGEIYKRW IILGLNKIVR MYSPTSILDI RQGPKEPFRD YVDRFYKTLR AEQASQEVKN WMTETLLVQN ANPDCKTILK ALGPAATLEE MMTACQGVGG PGHKARVLAE AMSQVTNTAT IMMQRGNFRN QRKMVKCFNC GKEGHTARNC RAPRKKGCWK CGKEGHQMKD CTERQANFLG KIWPSYKGRP |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 6 詳細: 50 mM sodium acetate (pH6.0), 100 microM ZnSO4 and 5 mM DTT |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: HOMEMADE PLUNGER |
| 詳細 | The sample forms a 3-dimensional lattice with ~8nm spacing. The sub-tomogram averages are aligned along the c6 symmetry axis, such that ~ 12 assymetric units are included for a total effective mass of ~ 0.45 MDa |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 77348 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 4.0 µm / 倍率(公称値): 59000 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称) / アルゴリズム: EXACT BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 18.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: matlab scripts / 使用したサブトモグラム数: 4000 |
|---|---|
| 抽出 | トモグラム数: 28 / 使用した粒子像数: 8400 / 参照モデル: EMD-2706 / 手法: Cross-correlation based template matching / ソフトウェア - 名称: matlab scripts |
| CTF補正 | ソフトウェア - 名称: matlab scripts 詳細: Phase flipping on the projections, amplitude correction applied to final average. |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)