+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8178 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Active human apoptosome with procaspase-9 | |||||||||
 マップデータ マップデータ | Active human apoptosome with procaspase-9 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Apaf-1 / apoptosis / programmed cell death / AAA+ ATPase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Release of apoptotic factors from the mitochondria / Formation of apoptosome / Activation of caspases through apoptosome-mediated cleavage / Pyroptosis / Regulation of the apoptosome activity / Transcriptional activation of mitochondrial biogenesis / caspase-9 / response to G1 DNA damage checkpoint signaling / caspase complex / regulation of apoptotic DNA fragmentation ...Release of apoptotic factors from the mitochondria / Formation of apoptosome / Activation of caspases through apoptosome-mediated cleavage / Pyroptosis / Regulation of the apoptosome activity / Transcriptional activation of mitochondrial biogenesis / caspase-9 / response to G1 DNA damage checkpoint signaling / caspase complex / regulation of apoptotic DNA fragmentation / Formation of apoptosome / Detoxification of Reactive Oxygen Species / apoptosome / TP53 Regulates Metabolic Genes / leukocyte apoptotic process / Cytoprotection by HMOX1 / glial cell apoptotic process / cysteine-type endopeptidase activator activity / response to cobalt ion / Respiratory electron transport / Caspase activation via Dependence Receptors in the absence of ligand / Activation of caspases through apoptosome-mediated cleavage / Regulation of the apoptosome activity / SMAC (DIABLO) binds to IAPs / SMAC(DIABLO)-mediated dissociation of IAP:caspase complexes / fibroblast apoptotic process / AKT phosphorylates targets in the cytosol / epithelial cell apoptotic process / mitochondrial electron transport, cytochrome c to oxygen / platelet formation / cysteine-type endopeptidase activator activity involved in apoptotic process / response to anesthetic / mitochondrial electron transport, ubiquinol to cytochrome c / TP53 Regulates Transcription of Caspase Activators and Caspases / Constitutive Signaling by AKT1 E17K in Cancer / forebrain development / Transcriptional Regulation by E2F6 / signal transduction in response to DNA damage / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / positive regulation of execution phase of apoptosis / cellular response to dexamethasone stimulus / cellular response to transforming growth factor beta stimulus / heat shock protein binding / cardiac muscle cell apoptotic process / response to nutrient / intrinsic apoptotic signaling pathway / protein maturation / response to ischemia / positive regulation of apoptotic signaling pathway / neural tube closure / kidney development / enzyme activator activity / NOD1/2 Signaling Pathway / ADP binding / protein processing / mitochondrial intermembrane space / SH3 domain binding / intrinsic apoptotic signaling pathway in response to DNA damage / cellular response to UV / response to estradiol / nervous system development / peptidase activity / positive regulation of neuron apoptotic process / neuron apoptotic process / secretory granule lumen / regulation of apoptotic process / response to lipopolysaccharide / ficolin-1-rich granule lumen / cell differentiation / response to hypoxia / electron transfer activity / positive regulation of apoptotic process / cysteine-type endopeptidase activity / nucleotide binding / apoptotic process / heme binding / DNA damage response / Neutrophil degranulation / protein kinase binding / protein-containing complex / mitochondrion / extracellular exosome / extracellular region / ATP binding / metal ion binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Cheng TC / Hong C / Akey IV / Yuan S / Akey CW | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2016 ジャーナル: Elife / 年: 2016タイトル: A near atomic structure of the active human apoptosome. 著者: Tat Cheung Cheng / Chuan Hong / Ildikó V Akey / Shujun Yuan / Christopher W Akey /  要旨: In response to cell death signals, an active apoptosome is assembled from Apaf-1 and procaspase-9 (pc-9). Here we report a near atomic structure of the active human apoptosome determined by cryo- ...In response to cell death signals, an active apoptosome is assembled from Apaf-1 and procaspase-9 (pc-9). Here we report a near atomic structure of the active human apoptosome determined by cryo-electron microscopy. The resulting model gives insights into cytochrome c binding, nucleotide exchange and conformational changes that drive assembly. During activation an acentric disk is formed on the central hub of the apoptosome. This disk contains four Apaf-1/pc-9 CARD pairs arranged in a shallow spiral with the fourth pc-9 CARD at lower occupancy. On average, Apaf-1 CARDs recruit 3 to 5 pc-9 molecules to the apoptosome and one catalytic domain may be parked on the hub, when an odd number of zymogens are bound. This suggests a stoichiometry of one or at most, two pc-9 dimers per active apoptosome. Thus, our structure provides a molecular framework to understand the role of the apoptosome in programmed cell death and disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8178.map.gz emd_8178.map.gz | 8.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8178-v30.xml emd-8178-v30.xml emd-8178.xml emd-8178.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8178.png emd_8178.png | 81.7 KB | ||
| Filedesc metadata |  emd-8178.cif.gz emd-8178.cif.gz | 7.5 KB | ||
| その他 |  emd_8178_additional.map.gz emd_8178_additional.map.gz | 6.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8178 http://ftp.pdbj.org/pub/emdb/structures/EMD-8178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8178 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8178_validation.pdf.gz emd_8178_validation.pdf.gz | 389 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8178_full_validation.pdf.gz emd_8178_full_validation.pdf.gz | 388.6 KB | 表示 | |
| XML形式データ |  emd_8178_validation.xml.gz emd_8178_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  emd_8178_validation.cif.gz emd_8178_validation.cif.gz | 7.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8178 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8178 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8178 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8178 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8178.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8178.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Active human apoptosome with procaspase-9 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.368 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Active human apoptosome with procaspase-9
| ファイル | emd_8178_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Active human apoptosome with procaspase-9 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
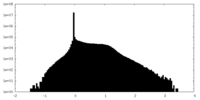
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Active complex of Apaf-1, cytochrome c and pro-caspase 9
| 全体 | 名称: Active complex of Apaf-1, cytochrome c and pro-caspase 9 |
|---|---|
| 要素 |
|
-超分子 #1: Active complex of Apaf-1, cytochrome c and pro-caspase 9
| 超分子 | 名称: Active complex of Apaf-1, cytochrome c and pro-caspase 9 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Complex stoichiometry: 4 full length Apaf-1 molecules with ordered N-terminal CARDs, 3 truncated Apaf-1 molecules without CARDs, 7 cytochrome c molecules and 4 N-terminal CARDs from procaspase-9 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.3 MDa |
-分子 #1: Apoptotic protease-activating factor 1
| 分子 | 名称: Apoptotic protease-activating factor 1 / タイプ: protein_or_peptide / ID: 1 / 詳細: without N-terminal CARDs / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 142.023672 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDAKARNCLL QHREALEKDI KTSYIMDHMI SDGFLTISEE EKVRNEPTQQ QRAAMLIKMI LKKDNDSYVS FYNALLHEGY KDLAALLHD GIPVVSSSSG KDSVSGITSY VRTVLCEGGV PQRPVVFVTR KKLVNAIQQK LSKLKGEPGW VTIHGMAGCG K SVLAAEAV ...文字列: MDAKARNCLL QHREALEKDI KTSYIMDHMI SDGFLTISEE EKVRNEPTQQ QRAAMLIKMI LKKDNDSYVS FYNALLHEGY KDLAALLHD GIPVVSSSSG KDSVSGITSY VRTVLCEGGV PQRPVVFVTR KKLVNAIQQK LSKLKGEPGW VTIHGMAGCG K SVLAAEAV RDHSLLEGCF PGGVHWVSVG KQDKSGLLMK LQNLCTRLDQ DESFSQRLPL NIEEAKDRLR ILMLRKHPRS LL ILDDVWD SWVLKAFDSQ CQILLTTRDK SVTDSVMGPK YVVPVESSLG KEKGLEILSL FVNMKKADLP EQAHSIIKEC KGS PLVVSL IGALLRDFPN RWEYYLKQLQ NKQFKRIRKS SSYDYEALDE AMSISVEMLR EDIKDYYTDL SILQKDVKVP TKVL CILWD METEEVEDIL QEFVNKSLLF CDRNGKSFRY YLHDLQVDFL TEKNCSQLQD LHKKIITQFQ RYHQPHTLSP DQEDC MYWY NFLAYHMASA KMHKELCALM FSLDWIKAKT ELVGPAHLIH EFVEYRHILD EKDCAVSENF QEFLSLNGHL LGRQPF PNI VQLGLCEPET SEVYQQAKLQ AKQEVDNGML YLEWINKKNI TNLSRLVVRP HTDAVYHACF SEDGQRIASC GADKTLQ VF KAETGEKLLE IKAHEDEVLC CAFSTDDRFI ATCSVDKKVK IWNSMTGELV HTYDEHSEQV NCCHFTNSSH HLLLATGS S DCFLKLWDLN QKECRNTMFG HTNSVNHCRF SPDDKLLASC SADGTLKLWD ATSANERKSI NVKQFFLNLE DPQEDMEVI VKCCSWSADG ARIMVAAKNK IFLFDIHTSG LLGEIHTGHH STIQYCDFSP QNHLAVVALS QYCVELWNTD SRSKVADCRG HLSWVHGVM FSPDGSSFLT SSDDQTIRLW ETKKVCKNSA VMLKQEVDVV FQENEVMVLA VDHIRRLQLI NGRTGQIDYL T EAQVSCCC LSPHLQYIAF GDENGAIEIL ELVNNRIFQS RFQHKKTVWH IQFTADEKTL ISSSDDAEIQ VWNWQLDKCI FL RGHQETV KDFRLLKNSR LLSWSFDGTV KVWNIITGNK EKDFVCHQGT VLSCDISHDA TKFSSTSADK TAKIWSFDLL LPL HELRGH NGCVRCSAFS VDSTLLATGD DNGEIRIWNV SNGELLHLCA PLSEEGAATH GGWVTDLCFS PDGKMLISAG GYIK WWNVV TGESSQTFYT NGTNLKKIHV SPDFKTYVTV DNLGILYILQ TLE UniProtKB: Apoptotic protease-activating factor 1 |
-分子 #2: Cytochrome c
| 分子 | 名称: Cytochrome c / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.595392 KDa |
| 配列 | 文字列: GDVEKGKKIF VQKCAQCHTV EKGGKHKTGP NLHGLFGRKT GQAPGFSYTD ANKNKGITWG EETLMEYLEN PKKYIPGTKM IFAGIKKKG EREDLIAYLK KATNE UniProtKB: Cytochrome c |
-分子 #3: Caspase-9
| 分子 | 名称: Caspase-9 / タイプ: protein_or_peptide / ID: 3 詳細: N-terminal caspase recognition domain (CARD) of procaspase-9 コピー数: 4 / 光学異性体: LEVO / EC番号: caspase-9 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.140723 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDEADRRLLR RCRLRLVEEL QVDQLWDALL SRELFRPHMI EDIQRAGSGS RRDQARQLII DLETRGSQAL PLFISCLEDT GQDMLASFL RTNRQA UniProtKB: Caspase-9 |
-分子 #4: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE
| 分子 | 名称: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 7 / 式: DTP |
|---|---|
| 分子量 | 理論値: 491.182 Da |
| Chemical component information |  ChemComp-DTP: |
-分子 #5: HEME C
| 分子 | 名称: HEME C / タイプ: ligand / ID: 5 / コピー数: 7 / 式: HEC |
|---|---|
| 分子量 | 理論値: 618.503 Da |
| Chemical component information |  ChemComp-HEC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.0 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Buffer A | |||||||||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF |
| 詳細 | data collected with a Cs aberration corrector and a GIF energy filter with a slit width of 20 eV. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - 画像ごとのフレーム数: 2-23 / 撮影したグリッド数: 1 / 実像数: 1991 / 平均露光時間: 0.3 sec. / 平均電子線量: 1.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial rigid body fitting with Chimera with a previously published starting model (PDB 3J2T), molecular dynamics flexible fitting with MDFF and real space density refinement with Phenix |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-5juy: |
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)