+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7810 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human nuclear exosome-MTR4 RNA complex - focused reconstruction on the nine-subunit core, MPP6, and EXOSC10 | |||||||||
 マップデータ マップデータ | Focused reconstruction of the human exosome core, Mpp6, and Rrp6 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
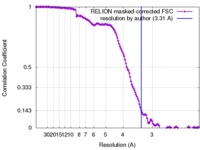
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.31 Å | |||||||||
 データ登録者 データ登録者 | Weick E-M / Lima CD | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2018 ジャーナル: Cell / 年: 2018タイトル: Helicase-Dependent RNA Decay Illuminated by a Cryo-EM Structure of a Human Nuclear RNA Exosome-MTR4 Complex. 著者: Eva-Maria Weick / M Rhyan Puno / Kurt Januszyk / John C Zinder / Michael A DiMattia / Christopher D Lima /  要旨: The ribonucleolytic RNA exosome interacts with RNA helicases to degrade RNA. To understand how the 3' to 5' Mtr4 helicase engages RNA and the nuclear exosome, we reconstituted 14-subunit Mtr4- ...The ribonucleolytic RNA exosome interacts with RNA helicases to degrade RNA. To understand how the 3' to 5' Mtr4 helicase engages RNA and the nuclear exosome, we reconstituted 14-subunit Mtr4-containing RNA exosomes from Saccharomyces cerevisiae, Schizosaccharomyces pombe, and human and show that they unwind structured substrates to promote degradation. We loaded a human exosome with an optimized DNA-RNA chimera that stalls MTR4 during unwinding and determined its structure to an overall resolution of 3.45 Å by cryoelectron microscopy (cryo-EM). The structure reveals an RNA-engaged helicase atop the non-catalytic core, with RNA captured within the central channel and DIS3 exoribonuclease active site. MPP6 tethers MTR4 to the exosome through contacts to the RecA domains of MTR4. EXOSC10 remains bound to the core, but its catalytic module and cofactor C1D are displaced by RNA-engaged MTR4. Competition for the exosome core may ensure that RNA is committed to degradation by DIS3 when engaged by MTR4. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7810.map.gz emd_7810.map.gz | 4.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7810-v30.xml emd-7810-v30.xml emd-7810.xml emd-7810.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7810_fsc.xml emd_7810_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7810.png emd_7810.png | 58.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7810 http://ftp.pdbj.org/pub/emdb/structures/EMD-7810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7810 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7810.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7810.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused reconstruction of the human exosome core, Mpp6, and Rrp6 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human nuclear exosome-MTR4 helicase captured after unwinding a DN...
| 全体 | 名称: Human nuclear exosome-MTR4 helicase captured after unwinding a DNA/RNA substrate |
|---|---|
| 要素 |
|
-超分子 #1: Human nuclear exosome-MTR4 helicase captured after unwinding a DN...
| 超分子 | 名称: Human nuclear exosome-MTR4 helicase captured after unwinding a DNA/RNA substrate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#15 詳細: Human C1D/Rrp47 also in the sample, but was not observed in density |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 690 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.037 kPa | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: 30 sec wait time, 2.5 sec blot time. | |||||||||||||||||||||
| 詳細 | Sample was monodisperse upon elution from gel filtration prior to vitrification. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 1439 / 平均電子線量: 85.23 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Models were rebuilt manually using Coot, with real space refinement with local scaling followed by Phenix real space refinement with suboptimal global scaling. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE / 温度因子: 47.4 / 当てはまり具合の基準: Correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー






















 Y (Sec.)
Y (Sec.) X (Row.)
X (Row.) Z (Col.)
Z (Col.)