+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7633 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of human enterovirus D68 full virion | |||||||||
 マップデータ マップデータ | sharpened map in which the inner density is masked out | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | virus / genome release / receptor | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / ribonucleoside triphosphate phosphatase activity / host cell cytoplasmic vesicle membrane / viral capsid / host cell / nucleoside-triphosphate phosphatase ...picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / ribonucleoside triphosphate phosphatase activity / host cell cytoplasmic vesicle membrane / viral capsid / host cell / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / host cell cytoplasm / RNA helicase activity / symbiont-mediated suppression of host innate immune response / endocytosis involved in viral entry into host cell / symbiont-mediated suppression of host gene expression / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / symbiont entry into host cell / virion attachment to host cell / host cell nucleus / structural molecule activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) | |||||||||
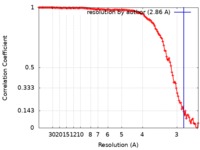
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.86 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Rossmann MG | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Bypassing pan-enterovirus host factor PLA2G16. 著者: Jim Baggen / Yue Liu / Heyrhyoung Lyoo / Arno L W van Vliet / Maryam Wahedi / Jost W de Bruin / Richard W Roberts / Pieter Overduin / Adam Meijer / Michael G Rossmann / Hendrik Jan Thibaut / ...著者: Jim Baggen / Yue Liu / Heyrhyoung Lyoo / Arno L W van Vliet / Maryam Wahedi / Jost W de Bruin / Richard W Roberts / Pieter Overduin / Adam Meijer / Michael G Rossmann / Hendrik Jan Thibaut / Frank J M van Kuppeveld /   要旨: Enteroviruses are a major cause of human disease. Adipose-specific phospholipase A2 (PLA2G16) was recently identified as a pan-enterovirus host factor and potential drug target. In this study, we ...Enteroviruses are a major cause of human disease. Adipose-specific phospholipase A2 (PLA2G16) was recently identified as a pan-enterovirus host factor and potential drug target. In this study, we identify a possible mechanism of PLA2G16 evasion by employing a dual glycan receptor-binding enterovirus D68 (EV-D68) strain. We previously showed that this strain does not strictly require the canonical EV-D68 receptor sialic acid. Here, we employ a haploid screen to identify sulfated glycosaminoglycans (sGAGs) as its second glycan receptor. Remarkably, engagement of sGAGs enables this virus to bypass PLA2G16. Using cryo-EM analysis, we reveal that, in contrast to sialic acid, sGAGs stimulate genome release from virions via structural changes that enlarge the putative openings for genome egress. Together, we describe an enterovirus that can bypass PLA2G16 and identify additional virion destabilization as a potential mechanism to circumvent PLA2G16. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7633.map.gz emd_7633.map.gz | 156.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7633-v30.xml emd-7633-v30.xml emd-7633.xml emd-7633.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7633_fsc.xml emd_7633_fsc.xml | 14.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7633.png emd_7633.png | 271.3 KB | ||
| Filedesc metadata |  emd-7633.cif.gz emd-7633.cif.gz | 6.4 KB | ||
| その他 |  emd_7633_half_map_1.map.gz emd_7633_half_map_1.map.gz emd_7633_half_map_2.map.gz emd_7633_half_map_2.map.gz | 57.5 MB 57.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7633 http://ftp.pdbj.org/pub/emdb/structures/EMD-7633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7633 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7633_validation.pdf.gz emd_7633_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7633_full_validation.pdf.gz emd_7633_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_7633_validation.xml.gz emd_7633_validation.xml.gz | 20.7 KB | 表示 | |
| CIF形式データ |  emd_7633_validation.cif.gz emd_7633_validation.cif.gz | 27.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7633 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7633 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6cv2MC  7632C  7634C  7635C  7636C  7638C  6cv1C  6cv3C  6cv4C  6cv5C  6cvbC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7633.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7633.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map in which the inner density is masked out | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
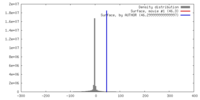
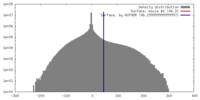
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: unsharpened half map in which the inner density is retained
| ファイル | emd_7633_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened half map in which the inner density is retained | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
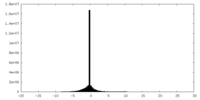
| 密度ヒストグラム |
-ハーフマップ: unsharpened half map in which the inner density is retained
| ファイル | emd_7633_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened half map in which the inner density is retained | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
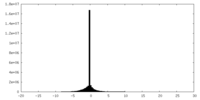
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Enterovirus D68
| 全体 | 名称:  Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Enterovirus D68
| 超分子 | 名称: Enterovirus D68 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Grown in RD cells / NCBI-ID: 42789 / 生物種: Enterovirus D68 / Sci species strain: 947 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: viral protein 1
| 分子 | 名称: viral protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterovirus D68 (エンテロウイルス) / 株: 947 Enterovirus D68 (エンテロウイルス) / 株: 947 |
| 分子量 | 理論値: 32.957449 KDa |
| 配列 | 文字列: IESIIKTATD TVKSEINAEL GVVPSLNAVE TGATSNTEPE EAIQTRTVIN QHGVSETLVE NFLSRAALVS KRSFEYKDHT SSAAQTDKN FFKWTINTRS FVQLRRKLEL FTYLRFDAEI TILTTVAVNG SSNNTYMGLP NLTLQAMFVP TGALTPEKQD S FHWQSGSN ...文字列: IESIIKTATD TVKSEINAEL GVVPSLNAVE TGATSNTEPE EAIQTRTVIN QHGVSETLVE NFLSRAALVS KRSFEYKDHT SSAAQTDKN FFKWTINTRS FVQLRRKLEL FTYLRFDAEI TILTTVAVNG SSNNTYMGLP NLTLQAMFVP TGALTPEKQD S FHWQSGSN ASVFFKISDP PARMTIPFMC INSAYSVFYD GFAGFEKSGL YGINPADTIG NLCVRIVNEH QPVGFTVTVR VY MKPKHIK AWAPRPPRTL PYMSIANANY KGKKRAPNAL NAIIGNRDSV KTMPHNIVTT UniProtKB: Genome polyprotein |
-分子 #2: viral protein 3
| 分子 | 名称: viral protein 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterovirus D68 (エンテロウイルス) / 株: 947 Enterovirus D68 (エンテロウイルス) / 株: 947 |
| 分子量 | 理論値: 27.170895 KDa |
| 配列 | 文字列: GVPTYLLPGS GQFLTTDDHS SAPVLPCFNP TPEMHIPGQV RNMLEVVQVE SMMEINNTES AVGMERLKVD ISALTDVDQL LFNIPLDIQ LDGPLRNTLV GNISRYYTHW SGSLEMTFMF CGSFMATGKL ILCYTPPGGS CPTTRETAML GTHIVWDFGL Q SSVTLIIP ...文字列: GVPTYLLPGS GQFLTTDDHS SAPVLPCFNP TPEMHIPGQV RNMLEVVQVE SMMEINNTES AVGMERLKVD ISALTDVDQL LFNIPLDIQ LDGPLRNTLV GNISRYYTHW SGSLEMTFMF CGSFMATGKL ILCYTPPGGS CPTTRETAML GTHIVWDFGL Q SSVTLIIP WISGSHYRMF NNDAKSTNAN VGYVTCFMQT NLIVPSESSD TCSLIGFIAA KDDFSLRLMR DSPDIGQIDH LH AAEAAYQ UniProtKB: Genome polyprotein |
-分子 #3: viral protein 2
| 分子 | 名称: viral protein 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterovirus D68 (エンテロウイルス) / 株: 947 Enterovirus D68 (エンテロウイルス) / 株: 947 |
| 分子量 | 理論値: 27.595215 KDa |
| 配列 | 文字列: SPSAEACGYS DRVLQLKLGN SAIVTQEAAN YCCAYGEWPN YLPDHEAVAI DKPTQPETAT DRFYTLKSVK WEAGSTGWWW KLPDALNNI GMFGQNVQHH YLYRSGFLIH VQCNATRFHQ GALLVVAIPE HQRGAHNTNT SPGFDDIMKG EEGGTFNHPY V LDDGTSLA ...文字列: SPSAEACGYS DRVLQLKLGN SAIVTQEAAN YCCAYGEWPN YLPDHEAVAI DKPTQPETAT DRFYTLKSVK WEAGSTGWWW KLPDALNNI GMFGQNVQHH YLYRSGFLIH VQCNATRFHQ GALLVVAIPE HQRGAHNTNT SPGFDDIMKG EEGGTFNHPY V LDDGTSLA CATIFPHQWI NLRTNNSATI VLPWMNAAPM DFPLRHNQWT LAIIPVVPLG TRTMSSMVPI TVSIAPMCCE FN GLRHAIT Q UniProtKB: Genome polyprotein |
-分子 #4: viral protein 4
| 分子 | 名称: viral protein 4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterovirus D68 (エンテロウイルス) / 株: 947 Enterovirus D68 (エンテロウイルス) / 株: 947 |
| 分子量 | 理論値: 7.33696 KDa |
| 配列 | 文字列: GAQVTRQQTG THENANIATN GSHITYNQIN FYKDSYAASA SKQDFSQDPS KFTEPVVEGL KAGAPVLK UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 / 詳細: phosphate buffer |
|---|---|
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 気圧: 0.02 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 2-28 / 撮影したグリッド数: 1 / 実像数: 218 / 平均露光時間: 7.0 sec. / 平均電子線量: 33.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 3.1 µm / 最小 デフォーカス(補正後): 0.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6cv2: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)