+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7105 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV-1 Envelope Glycoprotein clone PC64M7c022 SOSIP.664 | |||||||||
 マップデータ マップデータ | primary map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
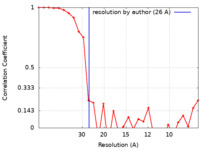
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Berndsen ZT / Rantalainen K / Ward AB | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2017 ジャーナル: Immunity / 年: 2017タイトル: HIV Envelope Glycoform Heterogeneity and Localized Diversity Govern the Initiation and Maturation of a V2 Apex Broadly Neutralizing Antibody Lineage. 著者: Elise Landais / Ben Murrell / Bryan Briney / Sasha Murrell / Kimmo Rantalainen / Zachary T Berndsen / Alejandra Ramos / Lalinda Wickramasinghe / Melissa Laird Smith / Kemal Eren / Natalia de ...著者: Elise Landais / Ben Murrell / Bryan Briney / Sasha Murrell / Kimmo Rantalainen / Zachary T Berndsen / Alejandra Ramos / Lalinda Wickramasinghe / Melissa Laird Smith / Kemal Eren / Natalia de Val / Mengyu Wu / Audrey Cappelletti / Jeffrey Umotoy / Yolanda Lie / Terri Wrin / Paul Algate / Po-Ying Chan-Hui / Etienne Karita / / / Andrew B Ward / Ian A Wilson / Dennis R Burton / Davey Smith / Sergei L Kosakovsky Pond / Pascal Poignard /    要旨: Understanding how broadly neutralizing antibodies (bnAbs) to HIV envelope (Env) develop during natural infection can help guide the rational design of an HIV vaccine. Here, we described a bnAb ...Understanding how broadly neutralizing antibodies (bnAbs) to HIV envelope (Env) develop during natural infection can help guide the rational design of an HIV vaccine. Here, we described a bnAb lineage targeting the Env V2 apex and the Ab-Env co-evolution that led to development of neutralization breadth. The lineage Abs bore an anionic heavy chain complementarity-determining region 3 (CDRH3) of 25 amino acids, among the shortest known for this class of Abs, and achieved breadth with only 10% nucleotide somatic hypermutation and no insertions or deletions. The data suggested a role for Env glycoform heterogeneity in the activation of the lineage germline B cell. Finally, we showed that localized diversity at key V2 epitope residues drove bnAb maturation toward breadth, mirroring the Env evolution pattern described for another donor who developed V2-apex targeting bnAbs. Overall, these findings suggest potential strategies for vaccine approaches based on germline-targeting and serial immunogen design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7105.map.gz emd_7105.map.gz | 734.6 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7105-v30.xml emd-7105-v30.xml emd-7105.xml emd-7105.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7105_fsc.xml emd_7105_fsc.xml | 2.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7105.png emd_7105.png | 53.6 KB | ||
| マスクデータ |  emd_7105_msk_1.map emd_7105_msk_1.map | 1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_7105_half_map_1.map.gz emd_7105_half_map_1.map.gz emd_7105_half_map_2.map.gz emd_7105_half_map_2.map.gz | 740.9 KB 739.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7105 http://ftp.pdbj.org/pub/emdb/structures/EMD-7105 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7105 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7105 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7105_validation.pdf.gz emd_7105_validation.pdf.gz | 78.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7105_full_validation.pdf.gz emd_7105_full_validation.pdf.gz | 77.2 KB | 表示 | |
| XML形式データ |  emd_7105_validation.xml.gz emd_7105_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7105 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7105 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7105 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7105 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7105.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7105.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_7105_msk_1.map emd_7105_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_7105_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_7105_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 Envelope Glycoprotein clone PC64M7c022 SOSIP.664
| 全体 | 名称: HIV-1 Envelope Glycoprotein clone PC64M7c022 SOSIP.664 |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 Envelope Glycoprotein clone PC64M7c022 SOSIP.664
| 超分子 | 名称: HIV-1 Envelope Glycoprotein clone PC64M7c022 SOSIP.664 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 225 KDa |
-分子 #1: HIV-1 Envelope Glycoprotein PC64M7c022 SOSIP.664
| 分子 | 名称: HIV-1 Envelope Glycoprotein PC64M7c022 SOSIP.664 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGAR ANNL WVTVYYGVPV WRDAETTLFC ASDAKAYDTE VHNVWATHAC VPTDPSPQEI HLANVTEKFD MWKNSMVEQM HTDIISLWDE SLKPCVKLTP LCITLNCTNI TKKNVTGGNL TEDGKEELKN CSFNATTELS ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGAR ANNL WVTVYYGVPV WRDAETTLFC ASDAKAYDTE VHNVWATHAC VPTDPSPQEI HLANVTEKFD MWKNSMVEQM HTDIISLWDE SLKPCVKLTP LCITLNCTNI TKKNVTGGNL TEDGKEELKN CSFNATTELS NKRQKVHSLF YRLDLVELNE GNSSNSNTSM YRLINCNTSA ITQACPKVSF EPIPIHYCAP AGFAILKCRE KEFNGTGPCK NVSTVQCTHG IKPVVSTQLL LNGSLAEGTV KIRCENISNN AKTILVQLTT PVRINCTRPN NNTRTSIRIG PGQSFYATGD IIGDIRKAYC NVSGSEWKEA LGKVVVQLRN HFNKTITFAS SSGGDLEITT HSFNCGGEFF YCNTSSLFNS TWDRNSTTNS TQEPNGTITL PCRIKQIINM WQRTGQAMYA PPIPGKIRCD SNITGLILTR DGENNNTESE TFRPEGGDMR NNWRSELYKY KVVKIDPLGV APTGCKRRVV ERRRRRRAVG IGAVLFGFLG AAGSTMGAAS LTLTVQARQL LSGIVQQQSN LLRAPEAQQH LLRLTVWGIK QLQARVLAVE RYLSDQQLLG IWGCSGKLIC CTTVPWNSSW SNKSQDEIWN NMTWLQWDKE ISNYTDTIYY LIEKSQNQQE VNEKDLLALD KWTNLWNWFD ISKWLWYIKI FIMIVGGLIG LRIIFAVLSV INRVRQGYSP VSFQTLTPNP RELDRPGGIE EGDGELGKTR SIRLVGGFLA LFWDDLRSLC LFSYHRLRDF ILIAARILEL LGHNSLKGLR LGWEGLKYLG NLLLYWGREL KNSAVNLVDT IAIVVAGWTD RVIEVLQGIG RAFLHVPRRI RQGLERALL |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: NanoW |
| グリッド | 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 詳細: unspecified |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 平均露光時間: 0.5 sec. / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)