+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6905 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human Dicer and its complexes with a pre-miRNA substrate | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Dicer / TRBP / Cryo-EM / RNA interference / PROTEIN BINDING / HYDROLASE-PROTEIN BINDING-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of siRNA processing / regulation of miRNA processing / negative regulation of cytoplasmic pattern recognition receptor signaling pathway / regulation of viral transcription / peripheral nervous system myelin formation / regulation of regulatory ncRNA processing / negative regulation of defense response to virus by host / global gene silencing by mRNA cleavage / pre-miRNA binding / negative regulation of Schwann cell proliferation ...regulation of siRNA processing / regulation of miRNA processing / negative regulation of cytoplasmic pattern recognition receptor signaling pathway / regulation of viral transcription / peripheral nervous system myelin formation / regulation of regulatory ncRNA processing / negative regulation of defense response to virus by host / global gene silencing by mRNA cleavage / pre-miRNA binding / negative regulation of Schwann cell proliferation / tRNA-derived small RNA (tsRNA or tRNA-related fragment, tRF) biogenesis / Small interfering RNA (siRNA) biogenesis / apoptotic DNA fragmentation / tRNA decay / ribonuclease III / deoxyribonuclease I activity / nerve development / positive regulation of Schwann cell differentiation / RISC-loading complex / miRNA metabolic process / positive regulation of myelination / RISC complex assembly / miRNA processing / ribonuclease III activity / skeletal muscle tissue regeneration / pre-miRNA processing / siRNA processing / siRNA binding / Regulation of MITF-M-dependent genes involved in apoptosis / M-decay: degradation of maternal mRNAs by maternally stored factors / pre-mRNA binding / RISC complex / neural precursor cell proliferation / miRNA binding / MicroRNA (miRNA) biogenesis / positive regulation of muscle cell differentiation / negative regulation of tumor necrosis factor production / spermatid development / negative regulation of tumor necrosis factor-mediated signaling pathway / single fertilization / positive regulation of viral genome replication / negative regulation of protein kinase activity / neuron projection morphogenesis / RNA endonuclease activity / protein sequestering activity / positive regulation of translation / helicase activity / PKR-mediated signaling / multicellular organism growth / double-stranded RNA binding / nuclear body / protein domain specific binding / negative regulation of gene expression / perinuclear region of cytoplasm / enzyme binding / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / RNA binding / extracellular exosome / nucleoplasm / ATP binding / metal ion binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Liu Z / Wang J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2018 ジャーナル: Cell / 年: 2018タイトル: Cryo-EM Structure of Human Dicer and Its Complexes with a Pre-miRNA Substrate. 著者: Zhongmin Liu / Jia Wang / Hang Cheng / Xin Ke / Lei Sun / Qiangfeng Cliff Zhang / Hong-Wei Wang /  要旨: Human Dicer (hDicer) is a multi-domain protein belonging to the RNase III family. It plays pivotal roles in small RNA biogenesis during the RNA interference (RNAi) pathway by processing a diverse ...Human Dicer (hDicer) is a multi-domain protein belonging to the RNase III family. It plays pivotal roles in small RNA biogenesis during the RNA interference (RNAi) pathway by processing a diverse range of double-stranded RNA (dsRNA) precursors to generate ∼22 nt microRNA (miRNA) or small interfering RNA (siRNA) products for sequence-directed gene silencing. In this work, we solved the cryoelectron microscopy (cryo-EM) structure of hDicer in complex with its cofactor protein TRBP and revealed the precise spatial arrangement of hDicer's multiple domains. We further solved structures of the hDicer-TRBP complex bound with pre-let-7 RNA in two distinct conformations. In combination with biochemical analysis, these structures reveal a property of the hDicer-TRBP complex to promote the stability of pre-miRNA's stem duplex in a pre-dicing state. These results provide insights into the mechanism of RNA processing by hDicer and illustrate the regulatory role of hDicer's N-terminal helicase domain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
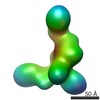
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6905.map.gz emd_6905.map.gz | 2.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6905-v30.xml emd-6905-v30.xml emd-6905.xml emd-6905.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6905.png emd_6905.png | 63.1 KB | ||
| Filedesc metadata |  emd-6905.cif.gz emd-6905.cif.gz | 7.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6905 http://ftp.pdbj.org/pub/emdb/structures/EMD-6905 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6905 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6905 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6905_validation.pdf.gz emd_6905_validation.pdf.gz | 366.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6905_full_validation.pdf.gz emd_6905_full_validation.pdf.gz | 366 KB | 表示 | |
| XML形式データ |  emd_6905_validation.xml.gz emd_6905_validation.xml.gz | 5.6 KB | 表示 | |
| CIF形式データ |  emd_6905_validation.cif.gz emd_6905_validation.cif.gz | 6.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6905 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6905 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6905 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6905 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6905.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6905.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.37 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
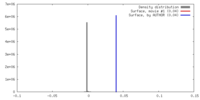
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Dicer and trbp
| 全体 | 名称: Dicer and trbp |
|---|---|
| 要素 |
|
-超分子 #1: Dicer and trbp
| 超分子 | 名称: Dicer and trbp / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 270 KDa |
-超分子 #2: Human Dicer-TRBP with Pre-let-7 complex Class I
| 超分子 | 名称: Human Dicer-TRBP with Pre-let-7 complex Class I / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Endoribonuclease Dicer
| 分子 | 名称: Endoribonuclease Dicer / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: ribonuclease III |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 218.947328 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKSPALQPLS MAGLQLMTPA SSPMGPFFGL PWQQEAIHDN IYTPRKYQVE LLEAALDHNT IVCLNTGSGK TFIAVLLTKE LSYQIRGDF SRNGKRTVFL VNSANQVAQQ VSAVRTHSDL KVGEYSNLEV NASWTKERWN QEFTKHQVLI MTCYVALNVL K NGYLSLSD ...文字列: MKSPALQPLS MAGLQLMTPA SSPMGPFFGL PWQQEAIHDN IYTPRKYQVE LLEAALDHNT IVCLNTGSGK TFIAVLLTKE LSYQIRGDF SRNGKRTVFL VNSANQVAQQ VSAVRTHSDL KVGEYSNLEV NASWTKERWN QEFTKHQVLI MTCYVALNVL K NGYLSLSD INLLVFDECH LAILDHPYRE IMKLCENCPS CPRILGLTAS ILNGKCDPEE LEEKIQKLEK ILKSNAETAT DL VVLDRYT SQPCEIVVDC GPFTDRSGLY ERLLMELEEA LNFINDCNIS VHSKERDSTL ISKQILSDCR AVLVVLGPWC ADK VAGMMV RELQKYIKHE QEELHRKFLL FTDTFLRKIH ALCEEHFSPA SLDLKFVTPK VIKLLEILRK YKPYERQQFE SVEW YNNRN QDNYVSWSDS EDDDEDEEIE EKEKPETNFP SPFTNILCGI IFVERRYTAV VLNRLIKEAG KQDPELAYIS SNFIT GHGI GKNQPRNKQM EAEFRKQEEV LRKFRAHETN LLIATSIVEE GVDIPKCNLV VRFDLPTEYR SYVQSKGRAR APISNY IML ADTDKIKSFE EDLKTYKAIE KILRNKCSKS VDTGETDIDP VMDDDDVFPP YVLRPDDGGP RVTINTAIGH INRYCAR LP SDPFTHLAPK CRTRELPDGT FYSTLYLPIN SPLRASIVGP PMSCVRLAER VVALICCEKL HKIGELDDHL MPVGKETV K YEEELDLHDE EETSVPGRPG STKRRQCYPK AIPECLRDSY PRPDQPCYLY VIGMVLTTPL PDELNFRRRK LYPPEDTTR CFGILTAKPI PQIPHFPVYT RSGEVTISIE LKKSGFMLSL QMLELITRLH QYIFSHILRL EKPALEFKPT DADSAYCVLP LNVVNDSST LDIDFKFMED IEKSEARIGI PSTKYTKETP FVFKLEDYQD AVIIPRYRNF DQPHRFYVAD VYTDLTPLSK F PSPEYETF AEYYKTKYNL DLTNLNQPLL DVDHTSSRLN LLTPRHLNQK GKALPLSSAE KRKAKWESLQ NKQILVPELC AI HPIPASL WRKAVCLPSI LYRLHCLLTA EELRAQTASD AGVGVRSLPA DFRYPNLDFG WKKSIDSKSF ISISNSSSAE NDN YCKHST IVPENAAHQG ANRTSSLENH DQMSVNCRTL LSESPGKLHV EVSADLTAIN GLSYNQNLAN GSYDLANRDF CQGN QLNYY KQEIPVQPTT SYSIQNLYSY ENQPQPSDEC TLLSNKYLDG NANKSTSDGS PVMAVMPGTT DTIQVLKGRM DSEQS PSIG YSSRTLGPNP GLILQALTLS NASDGFNLER LEMLGDSFLK HAITTYLFCT YPDAHEGRLS YMRSKKVSNC NLYRLG KKK GLPSRMVVSI FDPPVNWLPP GYVVNQDKSN TDKWEKDEMT KDCMLANGKL DEDYEEEDEE EESLMWRAPK EEADYED DF LEYDQEHIRF IDNMLMGSGA FVKKISLSPF STTDSAYEWK MPKKSSLGSM PFSSDFEDFD YSSWDAMCYL DPSKAVEE D DFVVGFWNPS EENCGVDTGK QSISYDLHTE QCIADKSIAD CVEALLGCYL TSCGERAAQL FLCSLGLKVL PVIKRTDRE KALCPTRENF NSQQKNLSVS CAAASVASSR SSVLKDSEYG CLKIPPRCMF DHPDADKTLN HLISGFENFE KKINYRFKNK AYLLQAFTH ASYHYNTITD CYQRLEFLGD AILDYLITKH LYEDPRQHSP GVLTDLRSAL VNNTIFASLA VKYDYHKYFK A VSPELFHV IDDFVQFQLE KNEMQGMDSE LRRSEEDEEK EEDIEVPKAM GDIFESLAGA IYMDSGMSLE TVWQVYYPMM RP LIEKFSA NVPRSPVREL LEMEPETAKF SPAERTYDGK VRVTVEVVGK GKFKGVGRSY RIAKSAAARR ALRSLKANQP QVP NS UniProtKB: Endoribonuclease Dicer |
-分子 #2: RISC-loading complex subunit TARBP2
| 分子 | 名称: RISC-loading complex subunit TARBP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 39.085277 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSEEEQGSGT TTGCGLPSIE QMLAANPGKT PISLLQEYGT RIGKTPVYDL LKAEGQAHQP NFTFRVTVGD TSCTGQGPSK KAAKHKAAE VALKHLKGGS MLEPALEDSS SFSPLDSSLP EDIPVFTAAA AATPVPSVVL TRSPPMELQP PVSPQQSECN P VGALQELV ...文字列: MSEEEQGSGT TTGCGLPSIE QMLAANPGKT PISLLQEYGT RIGKTPVYDL LKAEGQAHQP NFTFRVTVGD TSCTGQGPSK KAAKHKAAE VALKHLKGGS MLEPALEDSS SFSPLDSSLP EDIPVFTAAA AATPVPSVVL TRSPPMELQP PVSPQQSECN P VGALQELV VQKGWRLPEY TVTQESGPAH RKEFTMTCRV ERFIEIGSGT SKKLAKRNAA AKMLLRVHTV PLDARDGNEV EP DDDHFSI GVGSRLDGLR NRGPGCTWDS LRNSVGEKIL SLRSCSLGSL GALGPACCRV LSELSEEQAF HVSYLDIEEL SLS GLCQCL VELSTQPATV CHGSATTREA ARGEAARRAL QYLKIMAGSK UniProtKB: RISC-loading complex subunit TARBP2 |
-分子 #3: RNA (73-mer)
| 分子 | 名称: RNA (73-mer) / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.37583 KDa |
| 配列 | 文字列: UGAGGUAGUA GGUUGUAUAG UUUUAGGGUC ACACCCACCA CUGGGAGAUA ACUAUACAAU CUACUGUCUU ACC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 50 mM Tris-HCl (pH 8.0), 100 mM NaCl, 1 mM DTT, 2 mM CaCl2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 詳細: The grid was glow-discharged prior to use. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | This sample was mono-disperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| 特殊光学系 | 位相板: OTHER |
| 詳細 | no |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-32 / 撮影したグリッド数: 1 / 実像数: 2849 / 平均露光時間: 5.44 sec. / 平均電子線量: 50.0 e/Å2 / 詳細: no |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.5 µm / 最小 デフォーカス(補正後): 2.5 µm / 倍率(補正後): 36496 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 89000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 1-1922 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 100 / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-5zal: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)