+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6295 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
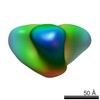
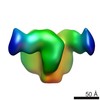
| タイトル | JRFL gp140 NFL2P liganded to PGV04 | |||||||||
 マップデータ マップデータ | JRFL gp140 NFL2P liganded to PGV04 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 22.0 Å | |||||||||
 データ登録者 データ登録者 | de Val N / Sharma S / Bale S | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2015 ジャーナル: Cell Rep / 年: 2015タイトル: Cleavage-independent HIV-1 Env trimers engineered as soluble native spike mimetics for vaccine design. 著者: Shailendra Kumar Sharma / Natalia de Val / Shridhar Bale / Javier Guenaga / Karen Tran / Yu Feng / Viktoriya Dubrovskaya / Andrew B Ward / Richard T Wyatt /  要旨: Viral glycoproteins mediate entry by pH-activated or receptor-engaged activation and exist in metastable pre-fusogenic states that may be stabilized by directed rational design. As recently reported, ...Viral glycoproteins mediate entry by pH-activated or receptor-engaged activation and exist in metastable pre-fusogenic states that may be stabilized by directed rational design. As recently reported, the conformationally fixed HIV-1 envelope glycoprotein (Env) trimers in the pre-fusion state (SOSIP) display molecular homogeneity and structural integrity at relatively high levels of resolution. However, the SOSIPs necessitate full Env precursor cleavage, which requires endogenous furin overexpression. Here, we developed an alternative strategy using flexible peptide covalent linkage of Env subdomains to produce soluble, homogeneous, and cleavage-independent Env mimics, called native flexibly linked (NFL) trimers, as vaccine candidates. This simplified design avoids the need for furin co-expression and, in one case, antibody affinity purification to accelerate trimer scale-up for preclinical and clinical applications. We have successfully translated the NFL design to multiple HIV-1 subtypes, establishing the potential to become a general method of producing native-like, well-ordered Env trimers for HIV-1 or other viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6295.map.gz emd_6295.map.gz | 11.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6295-v30.xml emd-6295-v30.xml emd-6295.xml emd-6295.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6295.gif 400_6295.gif 80_6295.gif 80_6295.gif | 13.3 KB 1.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6295 http://ftp.pdbj.org/pub/emdb/structures/EMD-6295 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6295 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6295 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6295_validation.pdf.gz emd_6295_validation.pdf.gz | 77.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6295_full_validation.pdf.gz emd_6295_full_validation.pdf.gz | 76.6 KB | 表示 | |
| XML形式データ |  emd_6295_validation.xml.gz emd_6295_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6295 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6295 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6295 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6295 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6295.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6295.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | JRFL gp140 NFL2P liganded to PGV04 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : JRFL gp140 NFL2P liganded to PGV04
| 全体 | 名称: JRFL gp140 NFL2P liganded to PGV04 |
|---|---|
| 要素 |
|
-超分子 #1000: JRFL gp140 NFL2P liganded to PGV04
| 超分子 | 名称: JRFL gp140 NFL2P liganded to PGV04 / タイプ: sample / ID: 1000 / 集合状態: trimer / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 570 KDa / 理論値: 570 KDa / 手法: Size exclusion chromatography (SEC) |
-分子 #1: JRFL gp140 NFL2P
| 分子 | 名称: JRFL gp140 NFL2P / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 集合状態: trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)別称: HIV-1 |
| 分子量 | 実験値: 570 KDa / 理論値: 570 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK 293T Homo sapiens (ヒト) / 組換細胞: HEK 293T |
-分子 #2: PGV04 Fab
| 分子 | 名称: PGV04 Fab / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: human Homo sapiens (ヒト) / 別称: human |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50 mM Tris-HCl, 150 mM NaCl |
| 染色 | タイプ: NEGATIVE / 詳細: 2% uranyl formate for 30 seconds |
| グリッド | 詳細: 400 mesh Cu grids, negatively glow discharged for 30 seconds |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 日付 | 2014年2月21日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 178 / 平均電子線量: 36.01 e/Å2 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 52000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.1 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 46000 |
| 試料ステージ | 試料ホルダーモデル: HOME BUILD / Tilt angle max: 50 |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 22.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN, sparx / 使用した粒子像数: 27335 |
|---|
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















