+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | portal-tail of Vibrio cholerae typing phage release VP1 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | phage / virus / vibrio cholera phage / VIRAL PROTEIN | ||||||||||||
| 生物種 |   | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.7 Å | ||||||||||||
 データ登録者 データ登録者 | Liu HR / Pang H | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: to be published ジャーナル: to be publishedタイトル: Three-dimensional structures of Vibrio cholerae typing podophage VP1 in two states 著者: Liu HR / Pang H | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_60199.map.gz emd_60199.map.gz | 226 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-60199-v30.xml emd-60199-v30.xml emd-60199.xml emd-60199.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_60199.png emd_60199.png | 65.9 KB | ||
| Filedesc metadata |  emd-60199.cif.gz emd-60199.cif.gz | 6 KB | ||
| その他 |  emd_60199_half_map_1.map.gz emd_60199_half_map_1.map.gz emd_60199_half_map_2.map.gz emd_60199_half_map_2.map.gz | 226 MB 226 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-60199 http://ftp.pdbj.org/pub/emdb/structures/EMD-60199 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60199 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60199 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_60199_validation.pdf.gz emd_60199_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_60199_full_validation.pdf.gz emd_60199_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_60199_validation.xml.gz emd_60199_validation.xml.gz | 15.6 KB | 表示 | |
| CIF形式データ |  emd_60199_validation.cif.gz emd_60199_validation.cif.gz | 18.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60199 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60199 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60199 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60199 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_60199.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_60199.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
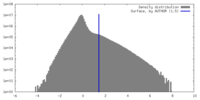
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_60199_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
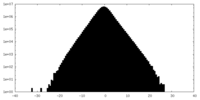
| 投影像・断面図 |
| ||||||||||||
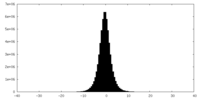
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_60199_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
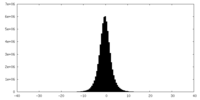
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vibrio cholerae phage
| 全体 | 名称:  |
|---|---|
| 要素 |
|
-超分子 #1: Vibrio cholerae phage
| 超分子 | 名称: Vibrio cholerae phage / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 666 / 生物種: Vibrio cholerae phage / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  |
-分子 #1: ring protein of release VP1
| 分子 | 名称: ring protein of release VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.311119 KDa |
| 配列 | 文字列: MSSWDRFDTP WDSIIDESTW EHYTFKYDAS FEAFSSMEVD EDLTITVSVL FASTSDNTVT VGQVLGLTMD NRAGSYFGAG SSWTSEAAV NNEAGSGFQS SHALQLSYSV EDGVISSFGS EAFCSFYNTV SFESSSDVQA AVNSLYNLDV LFSSGSGDSE Q HYVVFGET ...文字列: MSSWDRFDTP WDSIIDESTW EHYTFKYDAS FEAFSSMEVD EDLTITVSVL FASTSDNTVT VGQVLGLTMD NRAGSYFGAG SSWTSEAAV NNEAGSGFQS SHALQLSYSV EDGVISSFGS EAFCSFYNTV SFESSSDVQA AVNSLYNLDV LFSSGSGDSE Q HYVVFGET ASFESLAEHE TSSQYITHVE CMFNSVVEFE VKTYDWGRPV KPVGSDWSTD TPVVGVWVNE IYNGNKDWGE S |
-分子 #2: adaptor of release VP1
| 分子 | 名称: adaptor of release VP1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.59285 KDa |
| 配列 | 文字列: MDKATLVKTI AYRMGNVKGQ DTAIDFELAL SIERLEGQEF VPWFLLSENN FFEGTAQENR IPVPRGFIRE YEEGSLYLRR VAGTGKCLI KKSQDQLLKY EGMTGEPSHY SLTNQYFRIY PVPQEDFKVE LLFYRKSSTL NVEDNPWYEY AAELLVAETI W AMLSARRD ...文字列: MDKATLVKTI AYRMGNVKGQ DTAIDFELAL SIERLEGQEF VPWFLLSENN FFEGTAQENR IPVPRGFIRE YEEGSLYLRR VAGTGKCLI KKSQDQLLKY EGMTGEPSHY SLTNQYFRIY PVPQEDFKVE LLFYRKSSTL NVEDNPWYEY AAELLVAETI W AMLSARRD KMADYWKSVA ADQMRRLTIL DAERRLANQE IFMG |
-分子 #3: nozzle of release VP1
| 分子 | 名称: nozzle of release VP1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 58.925699 KDa |
| 配列 | 文字列: MEVTHKQFDL SGFLPDQAPD TLKSGAISRG FNVKPTILGW EKSGGFRETN TAPTNEKDFI FFWSPAIGDN RWFSGGDKTV QQVEGNVVS DVSRTGGYTA AAAARWNAVN FNGVLLMNNE LDSPQYLAAS GKLEDFPNLP SNVRFRTVAV YKNFILGLGV N FGSGFLDD ...文字列: MEVTHKQFDL SGFLPDQAPD TLKSGAISRG FNVKPTILGW EKSGGFRETN TAPTNEKDFI FFWSPAIGDN RWFSGGDKTV QQVEGNVVS DVSRTGGYTA AAAARWNAVN FNGVLLMNNE LDSPQYLAAS GKLEDFPNLP SNVRFRTVAV YKNFILGLGV N FGSGFLDD EIYWSHQADP GTMPPNWDYA NAASDSGRTP LPSEGYCVTS EELGSMNIVY KSDSIWTMQL IGGQWIFRFE NK FPGQGIL NKKSVVSFEG KHFVVTQKDI IVHDGYQVRS VADKRVRNFF FTDMNSDYFE RVFVVKDPRV AEVYVFYPSK NSV DGLCDR ALVWNWRDDV WSLLNLRPLK HAAYGYEITG VSITWDNFVG GWESTGLWQA DEDVAKYAPV LHYSFRDVPK LLAP TPQAL FIDEEIEAVW EREDIVIGSI SRDGVPYQDY ERNKSVSSIS FDVDTTEPFD VYIGYKGSLE DSVEWEFAGT VNPME DKRL FCLLTAGLFS MRIISKAQTF ILRSYKITYE FAGEMWA |
-分子 #4: portal of release VP1
| 分子 | 名称: portal of release VP1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 66.026672 KDa |
| 配列 | 文字列: MEILYTGASE SLHSTILKAL LERIDLGDTF ISDNYTRWQA TERYYMMYKI PNKKDKAAIE KWNKGDTDFK SLVMPYSYAQ LMTAHAYMV NVFLNRDPIF QTDSLNGDGT ERELALESML QYQVKAGEME PSLLVWFMDA LRYGVGVLGD YWEEHVFHQT V FEVKKTRV ...文字列: MEILYTGASE SLHSTILKAL LERIDLGDTF ISDNYTRWQA TERYYMMYKI PNKKDKAAIE KWNKGDTDFK SLVMPYSYAQ LMTAHAYMV NVFLNRDPIF QTDSLNGDGT ERELALESML QYQVKAGEME PSLLVWFMDA LRYGVGVLGD YWEEHVFHQT V FEVKKTRV VKGYEGCKTF NVMVYDFIPD PRVALCKYQE GEFFGRRLDL NVLDLKKGAK FGKYFNVEHA EALVAASKEE MY RRDPSIG QQRSLKDSTM TPKGKQVGDI SCVEIFVRLV PKDWGLGDSE FPEMWVFTVA DKKYIVAAEP VNTLDDKFPF HIL ECEIDG YMNKSRGLLE ISAPMNDILT WLFDSHMYNK RQIMNNQFIG DPSALVVKDV ESKEPGKFIR LRPIISQLPV TDVT AQNIQ DVQVVERNMQ RIVGVNDDVA GQSSPSSRRS ATEFRGTTSF ASSRLANLAY FFSVTGFRSL AKSLIVKTQQ LYTVE MKVK VAGDNIKGAQ SIIVKPEDIS GQFDIMPVDG TLPVDRMAQA QFWMQIMSMV AGNPVLGAEY RLGDIFSYTA RLAGLK GID KMKIRILDDD QILALILA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 32.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)