+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5447 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Molecular Architecture of the Unliganded Membrane-Bound HIV-1 Envelope Glycoprotein Trimer | |||||||||
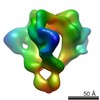
 マップデータ マップデータ | Reconstruction of the unliganded HIV-1 envelope glycoprotein trimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV / envelope spike / trimer / membrane glycoprotein | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.0 Å | |||||||||
 データ登録者 データ登録者 | Mao Y / Wang L / Sodroski J | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2013 ジャーナル: Proc Natl Acad Sci U S A / 年: 2013タイトル: Molecular architecture of the uncleaved HIV-1 envelope glycoprotein trimer. 著者: Youdong Mao / Liping Wang / Christopher Gu / Alon Herschhorn / Anik Désormeaux / Andrés Finzi / Shi-Hua Xiang / Joseph G Sodroski /  要旨: The human immunodeficiency virus type 1 (HIV-1) envelope glycoprotein (Env) trimer, a membrane-fusing machine, mediates virus entry into host cells and is the sole virus-specific target for ...The human immunodeficiency virus type 1 (HIV-1) envelope glycoprotein (Env) trimer, a membrane-fusing machine, mediates virus entry into host cells and is the sole virus-specific target for neutralizing antibodies. Binding the receptors, CD4 and CCR5/CXCR4, triggers Env conformational changes from the metastable unliganded state to the fusion-active state. We used cryo-electron microscopy to obtain a 6-Å structure of the membrane-bound, heavily glycosylated HIV-1 Env trimer in its uncleaved and unliganded state. The spatial organization of secondary structure elements reveals that the unliganded conformations of both glycoprotein (gp)120 and gp41 subunits differ from those induced by receptor binding. The gp120 trimer association domains, which contribute to interprotomer contacts in the unliganded Env trimer, undergo rearrangement upon CD4 binding. In the unliganded Env, intersubunit interactions maintain the gp41 ectodomain helical bundles in a "spring-loaded" conformation distinct from the extended helical coils of the fusion-active state. Quaternary structure regulates the virus-neutralizing potency of antibodies targeting the conserved CD4-binding site on gp120. The Env trimer architecture provides mechanistic insights into the metastability of the unliganded state, receptor-induced conformational changes, and quaternary structure-based strategies for immune evasion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5447.map.gz emd_5447.map.gz | 59.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5447-v30.xml emd-5447-v30.xml emd-5447.xml emd-5447.xml | 11.5 KB 11.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5447_1.tif emd_5447_1.tif | 248.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5447 http://ftp.pdbj.org/pub/emdb/structures/EMD-5447 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5447 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5447 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5447_validation.pdf.gz emd_5447_validation.pdf.gz | 78.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5447_full_validation.pdf.gz emd_5447_full_validation.pdf.gz | 77.8 KB | 表示 | |
| XML形式データ |  emd_5447_validation.xml.gz emd_5447_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5447 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5447 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5447 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5447 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10003 (タイトル: HIV-1 envelope glycoprotein trimer micrographs EMPIAR-10003 (タイトル: HIV-1 envelope glycoprotein trimer micrographsData size: 586.8 Data #1: 10 focal pair micrographs of HIV-1 Envelope Glycoprotein Trimer [picked particles - single frame - unprocessed] Data #2: Full set of 4683 focal pair micrographs [micrographs - focal pairs - contrast inverted])  EMPIAR-10007 (タイトル: Low-contrast particle stack for HIV-1 Env gp160 precursor (Spider stack) EMPIAR-10007 (タイトル: Low-contrast particle stack for HIV-1 Env gp160 precursor (Spider stack)Data size: 164.2 Data #1: U115 raw particle images [picked particles - multiframe - unprocessed])  EMPIAR-10008 (タイトル: Low-contrast particle stack for HIV-1 Env gp160 precursor (MRC stack) EMPIAR-10008 (タイトル: Low-contrast particle stack for HIV-1 Env gp160 precursor (MRC stack)Data size: 240.2 / Data #1: U115 [picked particles - multiframe - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5447.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5447.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the unliganded HIV-1 envelope glycoprotein trimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.74688 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 envelope glycoprotein trimer
| 全体 | 名称: HIV-1 envelope glycoprotein trimer |
|---|---|
| 要素 |
|
-超分子 #1000: HIV-1 envelope glycoprotein trimer
| 超分子 | 名称: HIV-1 envelope glycoprotein trimer / タイプ: sample / ID: 1000 詳細: The sample was monodisperse and solubilized in Cymal-6. 集合状態: three gp120 exterior subunits and three gp41 transmembrane subunits Number unique components: 3 |
|---|---|
| 分子量 | 実験値: 440 KDa / 理論値: 435 KDa / 手法: Sedimentation, gel electrophoresis |
-分子 #1: HIV-1 envelope glycoprotein trimer precursor
| 分子 | 名称: HIV-1 envelope glycoprotein trimer precursor / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Env trimer, gp160 trimer 詳細: The glycoprotein was expressed in 293F cells and was purified from the extracted plasma membranes. The cDNA sequence is derived from HIV-1 JR-FL isolate. 集合状態: trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)別称: HIV-1 |
| 分子量 | 実験値: 440 KDa / 理論値: 435 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換プラスミド: pcDNA3.1 in 293F cell line Homo sapiens (ヒト) / 組換プラスミド: pcDNA3.1 in 293F cell line |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris-HCl, 300 mM NaCl and 0.01% Cymal-6 |
| グリッド | 詳細: 200 mesh and 400 mesh C-flat grid with thin holey carbon support |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 93 K / 装置: FEI VITROBOT MARK IV 手法: Held for 2 s, blotted by filter papers for 2 s before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 89 K / 最高: 93 K / 平均: 91 K |
| 日付 | 2009年9月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) 実像数: 5991 / 平均電子線量: 10 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 200835 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 150000 |
| 試料ステージ | 試料ホルダー: CT3500 / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Wiener filter |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 6.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER 詳細: The final map was deconvoluted and amplitude-corrected with a B-factor of 250 and was low-pass filtered at 5.6 Angstrom with a cosine edge of 8-Fourier-pixel width. The final resolution of ...詳細: The final map was deconvoluted and amplitude-corrected with a B-factor of 250 and was low-pass filtered at 5.6 Angstrom with a cosine edge of 8-Fourier-pixel width. The final resolution of the refined cryo-EM map, measured by FSC-0.5 cutoff, is 6 Angstrom without masking of the background noise in the map and is 5.66 Angstrom with of masking the background noise. 使用した粒子像数: 582914 |
| 最終 角度割当 | 詳細: 0.5 degree of angular step |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: G |
|---|---|
| ソフトウェア | 名称: O, Coot, CNS, Chimera, Modeller |
| 詳細 | Protocol: Flexible fitting |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)