+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5307 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional cryo-electron microscopy density map of the 70S ribosome from Mycobacterium smegmatis | |||||||||
 マップデータ マップデータ | Reconstruction of 70S ribosome from Mycobacterium smegmatis | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cryoEM / prokaryotic / ribosome / 3D-structure | |||||||||
| 生物種 |  Mycobacterium smegmatis (バクテリア) Mycobacterium smegmatis (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Shasmal M / Sengupta J | |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2012 ジャーナル: PLoS One / 年: 2012タイトル: Structural diversity in bacterial ribosomes: mycobacterial 70S ribosome structure reveals novel features. 著者: Manidip Shasmal / Jayati Sengupta /  要旨: Here we present analysis of a 3D cryo-EM map of the 70S ribosome from Mycobacterium smegmatis, a saprophytic cousin of the etiological agent of tuberculosis in humans, Mycobacterium tuberculosis. In ...Here we present analysis of a 3D cryo-EM map of the 70S ribosome from Mycobacterium smegmatis, a saprophytic cousin of the etiological agent of tuberculosis in humans, Mycobacterium tuberculosis. In comparison with the 3D structures of other prokaryotic ribosomes, the density map of the M. smegmatis 70S ribosome reveals unique structural features and their relative orientations in the ribosome. Dramatic changes in the periphery due to additional rRNA segments and extra domains of some of the peripheral ribosomal proteins like S3, S5, S16, L17, L25, are evident. One of the most notable features appears in the large subunit near L1 stalk as a long helical structure next to helix 54 of the 23S rRNA. The sharp upper end of this structure is located in the vicinity of the mRNA exit channel. Although the M. smegmatis 70S ribosome possesses conserved core structure of bacterial ribosome, the new structural features, unveiled in this study, demonstrates diversity in the 3D architecture of bacterial ribosomes. We postulate that the prominent helical structure related to the 23S rRNA actively participates in the mechanisms of translation in mycobacteria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5307.map.gz emd_5307.map.gz | 34.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5307-v30.xml emd-5307-v30.xml emd-5307.xml emd-5307.xml | 9.3 KB 9.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5307_1.tif emd_5307_1.tif | 207.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5307 http://ftp.pdbj.org/pub/emdb/structures/EMD-5307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5307 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5307_validation.pdf.gz emd_5307_validation.pdf.gz | 78 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5307_full_validation.pdf.gz emd_5307_full_validation.pdf.gz | 77.1 KB | 表示 | |
| XML形式データ |  emd_5307_validation.xml.gz emd_5307_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5307 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5307 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5307 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5307 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5307.map.gz / 形式: CCP4 / 大きさ: 37 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5307.map.gz / 形式: CCP4 / 大きさ: 37 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of 70S ribosome from Mycobacterium smegmatis | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.69 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
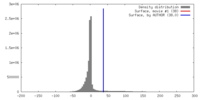
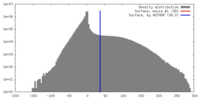
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 70S ribosome from Mycobacterium smegmatis
| 全体 | 名称: 70S ribosome from Mycobacterium smegmatis |
|---|---|
| 要素 |
|
-超分子 #1000: 70S ribosome from Mycobacterium smegmatis
| 超分子 | 名称: 70S ribosome from Mycobacterium smegmatis / タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 2.5 MDa |
-超分子 #1: 70S ribosome with P-site tRNA
| 超分子 | 名称: 70S ribosome with P-site tRNA / タイプ: complex / ID: 1 / Name.synonym: 70S ribosome / 組換発現: No / データベース: NCBI / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium smegmatis (バクテリア) Mycobacterium smegmatis (バクテリア) |
| 分子量 | 理論値: 2.5 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-HCl, pH 7.5, 10 mM magnesium acetate, 30 mM ammonium chloride, 5 mM 2-mercaptoethanol |
|---|---|
| グリッド | 詳細: Holey grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 94 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: Cryo plunger. Rapid-freezing in liquid ethane |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 日付 | 2010年11月2日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC CCD / デジタル化 - サンプリング間隔: 15 µm / 実像数: 269 / 平均電子線量: 15 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 88466 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 89000 |
| 試料ステージ | 試料ホルダー: Cryo transfer / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: CTF correction of 3D map |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 12.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER / 使用した粒子像数: 1 |
-原子モデル構築 1
| 初期モデル | PDB ID:  2i2u |
|---|---|
| ソフトウェア | 名称: PyMol |
| 詳細 | Manual fitting |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID:  2i2v |
|---|---|
| ソフトウェア | 名称: PyMol |
| 詳細 | Manual fitting |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)