+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mammalian quaternary complex of a translating 80S ribosome, NAC, MetAP1 and NatA/E-HYPK - local refinement | ||||||||||||||||||
 マップデータ マップデータ | map of the quaternary RNC-NAC-MetAP1-NatAE-HYPK complex refined with a mask on NatA/E-NAC-MetAP1 | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | translation / ribosome / NAC / N-terminal acetyltransferase / NatA / NatE / MetAP1 / HYPK | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.75 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Yudin D / Scaiola A / Ban N | ||||||||||||||||||
| 資金援助 |  米国, 米国,  ドイツ, ドイツ,  スイス, European Union, 5件 スイス, European Union, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: NAC guides a ribosomal multienzyme complex for nascent protein processing. 著者: Alfred M Lentzsch / Denis Yudin / Martin Gamerdinger / Sowmya Chandrasekar / Laurenz Rabl / Alain Scaiola / Elke Deuerling / Nenad Ban / Shu-Ou Shan /    要旨: Approximately 40% of the mammalian proteome undergoes N-terminal methionine excision and acetylation, mediated sequentially by methionine aminopeptidase (MetAP) and N-acetyltransferase A (NatA), ...Approximately 40% of the mammalian proteome undergoes N-terminal methionine excision and acetylation, mediated sequentially by methionine aminopeptidase (MetAP) and N-acetyltransferase A (NatA), respectively. Both modifications are strictly cotranslational and essential in higher eukaryotic organisms. The interaction, activity and regulation of these enzymes on translating ribosomes are poorly understood. Here we perform biochemical, structural and in vivo studies to demonstrate that the nascent polypeptide-associated complex (NAC) orchestrates the action of these enzymes. NAC assembles a multienzyme complex with MetAP1 and NatA early during translation and pre-positions the active sites of both enzymes for timely sequential processing of the nascent protein. NAC further releases the inhibitory interactions from the NatA regulatory protein huntingtin yeast two-hybrid protein K (HYPK) to activate NatA on the ribosome, enforcing cotranslational N-terminal acetylation. Our results provide a mechanistic model for the cotranslational processing of proteins in eukaryotic cells. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50129.map.gz emd_50129.map.gz | 331.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50129-v30.xml emd-50129-v30.xml emd-50129.xml emd-50129.xml | 44.7 KB 44.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
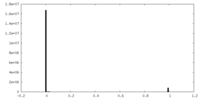
| FSC (解像度算出) |  emd_50129_fsc.xml emd_50129_fsc.xml | 18.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_50129.png emd_50129.png | 92.2 KB | ||
| マスクデータ |  emd_50129_msk_1.map emd_50129_msk_1.map emd_50129_msk_2.map emd_50129_msk_2.map | 669.9 MB 669.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-50129.cif.gz emd-50129.cif.gz | 4.9 KB | ||
| その他 |  emd_50129_additional_1.map.gz emd_50129_additional_1.map.gz emd_50129_additional_2.map.gz emd_50129_additional_2.map.gz emd_50129_half_map_1.map.gz emd_50129_half_map_1.map.gz emd_50129_half_map_2.map.gz emd_50129_half_map_2.map.gz | 627.6 MB 322.1 MB 620.8 MB 620.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50129 http://ftp.pdbj.org/pub/emdb/structures/EMD-50129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50129 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50129.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50129.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map of the quaternary RNC-NAC-MetAP1-NatAE-HYPK complex refined with a mask on NatA/E-NAC-MetAP1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
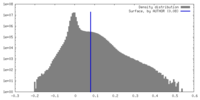
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_50129_msk_1.map emd_50129_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
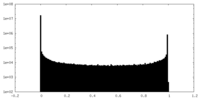
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_50129_msk_2.map emd_50129_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
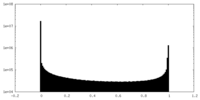
| 密度ヒストグラム |
-追加マップ: main map lowpass filtered to 8 A resolution
| ファイル | emd_50129_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map lowpass filtered to 8 A resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: resampled map used during the refinement of the...
| ファイル | emd_50129_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | resampled map used during the refinement of the quaternary complex model - see the methods section of the manuscript for details | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfmap A
| ファイル | emd_50129_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfmap B
| ファイル | emd_50129_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Quaternary complex of a translating 80S ribosome, NAC, MetAP1 and...
| 全体 | 名称: Quaternary complex of a translating 80S ribosome, NAC, MetAP1 and NatA/E-HYPK |
|---|---|
| 要素 |
|
-超分子 #1: Quaternary complex of a translating 80S ribosome, NAC, MetAP1 and...
| 超分子 | 名称: Quaternary complex of a translating 80S ribosome, NAC, MetAP1 and NatA/E-HYPK タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#91 |
|---|---|
| 分子量 | 理論値: 3.50 MDa |
-超分子 #2: Rabbit ribosome-nascent chain complex
| 超分子 | 名称: Rabbit ribosome-nascent chain complex / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#6, #14-#91 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Human NAC heterodimer
| 超分子 | 名称: Human NAC heterodimer / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #8-#9 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #4: Human NatA/E-HYPK complex
| 超分子 | 名称: Human NatA/E-HYPK complex / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #10-#13 |
|---|
-超分子 #5: Human NatA complex
| 超分子 | 名称: Human NatA complex / タイプ: complex / ID: 5 / 親要素: 4 / 含まれる分子: #11-#12 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #6: Human HYPK
| 超分子 | 名称: Human HYPK / タイプ: complex / ID: 6 / 親要素: 4 / 含まれる分子: #13 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #7: Human NatE with GST tag
| 超分子 | 名称: Human NatE with GST tag / タイプ: complex / ID: 7 / 親要素: 4 / 含まれる分子: #10 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)