+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of ancestral nitrogenase MoFe-protein | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Metalloprotein / OXIDOREDUCTASE | |||||||||
| 生物種 |  Azotobacter vinelandii (窒素固定) Azotobacter vinelandii (窒素固定) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.19 Å | |||||||||
 データ登録者 データ登録者 | Shen Y / Warmack RA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2025 ジャーナル: Structure / 年: 2025タイトル: CryoEM-enabled visual proteomics reveals de novo structures of oligomeric protein complexes. 著者: Yuanbo Shen / Ailiena O Maggiolo / Tianzheng Zhang / Rebeccah A Warmack /  要旨: Single particle cryoelectron microscopy (cryoEM) and cryoelectron tomography (cryoET) are powerful methods for unveiling unique and functionally relevant structural states. Aided by mass spectrometry ...Single particle cryoelectron microscopy (cryoEM) and cryoelectron tomography (cryoET) are powerful methods for unveiling unique and functionally relevant structural states. Aided by mass spectrometry and machine learning, they promise to facilitate the visual exploration of proteomes. Leveraging visual proteomics, we interrogate structures isolated from a complex cellular milieu by cryoEM to identify and classify molecular structures and complexes de novo. By comparing three automated model building programs, CryoID, DeepTracer, and ModelAngelo, we determine the identity of six distinct oligomeric protein complexes from partially purified extracts of the nitrogen-fixing bacterium Azotobacter vinelandii using both anaerobic and aerobic cryoEM, including two original oligomeric structures. Overall, by allowing the study of near-native oligomeric protein states, cryoEM-enabled visual proteomics reveals unique structures that correspond to relevant species observed in situ. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_49753.map.gz emd_49753.map.gz | 684.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-49753-v30.xml emd-49753-v30.xml emd-49753.xml emd-49753.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_49753_fsc.xml emd_49753_fsc.xml | 19 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_49753.png emd_49753.png | 104.3 KB | ||
| Filedesc metadata |  emd-49753.cif.gz emd-49753.cif.gz | 6.3 KB | ||
| その他 |  emd_49753_half_map_1.map.gz emd_49753_half_map_1.map.gz emd_49753_half_map_2.map.gz emd_49753_half_map_2.map.gz | 677.1 MB 677.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-49753 http://ftp.pdbj.org/pub/emdb/structures/EMD-49753 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-49753 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-49753 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_49753_validation.pdf.gz emd_49753_validation.pdf.gz | 985 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_49753_full_validation.pdf.gz emd_49753_full_validation.pdf.gz | 984.6 KB | 表示 | |
| XML形式データ |  emd_49753_validation.xml.gz emd_49753_validation.xml.gz | 28.3 KB | 表示 | |
| CIF形式データ |  emd_49753_validation.cif.gz emd_49753_validation.cif.gz | 37.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-49753 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-49753 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-49753 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-49753 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_49753.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_49753.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_49753_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_49753_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nitrogenase MoFe-protein
| 全体 | 名称: Nitrogenase MoFe-protein |
|---|---|
| 要素 |
|
-超分子 #1: Nitrogenase MoFe-protein
| 超分子 | 名称: Nitrogenase MoFe-protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii (窒素固定) / 株: DJ Azotobacter vinelandii (窒素固定) / 株: DJ |
-分子 #1: Nitrogenase MoFe-protein alpha chain
| 分子 | 名称: Nitrogenase MoFe-protein alpha chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii (窒素固定) / 株: DJ Azotobacter vinelandii (窒素固定) / 株: DJ |
| 分子量 | 理論値: 55.73318 KDa |
| 配列 | 文字列: MTGMSRDEVE SLIQEVLEVY PEKAKKNREK HLSPNDPELE QSKKCITSNK KSLPGVMTIR GCAYAGSKGV VWGPIKDMIH ISHGPVGCG QYSRAGRRNY YIGTTGVNAF VTMNFTSDFQ EKDIVFGGDK KLAKLIDEIE TLFPLNKGIS VQSECPIGLI G DDIEAVSK ...文字列: MTGMSRDEVE SLIQEVLEVY PEKAKKNREK HLSPNDPELE QSKKCITSNK KSLPGVMTIR GCAYAGSKGV VWGPIKDMIH ISHGPVGCG QYSRAGRRNY YIGTTGVNAF VTMNFTSDFQ EKDIVFGGDK KLAKLIDEIE TLFPLNKGIS VQSECPIGLI G DDIEAVSK QKGKEHGKTI VPVRCEGFRG VSQSLGHHIA NDAVRDWVLS ARDDDDSFET TDYDVAIIGD YNIGGDAWSS RI LLEEMGL RVVAQWSGDG TISEMELTPK VKLNLVHCYR SMNYISRHME EKYGIPWMEY NFFGPTKTIE SLRKIAAQFD ESI QKKCEE VIAKYQPEWE AVVAKYRPRL EGKRVMLYVG GLRPRHVIGA YEDLGMEVVG TGYEFGHNDD YDRTLKEMGD STLL YDDVT GYEFEEFVKK VKPDLIGSGI KEKYIFQKMG IPFRQMHSWD YSGPYHGFDG FAIFARDMDM TLNNPCWKKL QAPWK KSEQ ADEAVAASA |
-分子 #2: Nitrogenase MoFe-protein beta chain
| 分子 | 名称: Nitrogenase MoFe-protein beta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii (窒素固定) / 株: DJ Azotobacter vinelandii (窒素固定) / 株: DJ |
| 分子量 | 理論値: 60.071207 KDa |
| 配列 | 文字列: MSQQVDNIKP SYPLFRDQDY KDMLAKKRDN FEEKHPQEKI DEVFQWTTTE EYQELNFQRE ALTVNPAKAC QPLGAVLCSL GFEKTMPYV HGSQGCVAYF RTYFNRHFKE PISCVSDSMT EDAAVFGGQQ NMKDGLQNCK AIYKPDMIAV STTCMAEVIG D DLNAFINN ...文字列: MSQQVDNIKP SYPLFRDQDY KDMLAKKRDN FEEKHPQEKI DEVFQWTTTE EYQELNFQRE ALTVNPAKAC QPLGAVLCSL GFEKTMPYV HGSQGCVAYF RTYFNRHFKE PISCVSDSMT EDAAVFGGQQ NMKDGLQNCK AIYKPDMIAV STTCMAEVIG D DLNAFINN SKKEGHIPDE FPVPFAHTPS FVGSHVTGWD NMFEGIARYF TLNYMEDKEV GSNGKINIVP GFETYLGNFR VI KRMMNEM NVDYTLLSDP EEVLDTPADG QFRMYAGGTT QDEMKDAPNA LNTLMLQPWQ LTKTTKFVKN TWKHEVPKLN IPM GLDWTD EFLMKVSEIS GQPIPESLAK ERGRLVDMMT DSHTWLHGKK FALWGDPDFV MGMTKFLLEL GCEPIHILCN NANK RWKKA MDAILAESPY GANSEVHIGK DLWHMRSLVF TNKPDFMIGN SYGKFIQRDT LYKGKEFEVP LIRIGFPIFD RHHLH RQTT LGYEGAMQIL TTLVNSVLER LDEETRGMQT TDYNYDLVR |
-分子 #3: iron-sulfur-molybdenum cluster with interstitial carbon
| 分子 | 名称: iron-sulfur-molybdenum cluster with interstitial carbon タイプ: ligand / ID: 3 / コピー数: 2 / 式: ICS |
|---|---|
| 分子量 | 理論値: 787.451 Da |
| Chemical component information | 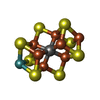 ChemComp-ICE: |
-分子 #4: 3-HYDROXY-3-CARBOXY-ADIPIC ACID
| 分子 | 名称: 3-HYDROXY-3-CARBOXY-ADIPIC ACID / タイプ: ligand / ID: 4 / コピー数: 2 / 式: HCA |
|---|---|
| 分子量 | 理論値: 206.15 Da |
| Chemical component information | 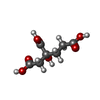 ChemComp-HCA: |
-分子 #5: FE(8)-S(7) CLUSTER
| 分子 | 名称: FE(8)-S(7) CLUSTER / タイプ: ligand / ID: 5 / コピー数: 2 / 式: CLF |
|---|---|
| 分子量 | 理論値: 671.215 Da |
| Chemical component information | 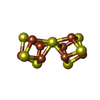 ChemComp-CLF: |
-分子 #6: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 1394 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





































