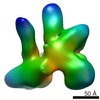
登録情報 データベース : EMDB / ID : EMD-4739タイトル Structural basis of Cullin-2 RING E3 ligase regulation by the COP9 signalosome 複合体 : Ternary complex of COP9 signalosome (CSN) and neddylated Cullin-Ring E3 ligase CRL (cullin-2 with Rbx1, Elongin-B, Elongin-C and VHL) / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 8.2 Å Faull SV / Lau AMC 資金援助 Organization Grant number 国 Cancer Research UK
ジャーナル : Nat Commun / 年 : 2019タイトル : Structural basis of Cullin 2 RING E3 ligase regulation by the COP9 signalosome.著者 : Sarah V Faull / Andy M C Lau / Chloe Martens / Zainab Ahdash / Kjetil Hansen / Hugo Yebenes / Carla Schmidt / Fabienne Beuron / Nora B Cronin / Edward P Morris / Argyris Politis / 要旨 : Cullin-Ring E3 Ligases (CRLs) regulate a multitude of cellular pathways through specific substrate receptors. The COP9 signalosome (CSN) deactivates CRLs by removing NEDD8 from activated Cullins. ... Cullin-Ring E3 Ligases (CRLs) regulate a multitude of cellular pathways through specific substrate receptors. The COP9 signalosome (CSN) deactivates CRLs by removing NEDD8 from activated Cullins. Here we present structures of the neddylated and deneddylated CSN-CRL2 complexes by combining single-particle cryo-electron microscopy (cryo-EM) with chemical cross-linking mass spectrometry (XL-MS). These structures suggest a conserved mechanism of CSN activation, consisting of conformational clamping of the CRL2 substrate by CSN2/CSN4, release of the catalytic CSN5/CSN6 heterodimer and finally activation of the CSN5 deneddylation machinery. Using hydrogen-deuterium exchange (HDX)-MS we show that CRL2 activates CSN5/CSN6 in a neddylation-independent manner. The presence of NEDD8 is required to activate the CSN5 active site. Overall, by synergising cryo-EM with MS, we identify sensory regions of the CSN that mediate its stepwise activation and provide a framework for understanding the regulatory mechanism of other Cullin family members. 履歴 登録 2019年3月28日 - ヘッダ(付随情報) 公開 2019年8月28日 - マップ公開 2019年8月28日 - 更新 2024年5月22日 - 現状 2024年5月22日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2019
ジャーナル: Nat Commun / 年: 2019


 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_4739.map.gz
emd_4739.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-4739-v30.xml
emd-4739-v30.xml emd-4739.xml
emd-4739.xml EMDBヘッダ
EMDBヘッダ emd_4739.png
emd_4739.png emd-4739.cif.gz
emd-4739.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-4739
http://ftp.pdbj.org/pub/emdb/structures/EMD-4739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4739
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4739 emd_4739_validation.pdf.gz
emd_4739_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_4739_full_validation.pdf.gz
emd_4739_full_validation.pdf.gz emd_4739_validation.xml.gz
emd_4739_validation.xml.gz emd_4739_validation.cif.gz
emd_4739_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4739
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4739 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4739
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4739 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_4739.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_4739.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー






































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















