+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4635 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of ovine transhydrogenase in the presence of NADP+ in a "double face-down" conformation | |||||||||
 マップデータ マップデータ | Combined map from focused refinement of two domains separately: (dII plus dIII) and (dI). | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | mitochondrial / proton-translocating / nicotinamide nucleotide transhydrogenase / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proton-translocating NAD(P)+ transhydrogenase activity / proton-translocating NAD(P)+ transhydrogenase / NADPH regeneration / NADP binding / oxidoreductase activity / mitochondrial inner membrane / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
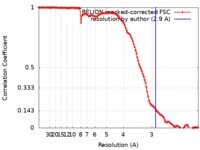
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Kampjut D / Sazanov LA | |||||||||
| 資金援助 |  オーストリア, 1件 オーストリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Structure and mechanism of mitochondrial proton-translocating transhydrogenase. 著者: Domen Kampjut / Leonid A Sazanov /  要旨: Proton-translocating transhydrogenase (also known as nicotinamide nucleotide transhydrogenase (NNT)) is found in the plasma membranes of bacteria and the inner mitochondrial membranes of eukaryotes. ...Proton-translocating transhydrogenase (also known as nicotinamide nucleotide transhydrogenase (NNT)) is found in the plasma membranes of bacteria and the inner mitochondrial membranes of eukaryotes. NNT catalyses the transfer of a hydride between NADH and NADP, coupled to the translocation of one proton across the membrane. Its main physiological function is the generation of NADPH, which is a substrate in anabolic reactions and a regulator of oxidative status; however, NNT may also fine-tune the Krebs cycle. NNT deficiency causes familial glucocorticoid deficiency in humans and metabolic abnormalities in mice, similar to those observed in type II diabetes. The catalytic mechanism of NNT has been proposed to involve a rotation of around 180° of the entire NADP(H)-binding domain that alternately participates in hydride transfer and proton-channel gating. However, owing to the lack of high-resolution structures of intact NNT, the details of this process remain unclear. Here we present the cryo-electron microscopy structure of intact mammalian NNT in different conformational states. We show how the NADP(H)-binding domain opens the proton channel to the opposite sides of the membrane, and we provide structures of these two states. We also describe the catalytically important interfaces and linkers between the membrane and the soluble domains and their roles in nucleotide exchange. These structures enable us to propose a revised mechanism for a coupling process in NNT that is consistent with a large body of previous biochemical work. Our results are relevant to the development of currently unavailable NNT inhibitors, which may have therapeutic potential in ischaemia reperfusion injury, metabolic syndrome and some cancers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4635.map.gz emd_4635.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4635-v30.xml emd-4635-v30.xml emd-4635.xml emd-4635.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4635_fsc.xml emd_4635_fsc.xml | 12.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4635.png emd_4635.png | 68.6 KB | ||
| Filedesc metadata |  emd-4635.cif.gz emd-4635.cif.gz | 6.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4635 http://ftp.pdbj.org/pub/emdb/structures/EMD-4635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4635 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4635.map.gz / 形式: CCP4 / 大きさ: 6.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4635.map.gz / 形式: CCP4 / 大きさ: 6.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Combined map from focused refinement of two domains separately: (dII plus dIII) and (dI). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Nicotinamide nucleotide transhydrogenase in the presence of NADP+
| 全体 | 名称: Nicotinamide nucleotide transhydrogenase in the presence of NADP+ |
|---|---|
| 要素 |
|
-超分子 #1: Nicotinamide nucleotide transhydrogenase in the presence of NADP+
| 超分子 | 名称: Nicotinamide nucleotide transhydrogenase in the presence of NADP+ タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 220 KDa |
-分子 #1: Nicotinamide nucleotide transhydrogenase
| 分子 | 名称: Nicotinamide nucleotide transhydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 114.010305 KDa |
| 配列 | 文字列: MANLLKTVVT GCSCPFLSNL GSCKVLPGKK NFLRTFHTHR ILWCKAPVKP GIPYKQLTVG VPKEIFQNEK RVALSPAGVQ ALVKQGFNV VVESGAGEAS KFSDDHYRAA GAQIQGAKEV LASDLVVKVR APMLNPTLGI HEADLLKTSG TLISFIYPAQ N PDLLNKLS ...文字列: MANLLKTVVT GCSCPFLSNL GSCKVLPGKK NFLRTFHTHR ILWCKAPVKP GIPYKQLTVG VPKEIFQNEK RVALSPAGVQ ALVKQGFNV VVESGAGEAS KFSDDHYRAA GAQIQGAKEV LASDLVVKVR APMLNPTLGI HEADLLKTSG TLISFIYPAQ N PDLLNKLS KRNTTVLAMD QVPRVTIAQG YDALSSMANI AGYKAVVLAA NHFGRFFTGQ ITAAGKVPPA KILIVGGGVA GL ASAGAAK SMGAIVRGFD TRAAALEQFK SLGAEPLEVD LKESGEGQGG YAKEMSKEFI EAEMKLFAQQ CKEVDILIST ALI PGKKAP ILFNKEMIES MKEGSVVVDL AAEAGGNFET TKPGELYVHK GITHIGYTDL PSRMATQAST LYSNNITKLL KAIS PDKDN FYFEVKDDFD FGTMGHVIRG TVVMKDGQVI FPAPTPKNIP QGAPVKQKTV AELEAEKAAT ITPFRKTMTS ASVYT AGLT GILGLGIAAP NLAFSQMVTT FGLAGIVGYH TVWGVTPALH SPLMSVTNAI SGLTAVGGLV LMGGHLYPST TSQGLA ALA TFISSVNIAG GFLVTQRMLD MFKRPTDPPE YNYLYLLPAG TFVGGYLASL YSGYNIEQIM YLGSGLCCVG ALAGLST QG TARLGNALGM IGVAGGLAAT LGGLKPCPEL LAQMSGAMAL GGTIGLTIAK RIQISDLPQL VAAFHSLVGL AAVLTCIA E YIIEYPHFAT DAAANLTKIV AYLGTYIGGV TFSGSLVAYG KLQGILKSAP LLLPGRHLLN AGLLAASVGG IIPFMMDPS FTTGITCLGS VSALSAVMGV TLTAAIGGAD MPVVITVLNS YSGWALCAEG FLLNNNLLTI VGALIGSSGA ILSYIMCVAM NRSLANVIL GGYGTTSTAG GKPMEISGTH TEINLDNAID MIREANSIII TPGYGLCAAK AQYPIADLVK MLSEQGKKVR F GIHPVAGR MPGQLNVLLA EAGVPYDIVL EMDEINHDFP DTDLVLVIGA NDTVNSAAQE DPNSIIAGMP VLEVWKSKQV IV MKRSLGV GYAAVDNPIF YKPNTAMLLG DAKKTCDALQ AKVRESYQK UniProtKB: NAD(P) transhydrogenase, mitochondrial |
-分子 #2: NADP NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE
| 分子 | 名称: NADP NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: NAP |
|---|---|
| 分子量 | 理論値: 743.405 Da |
| Chemical component information |  ChemComp-NAP: |
-分子 #3: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| 分子 | 名称: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: NAD |
|---|---|
| 分子量 | 理論値: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-分子 #4: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 4 / コピー数: 12 / 式: PC1 |
|---|---|
| 分子量 | 理論値: 790.145 Da |
| Chemical component information |  ChemComp-PC1: |
-分子 #5: DODECANE
| 分子 | 名称: DODECANE / タイプ: ligand / ID: 5 / コピー数: 3 / 式: D12 |
|---|---|
| 分子量 | 理論値: 170.335 Da |
| Chemical component information |  ChemComp-D12: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.0007 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 4-6 s blotting time. | ||||||||||||
| 詳細 | Sample was purified in LMNG (final conc. 0.06%) and contained 0.05% CHAPS as a secondary detergent as well as 5 mM NADP+. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 2160 / 平均露光時間: 10.0 sec. / 平均電子線量: 72.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 75.7 当てはまり具合の基準: Correlation coefficient, EMRinger score |
| 得られたモデル |  PDB-6qti: |
 ムービー
ムービー コントローラー
コントローラー



















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)