+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | V0-only V-ATPase in synaptophysin gene knock-out mouse brain isolated synaptic vesicles | |||||||||
 マップデータ マップデータ | Full map of V0-only V-ATPase in SYP-KO ISVs. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | V0only V-ATPase / synaptic vesicle / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Ion channel transport / Amino acids regulate mTORC1 / Transferrin endocytosis and recycling / Insulin receptor recycling / Metabolism of Angiotensinogen to Angiotensins / eye pigmentation / central nervous system maturation / rostrocaudal neural tube patterning / cellular response to increased oxygen levels / positive regulation of transforming growth factor beta1 production ...Ion channel transport / Amino acids regulate mTORC1 / Transferrin endocytosis and recycling / Insulin receptor recycling / Metabolism of Angiotensinogen to Angiotensins / eye pigmentation / central nervous system maturation / rostrocaudal neural tube patterning / cellular response to increased oxygen levels / positive regulation of transforming growth factor beta1 production / ROS and RNS production in phagocytes / transporter activator activity / proton-transporting V-type ATPase, V0 domain / RHOA GTPase cycle / synaptic vesicle lumen acidification / P-type proton-exporting transporter activity / clathrin-coated vesicle membrane / endosome to plasma membrane protein transport / vacuolar proton-transporting V-type ATPase, V0 domain / lysosomal lumen acidification / endosomal lumen acidification / proton-transporting V-type ATPase complex / vacuolar proton-transporting V-type ATPase complex / osteoclast development / head morphogenesis / vacuolar acidification / dendritic spine membrane / ATPase activator activity / regulation of MAPK cascade / autophagosome membrane / cilium assembly / positive regulation of Wnt signaling pathway / regulation of macroautophagy / angiotensin maturation / receptor-mediated endocytosis of virus by host cell / axon terminus / proton-transporting ATPase activity, rotational mechanism / Neutrophil degranulation / endoplasmic reticulum-Golgi intermediate compartment membrane / RNA endonuclease activity / endomembrane system / proton transmembrane transport / receptor-mediated endocytosis / small GTPase binding / synaptic vesicle membrane / positive regulation of canonical Wnt signaling pathway / melanosome / signaling receptor activity / ATPase binding / postsynaptic membrane / intracellular iron ion homeostasis / 加水分解酵素; エステル加水分解酵素 / early endosome / endosome / endosome membrane / nuclear speck / apical plasma membrane / axon / external side of plasma membrane / lysosomal membrane / centrosome / ubiquitin protein ligase binding / endoplasmic reticulum membrane / protein-containing complex binding / perinuclear region of cytoplasm / Golgi apparatus / membrane / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Wang C / Jiang W / Yang K / Wang X / Guo Q / Brunger AT | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structure and topography of the synaptic V-ATPase-synaptophysin complex. 著者: Chuchu Wang / Wenhong Jiang / Jeremy Leitz / Kailu Yang / Luis Esquivies / Xing Wang / Xiaotao Shen / Richard G Held / Daniel J Adams / Tamara Basta / Lucas Hampton / Ruiqi Jian / Lihua Jiang ...著者: Chuchu Wang / Wenhong Jiang / Jeremy Leitz / Kailu Yang / Luis Esquivies / Xing Wang / Xiaotao Shen / Richard G Held / Daniel J Adams / Tamara Basta / Lucas Hampton / Ruiqi Jian / Lihua Jiang / Michael H B Stowell / Wolfgang Baumeister / Qiang Guo / Axel T Brunger /     要旨: Synaptic vesicles are organelles with a precisely defined protein and lipid composition, yet the molecular mechanisms for the biogenesis of synaptic vesicles are mainly unknown. Here we discovered a ...Synaptic vesicles are organelles with a precisely defined protein and lipid composition, yet the molecular mechanisms for the biogenesis of synaptic vesicles are mainly unknown. Here we discovered a well-defined interface between the synaptic vesicle V-ATPase and synaptophysin by in situ cryo-electron tomography and single-particle cryo-electron microscopy of functional synaptic vesicles isolated from mouse brains. The synaptic vesicle V-ATPase is an ATP-dependent proton pump that establishes the proton gradient across the synaptic vesicle, which in turn drives the uptake of neurotransmitters. Synaptophysin and its paralogues synaptoporin and synaptogyrin belong to a family of abundant synaptic vesicle proteins whose function is still unclear. We performed structural and functional studies of synaptophysin-knockout mice, confirming the identity of synaptophysin as an interaction partner with the V-ATPase. Although there is little change in the conformation of the V-ATPase upon interaction with synaptophysin, the presence of synaptophysin in synaptic vesicles profoundly affects the copy number of V-ATPases. This effect on the topography of synaptic vesicles suggests that synaptophysin assists in their biogenesis. In support of this model, we observed that synaptophysin-knockout mice exhibit severe seizure susceptibility, suggesting an imbalance of neurotransmitter release as a physiological consequence of the absence of synaptophysin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44845.map.gz emd_44845.map.gz | 86.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44845-v30.xml emd-44845-v30.xml emd-44845.xml emd-44845.xml | 28.3 KB 28.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_44845.png emd_44845.png | 156.9 KB | ||
| Filedesc metadata |  emd-44845.cif.gz emd-44845.cif.gz | 7.8 KB | ||
| その他 |  emd_44845_additional_1.map.gz emd_44845_additional_1.map.gz emd_44845_additional_2.map.gz emd_44845_additional_2.map.gz emd_44845_half_map_1.map.gz emd_44845_half_map_1.map.gz emd_44845_half_map_2.map.gz emd_44845_half_map_2.map.gz | 81.9 MB 71.3 MB 84.5 MB 84.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44845 http://ftp.pdbj.org/pub/emdb/structures/EMD-44845 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44845 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44845 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9bryMC  9braC  9brqC  9brrC  9brsC  9brtC  9bruC  9brzC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44845.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44845.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full map of V0-only V-ATPase in SYP-KO ISVs. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1064 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: EMhancer full map of V0-only V-ATPase in SYP-KO ISVs.
| ファイル | emd_44845_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EMhancer full map of V0-only V-ATPase in SYP-KO ISVs. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
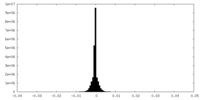
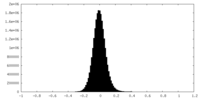
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_44845_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
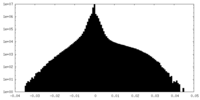
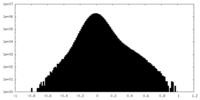
| 密度ヒストグラム |
-ハーフマップ: Half map B of V0-only V-ATPase in SYP-KO ISVs.
| ファイル | emd_44845_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of V0-only V-ATPase in SYP-KO ISVs. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
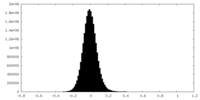
| 密度ヒストグラム |
-ハーフマップ: Half map A of V0-only V-ATPase in SYP-KO ISVs.
| ファイル | emd_44845_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of V0-only V-ATPase in SYP-KO ISVs. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
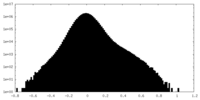
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Syptophysin gene knock-out mouse brain isolated glutamatergic syn...
+超分子 #1: Syptophysin gene knock-out mouse brain isolated glutamatergic syn...
+分子 #1: V-type proton ATPase subunit S1
+分子 #2: V-type proton ATPase 21 kDa proteolipid subunit c''
+分子 #3: V-type proton ATPase subunit d 1
+分子 #4: Ribonuclease kappa
+分子 #5: V-type proton ATPase 16 kDa proteolipid subunit c
+分子 #6: Renin receptor cytoplasmic fragment
+分子 #7: V-type proton ATPase subunit e 2
+分子 #8: V-type proton ATPase 116 kDa subunit a 1
+分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
+分子 #12: beta-D-mannopyranose
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 詳細 | The specimen state should be an intact subcellular component. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 53390 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 詳細 | The initial model is the model of wild-type V0-only V-ATPase. For fitting and refinement details, see https://doi.org/10.1038/s41586-024-07610-x. |
|---|---|
| 精密化 | プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-9bry: |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)