+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4406 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure. | |||||||||
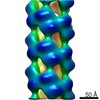
 マップデータ マップデータ | AtNIT4 R95T L169A S224Q filament | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Mulelu AE / Woodward JD | |||||||||
| 資金援助 |  南アフリカ, 1件 南アフリカ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2019 ジャーナル: Commun Biol / 年: 2019タイトル: Cryo-EM and directed evolution reveal how nitrilase specificity is influenced by its quaternary structure. 著者: Andani E Mulelu / Angela M Kirykowicz / Jeremy D Woodward /  要旨: Nitrilases are helical enzymes that convert nitriles to acids and/or amides. All plants have a nitrilase 4 homolog specific for ß-cyanoalanine, while in some plants neofunctionalization has produced ...Nitrilases are helical enzymes that convert nitriles to acids and/or amides. All plants have a nitrilase 4 homolog specific for ß-cyanoalanine, while in some plants neofunctionalization has produced nitrilases with altered specificity. Plant nitrilase substrate size and specificity correlate with helical twist, but molecular details of this relationship are lacking. Here we determine, to our knowledge, the first close-to-atomic resolution (3.4 Å) cryo-EM structure of an active helical nitrilase, the nitrilase 4 from . We apply site-saturation mutagenesis directed evolution to three residues (R95, S224, and L169) and generate a mutant with an altered helical twist that accepts substrates not catalyzed by known plant nitrilases. We reveal that a loop between α2 and α3 limits the length of the binding pocket and propose that it shifts position as a function of helical twist. These insights will allow us to start designing nitrilases for chemoenzymatic synthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4406.map.gz emd_4406.map.gz | 346.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4406-v30.xml emd-4406-v30.xml emd-4406.xml emd-4406.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4406.png emd_4406.png | 62.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4406 http://ftp.pdbj.org/pub/emdb/structures/EMD-4406 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4406 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4406 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4406_validation.pdf.gz emd_4406_validation.pdf.gz | 205.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4406_full_validation.pdf.gz emd_4406_full_validation.pdf.gz | 204.8 KB | 表示 | |
| XML形式データ |  emd_4406_validation.xml.gz emd_4406_validation.xml.gz | 5.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4406 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4406 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4406 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4406 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4406.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4406.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AtNIT4 R95T L169A S224Q filament | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Arabidopsis thaliana NITRILASE 4 mutant
| 全体 | 名称: Arabidopsis thaliana NITRILASE 4 mutant |
|---|---|
| 要素 |
|
-超分子 #1: Arabidopsis thaliana NITRILASE 4 mutant
| 超分子 | 名称: Arabidopsis thaliana NITRILASE 4 mutant / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Generated by directed evolution |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 400 KDa |
-分子 #1: NITRILASE 4 mutant
| 分子 | 名称: NITRILASE 4 mutant / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: nitrilase |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSMQQETSHM TAAPQTNGHQ IFPEIDMSAG DSSSIVRATV VQASTVFYDT PATLDKAERL LSEAAENGSQ LVVFPEAFIG GYPRGSTFEL AIGSTTAKGR DDFRKYHASA IDVPGPEVER LALMAKKYKV YLVMGVIERE GYTLYCTVLF FDSQGLFLGK HRKLMPTAAE ...文字列: MSMQQETSHM TAAPQTNGHQ IFPEIDMSAG DSSSIVRATV VQASTVFYDT PATLDKAERL LSEAAENGSQ LVVFPEAFIG GYPRGSTFEL AIGSTTAKGR DDFRKYHASA IDVPGPEVER LALMAKKYKV YLVMGVIERE GYTLYCTVLF FDSQGLFLGK HRKLMPTAAE RCIWGFGDGS TIPVFDTPIG KIGAAICWEN RMPSLRTAMY AKGIEIYCAP TADQRETWLA SMTHIALEGG CFVLSANQFC RRKDYPSPPE YMFSGSEESL TPDSVVCAGG SSIISPLGIV LAGPNYRGEA LITADLDLGD IARAKFDFDV VGHYSRPEVF SLNIREHPRK AVSFKTSKVM EDESVHHHHH H |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl acetate | |||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 15.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 撮影したグリッド数: 1 / 実像数: 50 / 平均露光時間: 1.0 sec. / 平均電子線量: 10.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.1 mm / 最大 デフォーカス(公称値): 0.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: PHILIPS ROTATION HOLDER |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















