+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the PP2A:B55-FAM122A complex, PP2Ac body | ||||||||||||
 マップデータ マップデータ | Relion multi-body refinement, PP2Ac body, full map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Protein Phosphatase 2A:B55 holoenzyme FAM122A inhibitor substrate binding cell cycle regulation / SIGNALING PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein serine/threonine phosphatase inhibitor activity / meiotic spindle elongation / Integration of energy metabolism / PP2A-mediated dephosphorylation of key metabolic factors / regulation of microtubule binding / MASTL Facilitates Mitotic Progression / regulation of meiotic cell cycle process involved in oocyte maturation / mitotic sister chromatid separation / protein serine/threonine phosphatase complex / protein phosphatase type 2A complex ...protein serine/threonine phosphatase inhibitor activity / meiotic spindle elongation / Integration of energy metabolism / PP2A-mediated dephosphorylation of key metabolic factors / regulation of microtubule binding / MASTL Facilitates Mitotic Progression / regulation of meiotic cell cycle process involved in oocyte maturation / mitotic sister chromatid separation / protein serine/threonine phosphatase complex / protein phosphatase type 2A complex / meiotic sister chromatid cohesion, centromeric / peptidyl-serine dephosphorylation / peptidyl-threonine dephosphorylation / FAR/SIN/STRIPAK complex / Regulation of glycolysis by fructose 2,6-bisphosphate metabolism / positive regulation of microtubule binding / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / female meiotic nuclear division / protein antigen binding / GABA receptor binding / protein phosphatase regulator activity / APC truncation mutants have impaired AXIN binding / AXIN missense mutants destabilize the destruction complex / Truncations of AMER1 destabilize the destruction complex / Initiation of Nuclear Envelope (NE) Reformation / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / ERKs are inactivated / Beta-catenin phosphorylation cascade / Signaling by GSK3beta mutants / CTNNB1 S33 mutants aren't phosphorylated / CTNNB1 S37 mutants aren't phosphorylated / CTNNB1 S45 mutants aren't phosphorylated / CTNNB1 T41 mutants aren't phosphorylated / mitotic G2/M transition checkpoint / regulation of growth / Disassembly of the destruction complex and recruitment of AXIN to the membrane / negative regulation of epithelial to mesenchymal transition / negative regulation of glycolytic process through fructose-6-phosphate / positive regulation of NLRP3 inflammasome complex assembly / Platelet sensitization by LDL / CTLA4 inhibitory signaling / protein serine/threonine phosphatase activity / myosin phosphatase activity / protein-serine/threonine phosphatase / regulation of cell differentiation / T cell homeostasis / ERK/MAPK targets / mesoderm development / regulation of G1/S transition of mitotic cell cycle / phosphoprotein phosphatase activity / chromosome, centromeric region / DARPP-32 events / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / lateral plasma membrane / negative regulation of hippo signaling / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / Cyclin A/B1/B2 associated events during G2/M transition / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / Loss of Nlp from mitotic centrosomes / Loss of proteins required for interphase microtubule organization from the centrosome / Recruitment of mitotic centrosome proteins and complexes / Recruitment of NuMA to mitotic centrosomes / Anchoring of the basal body to the plasma membrane / Resolution of Sister Chromatid Cohesion / protein dephosphorylation / AURKA Activation by TPX2 / protein tyrosine phosphatase activity / meiotic cell cycle / chromosome segregation / RHO GTPases Activate Formins / response to lead ion / RAF activation / Spry regulation of FGF signaling / regulation of protein phosphorylation / positive regulation of protein serine/threonine kinase activity / tau protein binding / Degradation of beta-catenin by the destruction complex / PKR-mediated signaling / spindle pole / Negative regulation of MAPK pathway / Separation of Sister Chromatids / Cyclin D associated events in G1 / microtubule cytoskeleton / Regulation of PLK1 Activity at G2/M Transition / Regulation of TP53 Degradation / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / mitotic cell cycle / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / positive regulation of cell growth / protein-containing complex assembly / nuclear body / intracellular signal transduction / neuron projection / protein heterodimerization activity / membrane raft / neuronal cell body / glutamatergic synapse / dendrite 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
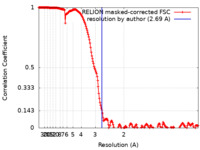
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.69 Å | ||||||||||||
 データ登録者 データ登録者 | Fuller JR / Padi SKR / Peti W / Page R | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 | ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018 タイトル: Real-space refinement in PHENIX for cryo-EM and crystallography. 著者: Pavel V Afonine / Billy K Poon / Randy J Read / Oleg V Sobolev / Thomas C Terwilliger / Alexandre Urzhumtsev / Paul D Adams /    要旨: This article describes the implementation of real-space refinement in the phenix.real_space_refine program from the PHENIX suite. The use of a simplified refinement target function enables very fast ...This article describes the implementation of real-space refinement in the phenix.real_space_refine program from the PHENIX suite. The use of a simplified refinement target function enables very fast calculation, which in turn makes it possible to identify optimal data-restraint weights as part of routine refinements with little runtime cost. Refinement of atomic models against low-resolution data benefits from the inclusion of as much additional information as is available. In addition to standard restraints on covalent geometry, phenix.real_space_refine makes use of extra information such as secondary-structure and rotamer-specific restraints, as well as restraints or constraints on internal molecular symmetry. The re-refinement of 385 cryo-EM-derived models available in the Protein Data Bank at resolutions of 6 Å or better shows significant improvement of the models and of the fit of these models to the target maps. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41668.map.gz emd_41668.map.gz | 306.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41668-v30.xml emd-41668-v30.xml emd-41668.xml emd-41668.xml | 26.7 KB 26.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41668_fsc.xml emd_41668_fsc.xml | 19.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41668.png emd_41668.png | 148.4 KB | ||
| マスクデータ |  emd_41668_msk_1.map emd_41668_msk_1.map | 600.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41668.cif.gz emd-41668.cif.gz | 7.6 KB | ||
| その他 |  emd_41668_additional_1.map.gz emd_41668_additional_1.map.gz emd_41668_half_map_1.map.gz emd_41668_half_map_1.map.gz emd_41668_half_map_2.map.gz emd_41668_half_map_2.map.gz | 364.2 MB 435.2 MB 435.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41668 http://ftp.pdbj.org/pub/emdb/structures/EMD-41668 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41668 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41668 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41668_validation.pdf.gz emd_41668_validation.pdf.gz | 732.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41668_full_validation.pdf.gz emd_41668_full_validation.pdf.gz | 731.8 KB | 表示 | |
| XML形式データ |  emd_41668_validation.xml.gz emd_41668_validation.xml.gz | 26.8 KB | 表示 | |
| CIF形式データ |  emd_41668_validation.cif.gz emd_41668_validation.cif.gz | 36 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41668 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41668 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41668 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41668 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41668.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41668.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion multi-body refinement, PP2Ac body, full map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.534 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41668_msk_1.map emd_41668_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
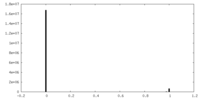
| 密度ヒストグラム |
-追加マップ: Map sharpened by an automatically-determined B-factor (-71.9061) and...
| ファイル | emd_41668_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map sharpened by an automatically-determined B-factor (-71.9061) and filtered to local resolution by the Relion locres implementation | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
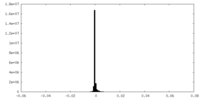
| 密度ヒストグラム |
-ハーフマップ: Relion multi-body refinement, PP2Ac body, half map 1
| ファイル | emd_41668_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion multi-body refinement, PP2Ac body, half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
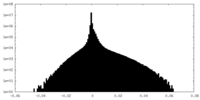
| 密度ヒストグラム |
-ハーフマップ: Relion multi-body refinement, PP2Ac body, half map 2
| ファイル | emd_41668_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion multi-body refinement, PP2Ac body, half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
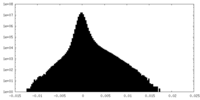
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Quadruple complex of PP2A:B55 (PP2Aa:PP2Ac:B55) bound to FAM122A
| 全体 | 名称: Quadruple complex of PP2A:B55 (PP2Aa:PP2Ac:B55) bound to FAM122A |
|---|---|
| 要素 |
|
-超分子 #1: Quadruple complex of PP2A:B55 (PP2Aa:PP2Ac:B55) bound to FAM122A
| 超分子 | 名称: Quadruple complex of PP2A:B55 (PP2Aa:PP2Ac:B55) bound to FAM122A タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-分子 #1: Serine/threonine-protein phosphatase 2A 65 kDa regulatory subunit...
| 分子 | 名称: Serine/threonine-protein phosphatase 2A 65 kDa regulatory subunit A alpha isoform タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 64.95798 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GHMSLYPIAV LIDELRNEDV QLRLNSIKKL STIALALGVE RTRSELLPFL TDTIYDEDEV LLALAEQLGT FTTLVGGPEY VHCLLPPLE SLATVEETVV RDKAVESLRA ISHEHSPSDL EAHFVPLVKR LAGGDWFTSR TSACGLFSVC YPRVSSAVKA E LRQYFRNL ...文字列: GHMSLYPIAV LIDELRNEDV QLRLNSIKKL STIALALGVE RTRSELLPFL TDTIYDEDEV LLALAEQLGT FTTLVGGPEY VHCLLPPLE SLATVEETVV RDKAVESLRA ISHEHSPSDL EAHFVPLVKR LAGGDWFTSR TSACGLFSVC YPRVSSAVKA E LRQYFRNL CSDDTPMVRR AAASKLGEFA KVLELDNVKS EIIPMFSNLA SDEQDSVRLL AVEACVNIAQ LLPQEDLEAL VM PTLRQAA EDKSWRVRYM VADKFTELQK AVGPEITKTD LVPAFQNLMK DCEAEVRAAA SHKVKEFCEN LSADCRENVI MSQ ILPCIK ELVSDANQHV KSALASVIMG LSPILGKDNT IEHLLPLFLA QLKDECPEVR LNIISNLDCV NEVIGIRQLS QSLL PAIVE LAEDAKWRVR LAIIEYMPLL AGQLGVEFFD EKLNSLCMAW LVDHVYAIRE AATSNLKKLV EKFGKEWAHA TIIPK VLAM SGDPNYLHRM TTLFCINVLS EVCGQDITTK HMLPTVLRMA GDPVANVRFN VAKSLQKIGP ILDNSTLQSE VKPILE KLT QDQDVDVKYF AQEALTVLSL A UniProtKB: Serine/threonine-protein phosphatase 2A 65 kDa regulatory subunit A alpha isoform |
-分子 #2: Serine/threonine-protein phosphatase 2A catalytic subunit alpha i...
| 分子 | 名称: Serine/threonine-protein phosphatase 2A catalytic subunit alpha isoform タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: protein-serine/threonine phosphatase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 35.845375 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GHMDEKVFTK ELDQWIEQLN ECKQLSESQV KSLCEKAKEI LTKESNVQEV RCPVTVCGDV HGQFHDLMEL FRIGGKSPDT NYLFMGDYV DRGYYSVETV TLLVALKVRY RERITILRGN HESRQITQVY GFYDECLRKY GNANVWKYFT DLFDYLPLTA L VDGQIFCL ...文字列: GHMDEKVFTK ELDQWIEQLN ECKQLSESQV KSLCEKAKEI LTKESNVQEV RCPVTVCGDV HGQFHDLMEL FRIGGKSPDT NYLFMGDYV DRGYYSVETV TLLVALKVRY RERITILRGN HESRQITQVY GFYDECLRKY GNANVWKYFT DLFDYLPLTA L VDGQIFCL HGGLSPSIDT LDHIRALDRL QEVPHEGPMC DLLWSDPDDR GGWGISPRGA GYTFGQDISE TFNHANGLTL VS RAHQLVM EGYNWCHDRN VVTIFSAPNY CYRCGNQAAI MELDDTLKYS FLQFDPAPRR GEPHVTRRTP DYF(MLL) UniProtKB: Serine/threonine-protein phosphatase 2A catalytic subunit alpha isoform |
-分子 #3: PPP2R1A-PPP2R2A-interacting phosphatase regulator 1
| 分子 | 名称: PPP2R1A-PPP2R2A-interacting phosphatase regulator 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.680907 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GHMGGGLRRS NSAPLIHGLS DSSPVFQDEA PSARRNRTTF PSRHGLLLPA SPVRMHSSRL HQIKQEEGMD LINRETVHER EVQTAMQIS HSWEES UniProtKB: PPP2R1A-PPP2R2A-interacting phosphatase regulator 1 |
-分子 #4: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: CHAPSO was added only immediately prior to vitrification | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 平均電子線量: 70.0 e/Å2 詳細: Camera was operated in CDS mode, writing super-resolution movies with 59 frames |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm 最大 デフォーカス(補正後): 2.5300000000000002 µm 最小 デフォーカス(補正後): 0.55 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 詳細 | Iterating between manual refinement in Coot and automated real-space refinement in Phenix | ||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Cross-correlation | ||||||
| 得られたモデル |  PDB-8twi: |
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)