+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4117 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryoEM structure of crenactin double helical filament at 3.8A resolution | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / cytoskeleton / hydrolase activity / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Pyrobaculum calidifontis (古細菌) / Pyrobaculum calidifontis (古細菌) /   Pyrobaculum calidifontis (strain JCM 11548 / VA1) (古細菌) Pyrobaculum calidifontis (strain JCM 11548 / VA1) (古細菌) | |||||||||
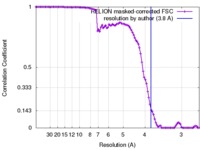
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Izore T / Lowe J | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2016 ジャーナル: Elife / 年: 2016タイトル: Crenactin forms actin-like double helical filaments regulated by arcadin-2. 著者: Thierry Izoré / Danguole Kureisaite-Ciziene / Stephen H McLaughlin / Jan Löwe /  要旨: The similarity of eukaryotic actin to crenactin, a filament-forming protein from the crenarchaeon supports the theory of a common origin of Crenarchaea and Eukaryotes. Monomeric structures of ...The similarity of eukaryotic actin to crenactin, a filament-forming protein from the crenarchaeon supports the theory of a common origin of Crenarchaea and Eukaryotes. Monomeric structures of crenactin and actin are similar, although their filament architectures were suggested to be different. Here we report that crenactin forms double helical filaments that show exceptional similarity to eukaryotic F-actin. With cryo-electron microscopy and helical reconstruction we solved the structure of the crenactin filament to 3.8 Å resolution. When forming double filaments, the 'hydrophobic plug' loop in crenactin rearranges. Arcadin-2, also encoded by the arcade gene cluster, binds tightly with its C-terminus to the hydrophobic groove of crenactin. Binding is reminiscent of eukaryotic actin modulators such as cofilin and thymosin β4 and arcadin-2 is a depolymeriser of crenactin filaments. Our work further supports the theory of shared ancestry of Eukaryotes and Crenarchaea. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4117.map.gz emd_4117.map.gz | 78.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4117-v30.xml emd-4117-v30.xml emd-4117.xml emd-4117.xml | 10.7 KB 10.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4117_fsc.xml emd_4117_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4117.png emd_4117.png | 38.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4117 http://ftp.pdbj.org/pub/emdb/structures/EMD-4117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4117 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4117_validation.pdf.gz emd_4117_validation.pdf.gz | 309.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4117_full_validation.pdf.gz emd_4117_full_validation.pdf.gz | 308.9 KB | 表示 | |
| XML形式データ |  emd_4117_validation.xml.gz emd_4117_validation.xml.gz | 11 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4117 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4117 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4117 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4117 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4117.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4117.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : crenactin
| 全体 | 名称: crenactin |
|---|---|
| 要素 |
|
-超分子 #1: crenactin
| 超分子 | 名称: crenactin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Pyrobaculum calidifontis (古細菌) Pyrobaculum calidifontis (古細菌) |
| 組換発現 | 生物種:  |
-分子 #1: Actin/actin family protein
| 分子 | 名称: Actin/actin family protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pyrobaculum calidifontis (strain JCM 11548 / VA1) (古細菌) Pyrobaculum calidifontis (strain JCM 11548 / VA1) (古細菌)株: JCM 11548 / VA1 |
| 分子量 | 理論値: 48.425484 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGVISDAYRL KYTFGVDFGT SYVKYGPITL NEPKMVQTRG LFLRDLPESV KMRIPPDVLA RGLVVGDEEV RKYLSSVRDV QRNLKYPLK DGVARRDDEE AWRVLKELAR YTLAQFPVSD PEFAGWLVAV ALSALAPDYM YKAIFDIYDE LASEFKIYAV T ILPQPLAV ...文字列: MGVISDAYRL KYTFGVDFGT SYVKYGPITL NEPKMVQTRG LFLRDLPESV KMRIPPDVLA RGLVVGDEEV RKYLSSVRDV QRNLKYPLK DGVARRDDEE AWRVLKELAR YTLAQFPVSD PEFAGWLVAV ALSALAPDYM YKAIFDIYDE LASEFKIYAV T ILPQPLAV AIAENAVNCV IVEGGHGNIQ VAPISFALIR EGLVALNRGG AEANAITREI LKDIGYSDIA REEYAVEVVK RA VGLVPRR LKEAIRAAKS DPDRFVTKVR LSPVVEVEIP REYAWTRFLI GEIVFDPNHE EIKSYIEQSR LRIENAVIGD VTL YGEMDV ASAIITSLRN VSVEIQERVA SQIILSGGAF SWRVPPGMED VAADSVTRVK IALEEKSPAL ASKVEVRLVS EPQY SVWRG AVIYGYALPL SLEWSDTTRE GWRFPRR |
-分子 #2: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 実像数: 1474 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: OTHER / 当てはまり具合の基準: R-factor & stereochemistry |
|---|---|
| 得られたモデル |  PDB-5mw1: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)