+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3928 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human mTORC2 core complex accessory factors | |||||||||
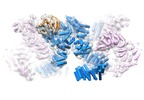
 マップデータ マップデータ | mTORC2 core complex accessory factor density | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / TORC2 signaling / TORC2 complex / regulation of cellular response to oxidative stress / phosphatidic acid binding / negative regulation of Ras protein signal transduction / serine/threonine protein kinase complex / embryo development ending in birth or egg hatching / phosphatidylinositol-3,4-bisphosphate binding / phosphatidylinositol-3,5-bisphosphate binding ...regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / TORC2 signaling / TORC2 complex / regulation of cellular response to oxidative stress / phosphatidic acid binding / negative regulation of Ras protein signal transduction / serine/threonine protein kinase complex / embryo development ending in birth or egg hatching / phosphatidylinositol-3,4-bisphosphate binding / phosphatidylinositol-3,5-bisphosphate binding / regulation of establishment of cell polarity / lipid biosynthetic process / Constitutive Signaling by AKT1 E17K in Cancer / positive regulation of actin filament polymerization / phosphatidylinositol-3,4,5-trisphosphate binding / CD28 dependent PI3K/Akt signaling / positive regulation of TOR signaling / enzyme-substrate adaptor activity / cellular response to nutrient levels / positive regulation of endothelial cell proliferation / phosphatidylinositol-4,5-bisphosphate binding / cytoskeleton organization / substantia nigra development / negative regulation of insulin receptor signaling pathway / protein serine/threonine kinase activator activity / VEGFR2 mediated vascular permeability / regulation of actin cytoskeleton organization / phosphatidylinositol 3-kinase/protein kinase B signal transduction / small GTPase binding / cellular response to insulin stimulus / late endosome membrane / Regulation of TP53 Degradation / late endosome / PIP3 activates AKT signaling / ribosome binding / regulation of gene expression / actin cytoskeleton organization / positive regulation of cell growth / regulation of inflammatory response / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / early endosome membrane / molecular adaptor activity / mitochondrial outer membrane / early endosome / lysosome / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / cilium / positive regulation of cell migration / Golgi membrane / lysosomal membrane / intracellular membrane-bounded organelle / endoplasmic reticulum membrane / protein kinase binding / negative regulation of apoptotic process / perinuclear region of cytoplasm / endoplasmic reticulum / Golgi apparatus / zinc ion binding / nucleoplasm / ATP binding / nucleus / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||
 データ登録者 データ登録者 | Scaiola A / Aylett CHS / Boehringer D / Stuttfeld E / Imseng S / Sauer E / Hall MN / Maier T / Ban N | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2018 ジャーナル: Elife / 年: 2018タイトル: Architecture of the human mTORC2 core complex. 著者: Edward Stuttfeld / Christopher Hs Aylett / Stefan Imseng / Daniel Boehringer / Alain Scaiola / Evelyn Sauer / Michael N Hall / Timm Maier / Nenad Ban /  要旨: The mammalian target of rapamycin (mTOR) is a key protein kinase controlling cellular metabolism and growth. It is part of the two structurally and functionally distinct multiprotein complexes mTORC1 ...The mammalian target of rapamycin (mTOR) is a key protein kinase controlling cellular metabolism and growth. It is part of the two structurally and functionally distinct multiprotein complexes mTORC1 and mTORC2. Dysregulation of mTOR occurs in diabetes, cancer and neurological disease. We report the architecture of human mTORC2 at intermediate resolution, revealing a conserved binding site for accessory proteins on mTOR and explaining the structural basis for the rapamycin insensitivity of the complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3928.map.gz emd_3928.map.gz | 194 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3928-v30.xml emd-3928-v30.xml emd-3928.xml emd-3928.xml | 12.8 KB 12.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3928_fsc.xml emd_3928_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3928.png emd_3928.png | 209.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3928 http://ftp.pdbj.org/pub/emdb/structures/EMD-3928 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3928 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3928 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3928_validation.pdf.gz emd_3928_validation.pdf.gz | 254.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3928_full_validation.pdf.gz emd_3928_full_validation.pdf.gz | 254 KB | 表示 | |
| XML形式データ |  emd_3928_validation.xml.gz emd_3928_validation.xml.gz | 13.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3928 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3928 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3928 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3928 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3928.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3928.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | mTORC2 core complex accessory factor density | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human mTORC2 core complex accessory factors
| 全体 | 名称: Human mTORC2 core complex accessory factors |
|---|---|
| 要素 |
|
-超分子 #1: Human mTORC2 core complex accessory factors
| 超分子 | 名称: Human mTORC2 core complex accessory factors / タイプ: complex / ID: 1 / 親要素: 0 詳細: Accessory factor density for the mTORC2 core complex; Rictor, mSIN1 and Protor-1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  組換細胞: Sf9 / 組換プラスミド: Multibac |
| 分子量 | 理論値: 250 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 150 mM NaCl, 15 mM NaBicine pH 8.0, 1 mM TCEP |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Two seconds blotting. |
| 詳細 | Sample was prepared by a modified GRAFIX protocol described in the paper, and was extremely dilute. Particles were adhered to a thin carbon film to obtain reasonable coverage. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 100.0 K / 最高: 100.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 3997 / 平均露光時間: 20.0 sec. / 平均電子線量: 80.0 e/Å2 / 詳細: Dose weighting applied |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 47100 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















