+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3669 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Closed dimer (C1) of human ATM (Ataxia telangiectasia mutated) | |||||||||
 マップデータ マップデータ | Closed dimer (C1) of human ATM (Ataxia telangiectasia mutated) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PIKK / kinase / DNA-repair / HEAT-repeats / SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報establishment of RNA localization to telomere / positive regulation of telomerase catalytic core complex assembly / cellular response to nitrosative stress / negative regulation of telomere capping / Sensing of DNA Double Strand Breaks / establishment of protein-containing complex localization to telomere / peptidyl-serine autophosphorylation / meiotic telomere clustering / positive regulation of telomere maintenance via telomere lengthening / pre-B cell allelic exclusion ...establishment of RNA localization to telomere / positive regulation of telomerase catalytic core complex assembly / cellular response to nitrosative stress / negative regulation of telomere capping / Sensing of DNA Double Strand Breaks / establishment of protein-containing complex localization to telomere / peptidyl-serine autophosphorylation / meiotic telomere clustering / positive regulation of telomere maintenance via telomere lengthening / pre-B cell allelic exclusion / DNA-dependent protein kinase activity / histone mRNA catabolic process / male meiotic nuclear division / extrinsic component of synaptic vesicle membrane / histone H2AXS139 kinase activity / regulation of telomere maintenance via telomerase / female meiotic nuclear division / lipoprotein catabolic process / DNA double-strand break processing / regulation of autophagosome assembly / cellular response to X-ray / V(D)J recombination / pexophagy / oocyte development / Impaired BRCA2 binding to PALB2 / reciprocal meiotic recombination / DNA repair complex / positive regulation of DNA damage response, signal transduction by p53 class mediator / Homologous DNA Pairing and Strand Exchange / Defective homologous recombination repair (HRR) due to BRCA1 loss of function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA1 binding function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA2/RAD51/RAD51C binding function / Resolution of D-loop Structures through Synthesis-Dependent Strand Annealing (SDSA) / 1-phosphatidylinositol-3-kinase activity / Resolution of D-loop Structures through Holliday Junction Intermediates / HDR through Single Strand Annealing (SSA) / response to ionizing radiation / negative regulation of B cell proliferation / TP53 Regulates Transcription of Caspase Activators and Caspases / mitotic spindle assembly checkpoint signaling / positive regulation of double-strand break repair / Impaired BRCA2 binding to RAD51 / cellular response to stress / mitotic G2 DNA damage checkpoint signaling / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / peroxisomal matrix / Presynaptic phase of homologous DNA pairing and strand exchange / replicative senescence / signal transduction in response to DNA damage / Regulation of HSF1-mediated heat shock response / somitogenesis / ovarian follicle development / regulation of cellular response to heat / cellular response to retinoic acid / negative regulation of TORC1 signaling / positive regulation of telomere maintenance via telomerase / telomere maintenance / positive regulation of cell adhesion / Pexophagy / DNA damage checkpoint signaling / thymus development / regulation of signal transduction by p53 class mediator / post-embryonic development / determination of adult lifespan / cellular response to reactive oxygen species / DNA damage response, signal transduction by p53 class mediator / TP53 Regulates Transcription of DNA Repair Genes / Stabilization of p53 / Nonhomologous End-Joining (NHEJ) / Autodegradation of the E3 ubiquitin ligase COP1 / cellular response to gamma radiation / double-strand break repair via homologous recombination / G2/M DNA damage checkpoint / brain development / Regulation of TP53 Activity through Methylation / HDR through Homologous Recombination (HRR) / DNA Damage/Telomere Stress Induced Senescence / double-strand break repair via nonhomologous end joining / Meiotic recombination / spindle / multicellular organism growth / intrinsic apoptotic signaling pathway in response to DNA damage / cellular senescence / Regulation of TP53 Degradation / double-strand break repair / positive regulation of neuron apoptotic process / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / chromosome / site of double-strand break / heart development / protein autophosphorylation / Processing of DNA double-strand break ends / neuron apoptotic process / regulation of apoptotic process / Regulation of TP53 Activity through Phosphorylation / protein phosphorylation / non-specific serine/threonine protein kinase / regulation of cell cycle / regulation of autophagy / positive regulation of cell migration 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
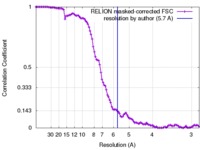
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.7 Å | |||||||||
 データ登録者 データ登録者 | Baretic D / Pollard HK | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2017 ジャーナル: Sci Adv / 年: 2017タイトル: Structures of closed and open conformations of dimeric human ATM. 著者: Domagoj Baretić / Hannah K Pollard / David I Fisher / Christopher M Johnson / Balaji Santhanam / Caroline M Truman / Tomas Kouba / Alan R Fersht / Christopher Phillips / Roger L Williams /  要旨: ATM (ataxia-telangiectasia mutated) is a phosphatidylinositol 3-kinase-related protein kinase (PIKK) best known for its role in DNA damage response. ATM also functions in oxidative stress response, ...ATM (ataxia-telangiectasia mutated) is a phosphatidylinositol 3-kinase-related protein kinase (PIKK) best known for its role in DNA damage response. ATM also functions in oxidative stress response, insulin signaling, and neurogenesis. Our electron cryomicroscopy (cryo-EM) suggests that human ATM is in a dynamic equilibrium between closed and open dimers. In the closed state, the PIKK regulatory domain blocks the peptide substrate-binding site, suggesting that this conformation may represent an inactive or basally active enzyme. The active site is held in this closed conformation by interaction with a long helical hairpin in the TRD3 (tetratricopeptide repeats domain 3) domain of the symmetry-related molecule. The open dimer has two protomers with only a limited contact interface, and it lacks the intermolecular interactions that block the peptide-binding site in the closed dimer. This suggests that the open conformation may be more active. The ATM structure shows the detailed topology of the regulator-interacting N-terminal helical solenoid. The ATM conformational dynamics shown by the structures represent an important step in understanding the enzyme regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3669.map.gz emd_3669.map.gz | 94.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3669-v30.xml emd-3669-v30.xml emd-3669.xml emd-3669.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3669_fsc.xml emd_3669_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3669.png emd_3669.png | 239.2 KB | ||
| Filedesc metadata |  emd-3669.cif.gz emd-3669.cif.gz | 8.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3669 http://ftp.pdbj.org/pub/emdb/structures/EMD-3669 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3669 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3669 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3669_validation.pdf.gz emd_3669_validation.pdf.gz | 283 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3669_full_validation.pdf.gz emd_3669_full_validation.pdf.gz | 282.1 KB | 表示 | |
| XML形式データ |  emd_3669_validation.xml.gz emd_3669_validation.xml.gz | 11.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3669 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3669 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3669 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3669 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3669.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3669.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Closed dimer (C1) of human ATM (Ataxia telangiectasia mutated) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.43 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Dimeric human ATM (Ataxia telangiectasia mutated) kinase
| 全体 | 名称: Dimeric human ATM (Ataxia telangiectasia mutated) kinase |
|---|---|
| 要素 |
|
-超分子 #1: Dimeric human ATM (Ataxia telangiectasia mutated) kinase
| 超分子 | 名称: Dimeric human ATM (Ataxia telangiectasia mutated) kinase タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: homodimer |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 705 KDa |
-分子 #1: Serine-protein kinase ATM
| 分子 | 名称: Serine-protein kinase ATM / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 352.393969 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDDDDKH MSLVLNDLLI CCRQLEHDRA TERKKEVEKF KRLIRDPETI KHLDRHSDSK QGKYLNWDAV FRFLQKYIQK ETECLRIAK PNVSASTQAS RQKKMQEISS LVKYFIKCAN RRAPRLKCQE LLNYIMDTVK DSSNGAIYGA DCSNILLKDI L SVRKYWCE ...文字列: MDYKDDDDKH MSLVLNDLLI CCRQLEHDRA TERKKEVEKF KRLIRDPETI KHLDRHSDSK QGKYLNWDAV FRFLQKYIQK ETECLRIAK PNVSASTQAS RQKKMQEISS LVKYFIKCAN RRAPRLKCQE LLNYIMDTVK DSSNGAIYGA DCSNILLKDI L SVRKYWCE ISQQQWLELF SVYFRLYLKP SQDVHRVLVA RIIHAVTKGC CSQTDGLNSK FLDFFSKAIQ CARQEKSSSG LN HILAALT IFLKTLAVNF RIRVCELGDE ILPTLLYIWT QHRLNDSLKE VIIELFQLQI YIHHPKGAKT QEKGAYESTK WRS ILYNLY DLLVNEISHI GSRGKYSSGF RNIAVKENLI ELMADICHQV FNEDTRSLEI SQSYTTTQRE SSDYSVPCKR KKIE LGWEV IKDHLQKSQN DFDLVPWLQI ATQLISKYPA SLPNCELSPL LMILSQLLPQ QRHGERTPYV LRCLTEVALC QDKRS NLES SQKSDLLKLW NKIWCITFRG ISSEQIQAEN FGLLGAIIQG SLVEVDREFW KLFTGSACRP SCPAVCCLTL ALTTSI VPG TVKMGIEQNM CEVNRSFSLK ESIMKWLLFY QLEGDLENST EVPPILHSNF PHLVLEKILV SLTMKNCKAA MNFFQSV PE CEHHQKDKEE LSFSEVEELF LQTTFDKMDF LTIVRECGIE KHQSSIGFSV HQNLKESLDR CLLGLSEQLL NNYSSEIT N SETLVRCSRL LVGVLGCYCY MGVIAEEEAY KSELFQKAKS LMQCAGESIT LFKNKTNEEF RIGSLRNMMQ LCTRCLSNC TKKSPNKIAS GFFLRLLTSK LMNDIADICK SLASFIKKPF DRGEVESMED DTNGNLMEVE DQSSMNLFND YPDSSVSDAN EPGESQSTI GAINPLAEEY LSKQDLLFLD MLKFLCLCVT TAQTNTVSFR AADIRRKLLM LIDSSTLEPT KSLHLHMYLM L LKELPGEE YPLPMEDVLE LLKPLSNVCS LYRRDQDVCK TILNHVLHVV KNLGQSNMDS ENTRDAQGQF LTVIGAFWHL TK ERKYIFS VRMALVNCLK TLLEADPYSK WAILNVMGKD FPVNEVFTQF LADNHHQVRM LAAESINRLF QDTKGDSSRL LKA LPLKLQ QTAFENAYLK AQEGMREMSH SAENPETLDE IYNRKSVLLT LIAVVLSCSP ICEKQALFAL CKSVKENGLE PHLV KKVLE KVSETFGYRR LEDFMASHLD YLVLEWLNLQ DTEYNLSSFP FILLNYTNIE DFYRSCYKVL IPHLVIRSHF DEVKS IANQ IQEDWKSLLT DCFPKILVNI LPYFAYEGTR DSGMAQQRET ATKVYDMLKS ENLLGKQIDH LFISNLPEIV VELLMT LHE PANSSASQST DLCDFSGDLD PAPNPPHFPS HVIKATFAYI SNCHKTKLKS ILEILSKSPD SYQKILLAIC EQAAETN NV YKKHRILKIY HLFVSLLLKD IKSGLGGAWA FVLRDVIYTL IHYINQRPSC IMDVSLRSFS LCCDLLSQVC QTAVTYCK D ALENHLHVIV GTLIPLVYEQ VEVQKQVLDL LKYLVIDNKD NENLYITIKL LDPFPDHVVF KDLRITQQKI KYSRGPFSL LEEINHFLSV SVYDALPLTR LEGLKDLRRQ LELHKDQMVD IMRASQDNPQ DGIMVKLVVN LLQLSKMAIN HTGEKEVLEA VGSCLGEVG PIDFSTIAIQ HSKDASYTKA LKLFEDKELQ WTFIMLTYLN NTLVEDCVKV RSAAVTCLKN ILATKTGHSF W EIYKMTTD PMLAYLQPFR TSRKKFLEVP RFDKENPFEG LDDINLWIPL SENHDIWIKT LTCAFLDSGG TKCEILQLLK PM CEVKTDF CQTVLPYLIH DILLQDTNES WRNLLSTHVQ GFFTSCLRHF SQTSRSTTPA NLDSESEHFF RCCLDKKSQR TML AVVDYM RRQKRPSSGT IFNDAFWLDL NYLEVAKVAQ SCAAHFTALL YAEIYADKKS MDDQEKRSLA FEEGSQSTTI SSLS EKSKE ETGISLQDLL LEIYRSIGEP DSLYGCGGGK MLQPITRLRT YEHEAMWGKA LVTYDLETAI PSSTRQAGII QALQN LGLC HILSVYLKGL DYENKDWCPE LEELHYQAAW RNMQWDHCTS VSKEVEGTSY HESLYNALQS LRDREFSTFY ESLKYA RVK EVEEMCKRSL ESVYSLYPTL SRLQAIGELE SIGELFSRSV THRQLSEVYI KWQKHSQLLK DSDFSFQEPI MALRTVI LE ILMEKEMDNS QRECIKDILT KHLVELSILA RTFKNTQLPE RAIFQIKQYN SVSCGVSEWQ LEEAQVFWAK KEQSLALS I LKQMIKKLDA SCAANNPSLK LTYTECLRVC GNWLAETCLE NPAVIMQTYL EKAVEVAGNY DGESSDELRN GKMKAFLSL ARFSDTQYQR IENYMKSSEF ENKQALLKRA KEEVGLLREH KIQTNRYTVK VQRELELDEL ALRALKEDRK RFLCKAVENY INCLLSGEE HDMWVFRLCS LWLENSGVSE VNGMMKRDGM KIPTYKFLPL MYQLAARMGT KMMGGLGFHE VLNNLISRIS M DHPHHTLF IILALANANR DEFLTKPEVA RRSRITKNVP KQSSQLDEDR TEAANRIICT IRSRRPQMVR SVEALCDAYI IL ANLDATQ WKTQRKGINI PADQPITKLK NLEDVVVPTM EIKVDHTGEY GNLVTIQSFK AEFRLAGGVN LPKIIDCVGS DGK ERRQLV KGRDDLRQDA VMQQVFQMCN TLLQRNTETR KRKLTICTYK VVPLSQRSGV LEWCTGTVPI GEFLVNNEDG AHKR YRPND FSAFQCQKKM MEVQKKSFEE KYEVFMDVCQ NFQPVFRYFC MEKFLDPAIW FEKRLAYTRS VATSSIVGYI LGLGD RHVQ NILINEQSAE LVHIDLGVAF EQGKILPTPE TVPFRLTRDI VDGMGITGVE GVFRRCCEKT MEVMRNSQET LLTIVE VLL YDPLFDWTMN PLKALYLQQR PEDETELHPT LNADDQECKR NLSDIDQSFN KVAERVLMRL QEKLKGVEEG TVLSVGG QV NLLIQQAIDP KNLSRLFPGW KAWV UniProtKB: Serine-protein kinase ATM |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER / 詳細: 3 uL of sample/grid blotted for 12 s. | ||||||||||||||||||
| 詳細 | N-terminally FLAG-tagged ATM kinase was purified by affinity chromatography (anti-FLAG), overnight dialysis and gel-filtration. The sample was flash-frozen in liquid nitrogen until used for cryo-EM. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.15 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-20 / 撮影したグリッド数: 4 / 実像数: 2720 / 平均露光時間: 0.8 sec. / 平均電子線量: 2.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 35714 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 97902 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)