+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3504 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Substrate specificity in plant nitrilase helical assemblies is determined by their twist. | |||||||||
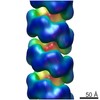
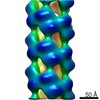
 マップデータ マップデータ | Lotus japonicus NITRILASE 4A filament | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Woodward JD / Trompetter I / Sewell BT / Piotrowski M | |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2018 ジャーナル: Commun Biol / 年: 2018タイトル: Substrate specificity of plant nitrilase complexes is affected by their helical twist. 著者: Jeremy D Woodward / Inga Trompetter / B Trevor Sewell / Markus Piotrowski /   要旨: Nitrilases are oligomeric, helix-forming enzymes from plants, fungi and bacteria that are involved in the metabolism of various natural and artificial nitriles. These biotechnologically important ...Nitrilases are oligomeric, helix-forming enzymes from plants, fungi and bacteria that are involved in the metabolism of various natural and artificial nitriles. These biotechnologically important enzymes are often specific for certain substrates, but directed attempts at modifying their substrate specificities by exchanging binding pocket residues have been largely unsuccessful. Thus, the basis for their selectivity is still unknown. Here we show, based on work with two highly similar nitrilases from the plant , that modifying nitrilase helical twist, either by exchanging an interface residue or by imposing a different twist, without altering any binding pocket residues, changes substrate preference. We reveal that helical twist and substrate size correlate and when binding pocket residues are exchanged between two nitrilases that show the same twist but different specificities, their specificities change. Based on these findings we propose that helical twist influences the overall size of the binding pocket. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3504.map.gz emd_3504.map.gz | 335.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3504-v30.xml emd-3504-v30.xml emd-3504.xml emd-3504.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3504.png emd_3504.png | 105.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3504 http://ftp.pdbj.org/pub/emdb/structures/EMD-3504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3504 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3504_validation.pdf.gz emd_3504_validation.pdf.gz | 217.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3504_full_validation.pdf.gz emd_3504_full_validation.pdf.gz | 216.2 KB | 表示 | |
| XML形式データ |  emd_3504_validation.xml.gz emd_3504_validation.xml.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3504 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3504 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3504.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3504.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Lotus japonicus NITRILASE 4A filament | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.22 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Lotus japonicus NITRILASE 4A filament
| 全体 | 名称: Lotus japonicus NITRILASE 4A filament |
|---|---|
| 要素 |
|
-超分子 #1: Lotus japonicus NITRILASE 4A filament
| 超分子 | 名称: Lotus japonicus NITRILASE 4A filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 24 kDa/nm |
-分子 #1: NITRILASE 4A
| 分子 | 名称: NITRILASE 4A / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: nitrilase |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTSNISLVTT PPPPEVDMGS DSNAPTTVRA TVVQASTIFY DTPATLDKAE RLLAEAAGSG SELVVFPEAF IGGYPRGSTF GMAVGNRTAK GREEFRKYHS SAIDVPGPEV DRLAAMAGKY KVHLVMGVIE RDGYTLYCSV LFFDSQGHYL GKHRKLMPTA MERVVWGFGD ...文字列: MTSNISLVTT PPPPEVDMGS DSNAPTTVRA TVVQASTIFY DTPATLDKAE RLLAEAAGSG SELVVFPEAF IGGYPRGSTF GMAVGNRTAK GREEFRKYHS SAIDVPGPEV DRLAAMAGKY KVHLVMGVIE RDGYTLYCSV LFFDSQGHYL GKHRKLMPTA MERVVWGFGD GSTIPVFETP VGKIGAVICW ENRMPLLRTA MYAKGVEIYC APTADAREVW QASMTHIALE GGCFVLSANQ FCRRKDYPPP PEYVFAGTEE DLTPDSVVCA GGSVIISPLG NVLAGPNYEG EALISADLDL GEIARAKFDF DVVGHYSRPE VLSLSVKDHP TNPVTFASTS TKVEDKTK |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate 詳細: The protein was allowed to adhere for 30 s, blotted, washed three-times with distilled water and stained with uranyl acetate, blotted again and allowed to dry at room temperature. | |||||||||
| グリッド | モデル: Grid-tech / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 20.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.02 kPa |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 30 / 平均露光時間: 1.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 0.5 µm / 最小 デフォーカス(補正後): 0.3 µm / 倍率(補正後): 50200 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1.2 mm / 最大 デフォーカス(公称値): 0.5 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: PHILIPS ROTATION HOLDER |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 使用したクラス数: 90 想定した対称性 - らせんパラメータ - Δz: 14.9 Å 想定した対称性 - らせんパラメータ - ΔΦ: -75 ° 想定した対称性 - らせんパラメータ - 軸対称性: D2 (2回x2回 2面回転対称) アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER (ver. 11) / ソフトウェア - 詳細: IHRSR / 使用した粒子像数: 1662 |
|---|---|
| Segment selection | 選択した数: 1662 / ソフトウェア - 名称: EMAN / ソフトウェア - 詳細: Boxer / 詳細: Picked using Eman Boxer in helix mode |
| 初期モデル | モデルのタイプ: INSILICO MODEL In silico モデル: Featureless cylinder approximating the diameter of the filament. |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: SPIDER (ver. 11) / ソフトウェア - 詳細: IHRSR |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















