+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3458 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | negative-stain volume of Sso DNA PolB1 | |||||||||
 マップデータ マップデータ | Final map from iteration 10 onto which FSC has been calculated and displayed (Figure S4C, right). Contour level in Chimera | |||||||||
 試料 試料 |
| |||||||||
| 生物種 | Archaea (unknown) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 22.8 Å | |||||||||
 データ登録者 データ登録者 | Abrescia NGA / Bell SD | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Identification and characterization of a heterotrimeric archaeal DNA polymerase holoenzyme. 著者: Jiangyu Yan / Thomas R Beattie / Adriana L Rojas / Kelly Schermerhorn / Tamzin Gristwood / Jonathan C Trinidad / Sonja V Albers / Pietro Roversi / Andrew F Gardner / Nicola G A Abrescia / Stephen D Bell /     要旨: Since their initial characterization over 30 years ago, it has been believed that the archaeal B-family DNA polymerases are single-subunit enzymes. This contrasts with the multi-subunit B-family ...Since their initial characterization over 30 years ago, it has been believed that the archaeal B-family DNA polymerases are single-subunit enzymes. This contrasts with the multi-subunit B-family replicative polymerases of eukaryotes. Here we reveal that the highly studied PolB1 from Sulfolobus solfataricus exists as a heterotrimeric complex in cell extracts. Two small subunits, PBP1 and PBP2, associate with distinct surfaces of the larger catalytic subunit and influence the enzymatic properties of the DNA polymerase. Thus, multi-subunit replicative DNA polymerase holoenzymes are present in all three domains of life. We reveal the architecture of the assembly by a combination of cross-linking coupled with mass spectrometry, X-ray crystallography and single-particle electron microscopy. The small subunits stabilize the holoenzyme assembly and the acidic tail of one small subunit mitigates the ability of the enzyme to perform strand-displacement synthesis, with important implications for lagging strand DNA synthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3458.map.gz emd_3458.map.gz | 6.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3458-v30.xml emd-3458-v30.xml emd-3458.xml emd-3458.xml | 21.5 KB 21.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3458.png emd_3458.png | 111.1 KB | ||
| マスクデータ |  emd_3458_msk_1.map emd_3458_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_3458_additional.map.gz emd_3458_additional.map.gz emd_3458_half_map_1.map.gz emd_3458_half_map_1.map.gz emd_3458_half_map_2.map.gz emd_3458_half_map_2.map.gz | 874.8 KB 6.1 MB 6.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3458 http://ftp.pdbj.org/pub/emdb/structures/EMD-3458 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3458 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3458 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3458_validation.pdf.gz emd_3458_validation.pdf.gz | 303.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3458_full_validation.pdf.gz emd_3458_full_validation.pdf.gz | 303.1 KB | 表示 | |
| XML形式データ |  emd_3458_validation.xml.gz emd_3458_validation.xml.gz | 8.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3458 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3458 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3458 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3458 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3458.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3458.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final map from iteration 10 onto which FSC has been calculated and displayed (Figure S4C, right). Contour level in Chimera | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.66 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_3458_msk_1.map emd_3458_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
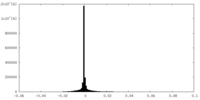
| 密度ヒストグラム |
-追加マップ: This is the masked map used in Figure...
| ファイル | emd_3458_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the masked map used in Figure 4A and in submitted figure. Contour level in Chimera. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
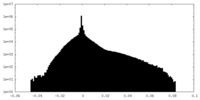
| 密度ヒストグラム |
-ハーフマップ: half2 map from iteration 10. Contour level in Chimera
| ファイル | emd_3458_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half2 map from iteration 10. Contour level in Chimera | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half1 map from iteration 10. Contour level in Chimera
| ファイル | emd_3458_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half1 map from iteration 10. Contour level in Chimera | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : archaeal DNA polymerase B1
| 全体 | 名称: archaeal DNA polymerase B1 |
|---|---|
| 要素 |
|
-超分子 #1: archaeal DNA polymerase B1
| 超分子 | 名称: archaeal DNA polymerase B1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: This is the apo enzyme. |
|---|---|
| 由来(天然) | 生物種: Archaea (unknown) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 101 KDa |
-分子 #1: DNA polymerase B1
| 分子 | 名称: DNA polymerase B1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: Archaea (unknown) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKQLTLFDI PSSKPAKSEQ NTQQSQQSAP VEEKKVVRRE WLEEAQENKI YFLLQVDYDG KKGKAVCKLF DKETQKIYAL YDNTGHKPYF LVDLEPDKVG KIPKIVRDPS FDHIETVSKI DPYTWNKFKL TKIVVRDPLA VRRLRNDVPK AYEAHIKYFN NYMYDIGLIP ...文字列: MAKQLTLFDI PSSKPAKSEQ NTQQSQQSAP VEEKKVVRRE WLEEAQENKI YFLLQVDYDG KKGKAVCKLF DKETQKIYAL YDNTGHKPYF LVDLEPDKVG KIPKIVRDPS FDHIETVSKI DPYTWNKFKL TKIVVRDPLA VRRLRNDVPK AYEAHIKYFN NYMYDIGLIP GMPYVVKNGK LESVYLSLDE KDVEEIKKAF ADSDEMTRQM AVDWLPIFET EIPKIKRVAI DIEVYTPVKG RIPDSQKAEF PIISIALAGS DGLKKVLVLN RNDVNEGSVK LDGISVERFN TEYELLGRFF DILLEYPIVL TFNGDDFDLP YIYFRALKLG YFPEEIPIDV AGKDEAKYLA GLHIDLYKFF FNKAVRNYAF EGKYNEYNLD AVAKALLGTS KVKVDTLISF LDVEKLIEYN FRDAEITLQL TTFNNDLTMK LIVLFSRISR LGIEELTRTE ISTWVKNLYY WEHRKRNWLI PLKEEILAKS SNIRTSALIK GKGYKGAVVI DPPAGIFFNI TVLDFASLYP SIIRTWNLSY ETVDIQQCKK PYEVKDETGE VLHIVCMDRP GITAVITGLL RDFRVKIYKK KAKNPNNSEE QKLLYDVVQR AMKVFINATY GVFGAETFPL YAPAVAESVT ALGRYVITST VKKAREEGLT VLYGDTDSLF LLNPPKNSLE NIIKWVKTTF NLDLEVDKTY KFVAFSGLKK NYFGVYQDGK VDIKGMLVKK RNTPEFVKKV FNEVKELMIS INSPNDVKEI KRKIVDVVKG SYEKLKNKGY NLDELAFKVM LSKPLDAYKK NTPQHVKAAL QLRPFGVNVL PRDIIYYVKV RSKDGVKPVQ LAKVTEIDAE KYLEALRSTF EQILRAFGVS WDEIAATMSI DSFFSYPSKG NS |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate 詳細: Negatively stained EM specimens were prepared using carbon-coated grids and stained with 2% of uranyl formate solution. | ||||||||||||
| グリッド | モデル: EMS / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.02 kPa |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FSC |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 108 / 平均露光時間: 0.5 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 90201 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.7 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)