+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3294 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Sub-tomogram averaging of Lassa virus glycoprotein spike from virus-like particles at pH 3 | |||||||||
 マップデータ マップデータ | Sub-tomogram average of the glycoprotein spike trimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | lassa virus / membrane protein / glycoprotein / receptor binding / membrane fusion | |||||||||
| 生物種 |  Lassa virus (ウイルス) Lassa virus (ウイルス) | |||||||||
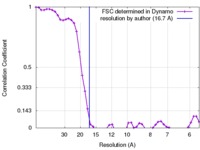
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 16.7 Å | |||||||||
 データ登録者 データ登録者 | Li S / Zhaoyang S / Pryce R / Parsy M-L / Fehling SK / Schlie K / Siebert CA / Garten W / Bowden TA / Strecker T / Huiskonen JT | |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2016 ジャーナル: PLoS Pathog / 年: 2016タイトル: Acidic pH-Induced Conformations and LAMP1 Binding of the Lassa Virus Glycoprotein Spike. 著者: Sai Li / Zhaoyang Sun / Rhys Pryce / Marie-Laure Parsy / Sarah K Fehling / Katrin Schlie / C Alistair Siebert / Wolfgang Garten / Thomas A Bowden / Thomas Strecker / Juha T Huiskonen /   要旨: Lassa virus is an enveloped, bi-segmented RNA virus and the most prevalent and fatal of all Old World arenaviruses. Virus entry into the host cell is mediated by a tripartite surface spike complex, ...Lassa virus is an enveloped, bi-segmented RNA virus and the most prevalent and fatal of all Old World arenaviruses. Virus entry into the host cell is mediated by a tripartite surface spike complex, which is composed of two viral glycoprotein subunits, GP1 and GP2, and the stable signal peptide. Of these, GP1 binds to cellular receptors and GP2 catalyzes fusion between the viral envelope and the host cell membrane during endocytosis. The molecular structure of the spike and conformational rearrangements induced by low pH, prior to fusion, remain poorly understood. Here, we analyzed the three-dimensional ultrastructure of Lassa virus using electron cryotomography. Sub-tomogram averaging yielded a structure of the glycoprotein spike at 14-Å resolution. The spikes are trimeric, cover the virion envelope, and connect to the underlying matrix. Structural changes to the spike, following acidification, support a viral entry mechanism dependent on binding to the lysosome-resident receptor LAMP1 and further dissociation of the membrane-distal GP1 subunits. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3294.map.gz emd_3294.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3294-v30.xml emd-3294-v30.xml emd-3294.xml emd-3294.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3294_fsc.xml emd_3294_fsc.xml | 4.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  EMD-3294.tif EMD-3294.tif emd_3294.tif emd_3294.tif | 1.1 MB 1.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3294 http://ftp.pdbj.org/pub/emdb/structures/EMD-3294 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3294 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3294 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3294_validation.pdf.gz emd_3294_validation.pdf.gz | 252.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3294_full_validation.pdf.gz emd_3294_full_validation.pdf.gz | 251.8 KB | 表示 | |
| XML形式データ |  emd_3294_validation.xml.gz emd_3294_validation.xml.gz | 8.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3294 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3294 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3294 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3294 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3294.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3294.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-tomogram average of the glycoprotein spike trimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Purified Lassa virus VLPs at pH 3
| 全体 | 名称: Purified Lassa virus VLPs at pH 3 |
|---|---|
| 要素 |
|
-超分子 #1000: Purified Lassa virus VLPs at pH 3
| 超分子 | 名称: Purified Lassa virus VLPs at pH 3 / タイプ: sample / ID: 1000 / 詳細: Unfixed virus-like particles / Number unique components: 1 |
|---|
-超分子 #1: Lassa virus
| 超分子 | 名称: Lassa virus / タイプ: virus / ID: 1 / Name.synonym: Lassa mammarenavirus / NCBI-ID: 11620 / 生物種: Lassa virus / Sci species strain: Josiah / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No / Syn species name: Lassa mammarenavirus |
|---|---|
| 宿主 | 生物種:  Mastomys (ネズミ) / 別称: VERTEBRATES Mastomys (ネズミ) / 別称: VERTEBRATES |
| Host system | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 3 詳細: 50 mM buffer of succinic acid, dihydrogen phosphate and glycine (2:7:7) |
|---|---|
| グリッド | 詳細: Grids (Cflat CF-2/1-2C-T) were glow-discharged for 15 s. 6-nm gold particles were added. |
| 凍結 | 凍結剤: ETHANE-PROPANE MIXTURE / チャンバー内湿度: 80 % / チャンバー内温度: 120 K / 装置: GATAN CRYOPLUNGE 3 / 手法: Blot for 3 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 80 K / 最高: 120 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 160,000 times magnification. |
| 特殊光学系 | エネルギーフィルター - 名称: GIF QUANTUM LS エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 詳細 | Super-resolution counting mode |
| 日付 | 2014年12月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - サンプリング間隔: 5 µm / 実像数: 10 / 平均電子線量: 60 e/Å2 詳細: Each image is a tilt series of 19 movies, acquired at 5 degree intervals. Each movie consists of 8 frames. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 37037 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.9 µm / 最小 デフォーカス(公称値): 3.0 µm / 倍率(公称値): 160000 |
| 試料ステージ | 試料ホルダー: Liquid nitrogen cooled / 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -45 ° / Tilt series - Axis1 - Max angle: 45 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)