+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30284 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | COVID-19 RNA-dependent RNA polymerase pre-translocated catalytic complex in conformation II | ||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 生物種 |    Foot-and-mouth disease virus (口蹄疫ウイルス) Foot-and-mouth disease virus (口蹄疫ウイルス) | ||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.12 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Wang Q / Gao Y / Ji W / Mu A / Rao Z | ||||||||||||||||||||||||
| 資金援助 |  中国, 7件 中国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: Structural Basis for RNA Replication by the SARS-CoV-2 Polymerase. 著者: Quan Wang / Jiqin Wu / Haofeng Wang / Yan Gao / Qiaojie Liu / An Mu / Wenxin Ji / Liming Yan / Yan Zhu / Chen Zhu / Xiang Fang / Xiaobao Yang / Yucen Huang / Hailong Gao / Fengjiang Liu / Ji ...著者: Quan Wang / Jiqin Wu / Haofeng Wang / Yan Gao / Qiaojie Liu / An Mu / Wenxin Ji / Liming Yan / Yan Zhu / Chen Zhu / Xiang Fang / Xiaobao Yang / Yucen Huang / Hailong Gao / Fengjiang Liu / Ji Ge / Qianqian Sun / Xiuna Yang / Wenqing Xu / Zhijie Liu / Haitao Yang / Zhiyong Lou / Biao Jiang / Luke W Guddat / Peng Gong / Zihe Rao /   要旨: Nucleotide analog inhibitors, including broad-spectrum remdesivir and favipiravir, have shown promise in in vitro assays and some clinical studies for COVID-19 treatment, this despite an incomplete ...Nucleotide analog inhibitors, including broad-spectrum remdesivir and favipiravir, have shown promise in in vitro assays and some clinical studies for COVID-19 treatment, this despite an incomplete mechanistic understanding of the viral RNA-dependent RNA polymerase nsp12 drug interactions. Here, we examine the molecular basis of SARS-CoV-2 RNA replication by determining the cryo-EM structures of the stalled pre- and post- translocated polymerase complexes. Compared with the apo complex, the structures show notable structural rearrangements happening to nsp12 and its co-factors nsp7 and nsp8 to accommodate the nucleic acid, whereas there are highly conserved residues in nsp12, positioning the template and primer for an in-line attack on the incoming nucleotide. Furthermore, we investigate the inhibition mechanism of the triphosphate metabolite of remdesivir through structural and kinetic analyses. A transition model from the nsp7-nsp8 hexadecameric primase complex to the nsp12-nsp7-nsp8 polymerase complex is also proposed to provide clues for the understanding of the coronavirus transcription and replication machinery. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30284.map.gz emd_30284.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30284-v30.xml emd-30284-v30.xml emd-30284.xml emd-30284.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_30284.png emd_30284.png | 95.3 KB | ||
| マスクデータ |  emd_30284_msk_1.map emd_30284_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_30284_half_map_1.map.gz emd_30284_half_map_1.map.gz emd_30284_half_map_2.map.gz emd_30284_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30284 http://ftp.pdbj.org/pub/emdb/structures/EMD-30284 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30284 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30284 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30284_validation.pdf.gz emd_30284_validation.pdf.gz | 79.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30284_full_validation.pdf.gz emd_30284_full_validation.pdf.gz | 78.6 KB | 表示 | |
| XML形式データ |  emd_30284_validation.xml.gz emd_30284_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30284 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30284 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30284 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30284 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30284.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30284.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_30284_msk_1.map emd_30284_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
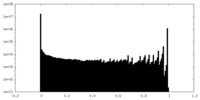
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_30284_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
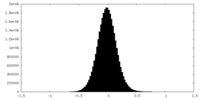
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_30284_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
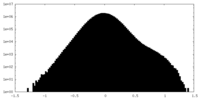
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : COVID-19 RNA-dependent RNA polymerase pre-translocated catalytic ...
| 全体 | 名称: COVID-19 RNA-dependent RNA polymerase pre-translocated catalytic complex in conformation II |
|---|---|
| 要素 |
|
-超分子 #1: COVID-19 RNA-dependent RNA polymerase pre-translocated catalytic ...
| 超分子 | 名称: COVID-19 RNA-dependent RNA polymerase pre-translocated catalytic complex in conformation II タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: full-length COVID-19 nsp12 (residues S1-Q932) was incubated with nsp7 (residues S1-Q83) and nsp8 (A1-Q198), and in complex with RNA template and product. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #1: RNA-directed RNA polymerase
| 分子 | 名称: RNA-directed RNA polymerase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSADAQSFL NRVCGVSAAR LTPCGTGTST DVVYRAFDIY NDKVAGFAKF LKTNCCRFQE KDEDDNLIDS YFVVKRHTFS NYQHEETIY NLLKDCPAVA KHDFFKFRID GDMVPHISRQ RLTKYTMADL VYALRHFDEG NCDTLKEILV TYNCCDDDYF N KKDWYDFV ...文字列: MGSADAQSFL NRVCGVSAAR LTPCGTGTST DVVYRAFDIY NDKVAGFAKF LKTNCCRFQE KDEDDNLIDS YFVVKRHTFS NYQHEETIY NLLKDCPAVA KHDFFKFRID GDMVPHISRQ RLTKYTMADL VYALRHFDEG NCDTLKEILV TYNCCDDDYF N KKDWYDFV ENPDILRVYA NLGERVRQAL LKTVQFCDAM RNAGIVGVLT LDNQDLNGNW YDFGDFIQTT PGSGVPVVDS YY SLLMPIL TLTRALTAES HVDTDLTKPY IKWDLLKYDF TEERLKLFDR YFKYWDQTYH PNCVNCLDDR CILHCANFNV LFS TVFPPT SFGPLVRKIF VDGVPFVVST GYHFRELGVV HNQDVNLHSS RLSFKELLVY AADPAMHAAS GNLLLDKRTT CFSV AALTN NVAFQTVKPG NFNKDFYDFA VSKGFFKEGS SVELKHFFFA QDGNAAISDY DYYRYNLPTM CDIRQLLFVV EVVDK YFDC YDGGCINANQ VIVNNLDKSA GFPFNKWGKA RLYYDSMSYE DQDALFAYTK RNVIPTITQM NLKYAISAKN RARTVA GVS ICSTMTNRQF HQKLLKSIAA TRGATVVIGT SKFYGGWHNM LKTVYSDVEN PHLMGWDYPK CDRAMPNMLR IMASLVL AR KHTTCCSLSH RFYRLANECA QVLSEMVMCG GSLYVKPGGT SSGDATTAYA NSVFNICQAV TANVNALLST DGNKIADK Y VRNLQHRLYE CLYRNRDVDT DFVNEFYAYL RKHFSMMILS DDAVVCFNST YASQGLVASI KNFKSVLYYQ NNVFMSEAK CWTETDLTKG PHEFCSQHTM LVKQGDDYVY LPYPDPSRIL GAGCFVDDIV KTDGTLMIER FVSLAIDAYP LTKHPNQEYA DVFHLYLQY IRKLHDELTG HMLDMYSVML TNDNTSRYWE PEFYEAMYTP HTVLQHHHHH HHHHH |
-分子 #2: Non-structural protein 8
| 分子 | 名称: Non-structural protein 8 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPAIASEFSS LPSYAAFATA QEAYEQAVAN GDSEVVLKKL KKSLNVAKSE FDRDAAMQRK LEKMADQAMT QMYKQARSED KRAKVTSAM QTMLFTMLRK LDNDALNNII NNARDGCVPL NIIPLTTAAK LMVVIPDYNT YKNTCDGTTF TYASALWEIQ Q VVDADSKI ...文字列: GPAIASEFSS LPSYAAFATA QEAYEQAVAN GDSEVVLKKL KKSLNVAKSE FDRDAAMQRK LEKMADQAMT QMYKQARSED KRAKVTSAM QTMLFTMLRK LDNDALNNII NNARDGCVPL NIIPLTTAAK LMVVIPDYNT YKNTCDGTTF TYASALWEIQ Q VVDADSKI VQLSEISMDN SPNLAWPLIV TALRANSAVK LQ |
-分子 #3: Non-structural protein 7
| 分子 | 名称: Non-structural protein 7 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPSKMSDVKC TSVVLLSVLQ QLRVESSSKL WAQCVQLHND ILLAKDTTEA FEKMVSLLSV LLSMQGAVDI NKLCEEMLDN RATLQ |
-分子 #4: RNA (5'-R(P*GP*GP*GP*AP*GP*CP*UP*CP*CP*UP*CP*CP*UP*GP*UP*GP*UP*CP...
| 分子 | 名称: RNA (5'-R(P*GP*GP*GP*AP*GP*CP*UP*CP*CP*UP*CP*CP*UP*GP*UP*GP*UP*CP*GP*U)-3') タイプ: rna / ID: 4 |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus (口蹄疫ウイルス) Foot-and-mouth disease virus (口蹄疫ウイルス) |
| 配列 | 文字列: GGGAGAUGUC UCCUCCUGUG UCGUCGAAA |
-分子 #5: RNA (5'-R(P*AP*CP*GP*AP*CP*AP*CP*AP*GP*G*(GS4)P*G)-3')
| 分子 | 名称: RNA (5'-R(P*AP*CP*GP*AP*CP*AP*CP*AP*GP*G*(GS4)P*G)-3') タイプ: rna / ID: 5 |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus (口蹄疫ウイルス) Foot-and-mouth disease virus (口蹄疫ウイルス) |
| 配列 | 文字列: UGUUCGACGA CACAGG(GS4)G |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) #0 - 検出モード: SUPER-RESOLUTION / #0 - 平均電子線量: 60.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) #1 - 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)