+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | mRNA decoding in human is kinetically and structurally distinct from bacteria (80S consensus refined structure) | |||||||||
 マップデータ マップデータ | Unsharpened refine3D map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.1 Å | |||||||||
 データ登録者 データ登録者 | Holm M / Natchiar KS / Rundlet EJ / Myasnikov AG / Altman RB / Blanchard SC | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: mRNA decoding in human is kinetically and structurally distinct from bacteria. 著者: Mikael Holm / S Kundhavai Natchiar / Emily J Rundlet / Alexander G Myasnikov / Zoe L Watson / Roger B Altman / Hao-Yuan Wang / Jack Taunton / Scott C Blanchard /   要旨: In all species, ribosomes synthesize proteins by faithfully decoding messenger RNA (mRNA) nucleotide sequences using aminoacyl-tRNA substrates. Current knowledge of the decoding mechanism derives ...In all species, ribosomes synthesize proteins by faithfully decoding messenger RNA (mRNA) nucleotide sequences using aminoacyl-tRNA substrates. Current knowledge of the decoding mechanism derives principally from studies on bacterial systems. Although key features are conserved across evolution, eukaryotes achieve higher-fidelity mRNA decoding than bacteria. In human, changes in decoding fidelity are linked to ageing and disease and represent a potential point of therapeutic intervention in both viral and cancer treatment. Here we combine single-molecule imaging and cryogenic electron microscopy methods to examine the molecular basis of human ribosome fidelity to reveal that the decoding mechanism is both kinetically and structurally distinct from that of bacteria. Although decoding is globally analogous in both species, the reaction coordinate of aminoacyl-tRNA movement is altered on the human ribosome and the process is an order of magnitude slower. These distinctions arise from eukaryote-specific structural elements in the human ribosome and in the elongation factor eukaryotic elongation factor 1A (eEF1A) that together coordinate faithful tRNA incorporation at each mRNA codon. The distinct nature and timing of conformational changes within the ribosome and eEF1A rationalize how increased decoding fidelity is achieved and potentially regulated in eukaryotic species. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29782.map.gz emd_29782.map.gz | 2.2 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29782-v30.xml emd-29782-v30.xml emd-29782.xml emd-29782.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29782_fsc.xml emd_29782_fsc.xml | 31.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29782.png emd_29782.png | 157.6 KB | ||
| その他 |  emd_29782_additional_1.map.gz emd_29782_additional_1.map.gz emd_29782_additional_2.map.gz emd_29782_additional_2.map.gz emd_29782_additional_3.map.gz emd_29782_additional_3.map.gz emd_29782_half_map_1.map.gz emd_29782_half_map_1.map.gz emd_29782_half_map_2.map.gz emd_29782_half_map_2.map.gz | 220.8 MB 2.5 GB 1.5 GB 2.4 GB 2.4 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29782 http://ftp.pdbj.org/pub/emdb/structures/EMD-29782 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29782 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29782 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29782_validation.pdf.gz emd_29782_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29782_full_validation.pdf.gz emd_29782_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_29782_validation.xml.gz emd_29782_validation.xml.gz | 39.9 KB | 表示 | |
| CIF形式データ |  emd_29782_validation.cif.gz emd_29782_validation.cif.gz | 54 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29782 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29782 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29782 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29782 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29782.map.gz / 形式: CCP4 / 大きさ: 2.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29782.map.gz / 形式: CCP4 / 大きさ: 2.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened refine3D map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.6472 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Post-processed masked map
| ファイル | emd_29782_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed masked map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Post-processed map
| ファイル | emd_29782_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Post-processed locally filtered map
| ファイル | emd_29782_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed locally filtered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_29782_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_29782_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human ribosome
| 全体 | 名称: Human ribosome |
|---|---|
| 要素 |
|
-超分子 #1: Human ribosome
| 超分子 | 名称: Human ribosome / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 79.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -1.5 µm / 最小 デフォーカス(公称値): -0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 434579 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| FSC曲線 (解像度の算出) | 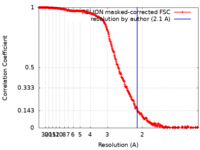 |
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)