+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Homocitrate-deficient nitrogenase MoFe-protein from A. vinelandii nifV knockout in complex with NafT | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | nitrogenase / nitrogen fixation / reductase / MoFe / OXIDOREDUCTASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報molybdenum-iron nitrogenase complex / nitrogenase / nitrogenase activity / nitrogen fixation / iron-sulfur cluster binding / ATP binding / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) | ||||||||||||
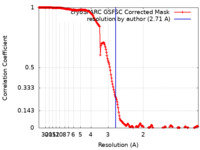
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.71 Å | ||||||||||||
 データ登録者 データ登録者 | Warmack RA / Maggiolo AO / Rees DC | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural consequences of turnover-induced homocitrate loss in nitrogenase. 著者: Rebeccah A Warmack / Ailiena O Maggiolo / Andres Orta / Belinda B Wenke / James B Howard / Douglas C Rees /  要旨: Nitrogenase catalyzes the ATP-dependent reduction of dinitrogen to ammonia during the process of biological nitrogen fixation that is essential for sustaining life. The active site FeMo-cofactor ...Nitrogenase catalyzes the ATP-dependent reduction of dinitrogen to ammonia during the process of biological nitrogen fixation that is essential for sustaining life. The active site FeMo-cofactor contains a [7Fe:1Mo:9S:1C] metallocluster coordinated with an R-homocitrate (HCA) molecule. Here, we establish through single particle cryoEM and chemical analysis of two forms of the Azotobacter vinelandii MoFe-protein - a high pH turnover inactivated species and a ∆NifV variant that cannot synthesize HCA - that loss of HCA is coupled to α-subunit domain and FeMo-cofactor disordering, and formation of a histidine coordination site. We further find a population of the ∆NifV variant complexed to an endogenous protein identified through structural and proteomic approaches as the uncharacterized protein NafT. Recognition by endogenous NafT demonstrates the physiological relevance of the HCA-compromised form, perhaps for cofactor insertion or repair. Our results point towards a dynamic active site in which HCA plays a role in enabling nitrogenase catalysis by facilitating activation of the FeMo-cofactor from a relatively stable form to a state capable of reducing dinitrogen under ambient conditions. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28275.map.gz emd_28275.map.gz | 372 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28275-v30.xml emd-28275-v30.xml emd-28275.xml emd-28275.xml | 23.9 KB 23.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28275_fsc.xml emd_28275_fsc.xml | 19.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28275.png emd_28275.png | 59.9 KB | ||
| Filedesc metadata |  emd-28275.cif.gz emd-28275.cif.gz | 6.9 KB | ||
| その他 |  emd_28275_additional_1.map.gz emd_28275_additional_1.map.gz emd_28275_half_map_1.map.gz emd_28275_half_map_1.map.gz emd_28275_half_map_2.map.gz emd_28275_half_map_2.map.gz | 370.7 MB 675.7 MB 675.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28275 http://ftp.pdbj.org/pub/emdb/structures/EMD-28275 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28275 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28275 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28275_validation.pdf.gz emd_28275_validation.pdf.gz | 1021.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28275_full_validation.pdf.gz emd_28275_full_validation.pdf.gz | 1021 KB | 表示 | |
| XML形式データ |  emd_28275_validation.xml.gz emd_28275_validation.xml.gz | 28.5 KB | 表示 | |
| CIF形式データ |  emd_28275_validation.cif.gz emd_28275_validation.cif.gz | 37.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28275 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28275 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28275 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28275 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28275.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28275.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
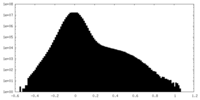
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Blurred version of the main map.
| ファイル | emd_28275_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Blurred version of the main map. | ||||||||||||
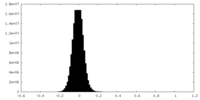
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28275_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
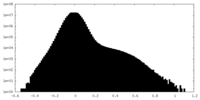
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28275_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Heterotetrameric nitrogenase MoFe-protein in complex with NafT is...
| 全体 | 名称: Heterotetrameric nitrogenase MoFe-protein in complex with NafT isolated from nifV mutant strain |
|---|---|
| 要素 |
|
-超分子 #1: Heterotetrameric nitrogenase MoFe-protein in complex with NafT is...
| 超分子 | 名称: Heterotetrameric nitrogenase MoFe-protein in complex with NafT isolated from nifV mutant strain タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii DJ (窒素固定) / 株: DJ605 Azotobacter vinelandii DJ (窒素固定) / 株: DJ605 |
-分子 #1: Nitrogenase molybdenum-iron protein alpha chain
| 分子 | 名称: Nitrogenase molybdenum-iron protein alpha chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: nitrogenase |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii DJ (窒素固定) / 株: DJ / ATCC BAA-1303 Azotobacter vinelandii DJ (窒素固定) / 株: DJ / ATCC BAA-1303 |
| 分子量 | 理論値: 54.000559 KDa |
| 配列 | 文字列: MSREEVESLI QEVLEVYPEK ARKDRNKHLA VNDPAVTQSK KCIISNKKSQ PGLMTIRGCA YAGSKGVVWG PIKDMIHISH GPVGCGQYS RAGRRNYYIG TTGVNAFVTM NFTSDFQEKD IVFGGDKKLA KLIDEVETLF PLNKGISVQS ECPIGLIGDD I ESVSKVKG ...文字列: MSREEVESLI QEVLEVYPEK ARKDRNKHLA VNDPAVTQSK KCIISNKKSQ PGLMTIRGCA YAGSKGVVWG PIKDMIHISH GPVGCGQYS RAGRRNYYIG TTGVNAFVTM NFTSDFQEKD IVFGGDKKLA KLIDEVETLF PLNKGISVQS ECPIGLIGDD I ESVSKVKG AELSKTIVPV RCEGFRGVSQ SLGHHIANDA VRDWVLGKRD EDTTFASTPY DVAIIGDYNI GGDAWSSRIL LE EMGLRCV AQWSGDGSIS EIELTPKVKL NLVHCYRSMN YISRHMEEKY GIPWMEYNFF GPTKTIESLR AIAAKFDESI QKK CEEVIA KYKPEWEAVV AKYRPRLEGK RVMLYIGGLR PRHVIGAYED LGMEVVGTGY EFAHNDDYDR TMKEMGDSTL LYDD VTGYE FEEFVKRIKP DLIGSGIKEK FIFQKMGIPF REMHSWDYSG PYHGFDGFAI FARDMDMTLN NPCWKKLQAP WE UniProtKB: Nitrogenase molybdenum-iron protein alpha chain |
-分子 #2: Nitrogenase molybdenum-iron protein beta chain
| 分子 | 名称: Nitrogenase molybdenum-iron protein beta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO / EC番号: nitrogenase |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii DJ (窒素固定) / 株: DJ / ATCC BAA-1303 Azotobacter vinelandii DJ (窒素固定) / 株: DJ / ATCC BAA-1303 |
| 分子量 | 理論値: 59.404684 KDa |
| 配列 | 文字列: SQQVDKIKAS YPLFLDQDYK DMLAKKRDGF EEKYPQDKID EVFQWTTTKE YQELNFQREA LTVNPAKACQ PLGAVLCALG FEKTMPYVH GSQGCVAYFR SYFNRHFREP VSCVSDSMTE DAAVFGGQQN MKDGLQNCKA TYKPDMIAVS TTCMAEVIGD D LNAFINNS ...文字列: SQQVDKIKAS YPLFLDQDYK DMLAKKRDGF EEKYPQDKID EVFQWTTTKE YQELNFQREA LTVNPAKACQ PLGAVLCALG FEKTMPYVH GSQGCVAYFR SYFNRHFREP VSCVSDSMTE DAAVFGGQQN MKDGLQNCKA TYKPDMIAVS TTCMAEVIGD D LNAFINNS KKEGFIPDEF PVPFAHTPSF VGSHVTGWDN MFEGIARYFT LKSMDDKVVG SNKKINIVPG FETYLGNFRV IK RMLSEMG VGYSLLSDPE EVLDTPADGQ FRMYAGGTTQ EEMKDAPNAL NTVLLQPWHL EKTKKFVEGT WKHEVPKLNI PMG LDWTDE FLMKVSEISG QPIPASLTKE RGRLVDMMTD SHTWLHGKRF ALWGDPDFVM GLVKFLLELG CEPVHILCHN GNKR WKKAV DAILAASPYG KNATVYIGKD LWHLRSLVFT DKPDFMIGNS YGKFIQRDTL HKGKEFEVPL IRIGFPIFDR HHLHR STTL GYEGAMQILT TLVNSILERL DEETRGMQAT DYNHDLVR UniProtKB: Nitrogenase molybdenum-iron protein beta chain |
-分子 #3: nitrogenase-associated factor T
| 分子 | 名称: nitrogenase-associated factor T / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Azotobacter vinelandii DJ (窒素固定) / 株: DJ / ATCC BAA-1303 Azotobacter vinelandii DJ (窒素固定) / 株: DJ / ATCC BAA-1303 |
| 分子量 | 理論値: 14.847405 KDa |
| 配列 | 文字列: MSWRILLCHK HPVSARLRFL IPTGGGVVLP QTLPRLAVIA EDQEAPVQCH PASALRALQE TMALGWQLEL IGEFRLNMEV PGQIMPIYL AALAGHELPP PPEGTRWIEL TQSIGMPWLD RELLRRVYEE LIG UniProtKB: Uncharacterized protein |
-分子 #4: iron-sulfur-molybdenum cluster with interstitial carbon
| 分子 | 名称: iron-sulfur-molybdenum cluster with interstitial carbon タイプ: ligand / ID: 4 / コピー数: 2 / 式: ICS |
|---|---|
| 分子量 | 理論値: 787.451 Da |
| Chemical component information | 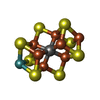 ChemComp-ICE: |
-分子 #5: CITRIC ACID
| 分子 | 名称: CITRIC ACID / タイプ: ligand / ID: 5 / コピー数: 1 / 式: CIT |
|---|---|
| 分子量 | 理論値: 192.124 Da |
| Chemical component information |  ChemComp-CIT: |
-分子 #6: FE(8)-S(7) CLUSTER
| 分子 | 名称: FE(8)-S(7) CLUSTER / タイプ: ligand / ID: 6 / コピー数: 2 / 式: CLF |
|---|---|
| 分子量 | 理論値: 671.215 Da |
| Chemical component information | 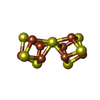 ChemComp-CLF: |
-分子 #7: CHAPSO
| 分子 | 名称: CHAPSO / タイプ: ligand / ID: 7 / コピー数: 2 / 式: 1N7 |
|---|---|
| 分子量 | 理論値: 631.884 Da |
| Chemical component information | 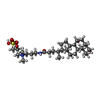 ChemComp-1N7: |
-分子 #8: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 8 / コピー数: 2 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.41 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)