+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | T4 Bacteriophage primosome with single strand DNA, state 1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | phage / complex / helicase / REPLICATION-DNA-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bidirectional double-stranded viral DNA replication / DNA replication, synthesis of primer / viral DNA genome replication / single-stranded DNA helicase activity / DNA helicase activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / DNA-directed RNA polymerase activity / DNA replication / hydrolase activity ...bidirectional double-stranded viral DNA replication / DNA replication, synthesis of primer / viral DNA genome replication / single-stranded DNA helicase activity / DNA helicase activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / DNA-directed RNA polymerase activity / DNA replication / hydrolase activity / DNA binding / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) | |||||||||
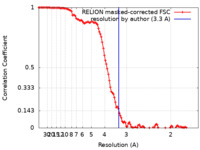
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Feng X / Li H | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 | ジャーナル: bioRxiv / 年: 2023 タイトル: Structural basis of the T4 bacteriophage primosome assembly and primer synthesis. 著者: Xiang Feng / Michelle M Spiering / Ruda de Luna Almeida Santos / Stephen J Benkovic / Huilin Li /  要旨: The T4 bacteriophage gp41 helicase and gp61 primase assemble into a primosome complex to couple DNA unwinding with RNA primer synthesis for DNA replication. How a primosome is assembled and how the ...The T4 bacteriophage gp41 helicase and gp61 primase assemble into a primosome complex to couple DNA unwinding with RNA primer synthesis for DNA replication. How a primosome is assembled and how the length of the RNA primer is defined in the T4 bacteriophage, or in any model system, are unclear. Here we report a series of cryo-EM structures of T4 primosome assembly intermediates at resolutions up to 2.7 Å. We show that the gp41 helicase is an open spiral in the absence of ssDNA, and ssDNA binding triggers a large-scale scissor-like conformational change that drives the open spiral to a closed ring that activates the helicase. We found that the activation of the gp41 helicase exposes a cryptic hydrophobic primase-binding surface allowing for the recruitment of the gp61 primase. The primase binds the gp41 helicase in a bipartite mode in which the N-terminal Zn-binding domain (ZBD) and the C-terminal RNA polymerase domain (RPD) each contain a helicase-interacting motif (HIM1 and HIM2, respectively) that bind to separate gp41 N-terminal hairpin dimers, leading to the assembly of one primase on the helicase hexamer. Based on two observed primosome conformations - one in a DNA-scanning mode and the other in a post RNA primer-synthesis mode - we suggest that the linker loop between the gp61 ZBD and RPD contributes to the T4 pentaribonucleotide primer. Our study reveals T4 primosome assembly process and sheds light on RNA primer synthesis mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27737.map.gz emd_27737.map.gz | 116.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27737-v30.xml emd-27737-v30.xml emd-27737.xml emd-27737.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27737_fsc.xml emd_27737_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27737.png emd_27737.png | 92.8 KB | ||
| その他 |  emd_27737_half_map_1.map.gz emd_27737_half_map_1.map.gz emd_27737_half_map_2.map.gz emd_27737_half_map_2.map.gz | 98.3 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27737 http://ftp.pdbj.org/pub/emdb/structures/EMD-27737 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27737 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27737 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27737_validation.pdf.gz emd_27737_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27737_full_validation.pdf.gz emd_27737_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_27737_validation.xml.gz emd_27737_validation.xml.gz | 18.3 KB | 表示 | |
| CIF形式データ |  emd_27737_validation.cif.gz emd_27737_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27737 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27737 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27737 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27737 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dvfMC  8dtpC  8dueC  8duoC  8dviC  8dw6C  8dwjC  8g0zC  8gaoC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27737.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27737.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_27737_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
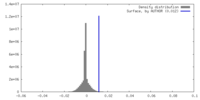
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27737_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
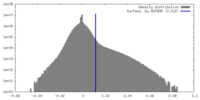
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of T4 bacteriophage primosome including helicase gp41, pr...
| 全体 | 名称: Complex of T4 bacteriophage primosome including helicase gp41, primase gp61 and ssDNA/RNA hybrid |
|---|---|
| 要素 |
|
-超分子 #1: Complex of T4 bacteriophage primosome including helicase gp41, pr...
| 超分子 | 名称: Complex of T4 bacteriophage primosome including helicase gp41, primase gp61 and ssDNA/RNA hybrid タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
-分子 #1: DnaB-like replicative helicase
| 分子 | 名称: DnaB-like replicative helicase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 分子量 | 理論値: 53.662848 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVEIILSHLI FDQAYFSKVW PYMDSEYFES GPAKNTFKLI KSHVNEYHSV PSINALNVAL ENSSFTETEY SGVKTLISKL ADSPEDHSW LVKETEKYVQ QRAMFNATSK IIEIQTNAEL PPEKRNKKMP DVGAIPDIMR QALSISFDSY VGHDWMDDYE A RWLSYMNK ...文字列: MVEIILSHLI FDQAYFSKVW PYMDSEYFES GPAKNTFKLI KSHVNEYHSV PSINALNVAL ENSSFTETEY SGVKTLISKL ADSPEDHSW LVKETEKYVQ QRAMFNATSK IIEIQTNAEL PPEKRNKKMP DVGAIPDIMR QALSISFDSY VGHDWMDDYE A RWLSYMNK ARKVPFKLRI LNKITKGGAE TGTLNVLMAG VNVGKSLGLC SLAADYLQLG HNVLYISMEM AEEVCAKRID AN MLDVSLD DIDDGHISYA EYKGKMEKWR EKSTLGRLIV KQYPTGGADA NTFRSLLNEL KLKKNFVPTI IIVDYLGICK SCR IRVYSE NSYTTVKAIA EELRALAVET ETVLWTAAQV GKQAWDSSDV NMSDIAESAG LPATADFMLA VIETEELAAA EQQL IKQIK SRYGDKNKWN KFLMGVQKGN QKWVEIEQDS TPTEVNEVAG SQQIQAEQNR YQRNESTRAQ LDALANELKF UniProtKB: DnaB-like replicative helicase |
-分子 #3: DNA primase
| 分子 | 名称: DNA primase / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: 転移酵素; リンを含む基を移すもの; 核酸を移すもの |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 分子量 | 理論値: 39.842043 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSIPWIDNE FAYRALAHLP KFTQVNNSST FKLRFRCPVC GDSKTDQNKA RGWYYGDNNE GNIHCYNCNY HAPIGIYLKE FEPDLYREY IFEIRKEKGK SRPIEKPKEL PKQPEKKIIK SLPSCVRLDK LAEDHPIIKY VKARCIPKDK WKYLWFTTEW P KLVNSIAP ...文字列: MSSIPWIDNE FAYRALAHLP KFTQVNNSST FKLRFRCPVC GDSKTDQNKA RGWYYGDNNE GNIHCYNCNY HAPIGIYLKE FEPDLYREY IFEIRKEKGK SRPIEKPKEL PKQPEKKIIK SLPSCVRLDK LAEDHPIIKY VKARCIPKDK WKYLWFTTEW P KLVNSIAP GTYKKEISEP RLVIPIYNAN GKAESFQGRA LKKDAPQKYI TIEAYPEATK IYGVERVKDG DVYVLEGPID SL FIENGIA ITGGQLDLEV VPFKDRRVWV LDNEPRHPDT IKRMTKLVDA GERVMFWDKS PWKSKDVNDM IRKEGATPEQ IME YMKNNI AQGLMAKMRL SKYAKI UniProtKB: DNA primase |
-分子 #2: DNA (5'-D(P*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*T)-3')
| 分子 | 名称: DNA (5'-D(P*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*T)-3') / タイプ: dna / ID: 2 詳細: The observed DNA sequence is an average of different nucleotides because the protein lacks sequence specificity. Thus, each base was modeled as a T residue. コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 分子量 | 理論値: 3.605356 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT) |
-分子 #4: DNA (5'-D(P*GP*GP*CP*TP*G)-3')
| 分子 | 名称: DNA (5'-D(P*GP*GP*CP*TP*G)-3') / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 分子量 | 理論値: 1.536035 KDa |
| 配列 | 文字列: (DG)(DG)(DC)(DT)(DG) |
-分子 #5: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 5 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 5 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 構成要素:
詳細: 20 mM HEPES pH 7.8, 100 mM NaCl, 10 mM MgCl2 and 2 mM DTT | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)