+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-25691 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AKT1 K+ channel from A. thaliana in MSP2N2 lipid nanodisc | |||||||||
 マップデータ マップデータ | Sharpened electrostatic potential map from cryoSPARC non-uniform refinement. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | K+ channel / ion channel / hyperpolarization-activated channel / voltage-gated channel / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報root hair elongation / regulation of stomatal closure / response to water deprivation / inward rectifier potassium channel activity / potassium ion import across plasma membrane / monoatomic ion channel complex / response to salt stress / potassium ion transport / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Dickinson MS / Pourmal S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2022 ジャーナル: Biochemistry / 年: 2022タイトル: Symmetry Reduction in a Hyperpolarization-Activated Homotetrameric Ion Channel. 著者: Miles Sasha Dickinson / Sergei Pourmal / Meghna Gupta / Maxine Bi / Robert M Stroud /  要旨: Plants obtain nutrients from the soil via transmembrane transporters and channels in their root hairs, from which ions radially transport in toward the xylem for distribution across the plant body. ...Plants obtain nutrients from the soil via transmembrane transporters and channels in their root hairs, from which ions radially transport in toward the xylem for distribution across the plant body. We determined structures of the hyperpolarization-activated channel AKT1 from , which mediates K uptake from the soil into plant roots. These structures of AtAKT1 embedded in lipid nanodiscs show that the channel undergoes a reduction of C4 to C2 symmetry, possibly to regulate its electrical activation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25691.map.gz emd_25691.map.gz | 290.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25691-v30.xml emd-25691-v30.xml emd-25691.xml emd-25691.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_25691.png emd_25691.png | 164.6 KB | ||
| Filedesc metadata |  emd-25691.cif.gz emd-25691.cif.gz | 6.8 KB | ||
| その他 |  emd_25691_additional_1.map.gz emd_25691_additional_1.map.gz emd_25691_additional_2.map.gz emd_25691_additional_2.map.gz emd_25691_half_map_1.map.gz emd_25691_half_map_1.map.gz emd_25691_half_map_2.map.gz emd_25691_half_map_2.map.gz | 270.8 MB 13.2 MB 285.5 MB 285.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25691 http://ftp.pdbj.org/pub/emdb/structures/EMD-25691 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25691 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25691 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25691_validation.pdf.gz emd_25691_validation.pdf.gz | 822.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25691_full_validation.pdf.gz emd_25691_full_validation.pdf.gz | 821.7 KB | 表示 | |
| XML形式データ |  emd_25691_validation.xml.gz emd_25691_validation.xml.gz | 17 KB | 表示 | |
| CIF形式データ |  emd_25691_validation.cif.gz emd_25691_validation.cif.gz | 20.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25691 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25691 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25691 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25691 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25691.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25691.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened electrostatic potential map from cryoSPARC non-uniform refinement. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Density modification using DeepEmhancer. Inputs are two half...
| ファイル | emd_25691_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density modification using DeepEmhancer. Inputs are two half maps from cryoSPARC non-uniform refinement, using the "high resolution" protocol. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
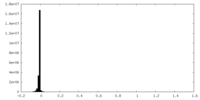
| 密度ヒストグラム |
-追加マップ: Density modification using phenix.resolve cryo em. Input is two half...
| ファイル | emd_25691_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density modification using phenix.resolve_cryo_em. Input is two half maps from C4-symmetrized non-uniform refinement (cryoSPARC). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
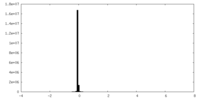
| 密度ヒストグラム |
-ハーフマップ: Half map 1 from cryoSPARC non-uniform refinement
| ファイル | emd_25691_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 from cryoSPARC non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 from cryoSPARC non-uniform refinement
| ファイル | emd_25691_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 from cryoSPARC non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homotetramer of AKT1 subunits
| 全体 | 名称: Homotetramer of AKT1 subunits |
|---|---|
| 要素 |
|
-超分子 #1: Homotetramer of AKT1 subunits
| 超分子 | 名称: Homotetramer of AKT1 subunits / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 969.89 KDa |
-分子 #1: Potassium channel AKT1
| 分子 | 名称: Potassium channel AKT1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 97.109625 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRGGALLCGQ VQDEIEQLSR ESSHFSLSTG ILPSLGARSN RRVKLRRFVV SPYDHKYRIW EAFLVVLVVY TAWVSPFEFG FLRKPRPPL SITDNIVNAF FAIDIIMTFF VGYLDKSTYL IVDDRKQIAF KYLRSWFLLD LVSTIPSEAA MRISSQSYGL F NMLRLWRL ...文字列: MRGGALLCGQ VQDEIEQLSR ESSHFSLSTG ILPSLGARSN RRVKLRRFVV SPYDHKYRIW EAFLVVLVVY TAWVSPFEFG FLRKPRPPL SITDNIVNAF FAIDIIMTFF VGYLDKSTYL IVDDRKQIAF KYLRSWFLLD LVSTIPSEAA MRISSQSYGL F NMLRLWRL RRVGALFARL EKDRNFNYFW VRCAKLVCVT LFAVHCAACF YYLIAARNSN PAKTWIGANV ANFLEESLWM RY VTSMYWS ITTLTTVGYG DLHPVNTKEM IFDIFYMLFN LGLTAYLIGN MTNLVVHGTS RTRNFRDTIQ AASNFAHRNH LPP RLQDQM LAHLCLKYRT DSEGLQQQET LDALPKAIRS SISHFLFYSL MDKVYLFRGV SNDLLFQLVS EMKAEYFPPK EDVI LQNEA PTDFYILVNG TADLVDVDTG TESIVREVKA GDIIGEIGVL CYRPQLFTVR TKRLCQLLRM NRTTFLNIIQ ANVGD GTII MNNLLQHLKE MNDPVMTNVL LEIENMLARG KMDLPLNLCF AAIREDDLLL HQLLKRGLDP NESDNNGRTP LHIAAS KGT LNCVLLLLEY HADPNCRDAE GSVPLWEAMV EGHEKVVKVL LEHGSTIDAG DVGHFACTAA EQGNLKLLKE IVLHGGD VT RPRATGTSAL HTAVCEENIE MVKYLLEQGA DVNKQDMHGW TPRDLAEQQG HEDIKALFRE KLHERRVHIE TSSSVPIL K TGIRFLGRFT SEPNIRPASR EVSFRIRETR ARRKTNNFDN SLFGILANQS VPKNGLATVD EGRTGNPVRV TISCAEKDD IAGKLVLLPG SFKELLELGS NKFGIVATKV MNKDNNAEID DVDVIRDGDH LIFATDS UniProtKB: Potassium channel AKT1 |
-分子 #2: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 2 / コピー数: 5 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-分子 #3: PHOSPHATIDYLETHANOLAMINE
| 分子 | 名称: PHOSPHATIDYLETHANOLAMINE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: PTY |
|---|---|
| 分子量 | 理論値: 734.039 Da |
| Chemical component information |  ChemComp-PTY: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 13 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | Homotetramer of AKT1 subunits in MSP2N2 lipid nanodisc |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 4660 / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)