+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
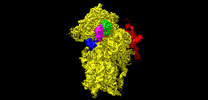
| タイトル | Structure of the wt IRES eIF2-containing 48S initiation complex, closed conformation. Structure 12(wt). | ||||||||||||
 マップデータ マップデータ | Post processed map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HCV / IRES / 40S / RIBOSOME | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報translation initiation ternary complex / regulation of translation in response to endoplasmic reticulum stress / glial limiting end-foot / HRI-mediated signaling / Cellular response to mitochondrial stress / response to manganese-induced endoplasmic reticulum stress / positive regulation of type B pancreatic cell apoptotic process / Response of EIF2AK1 (HRI) to heme deficiency / Recycling of eIF2:GDP / negative regulation of translational initiation in response to stress ...translation initiation ternary complex / regulation of translation in response to endoplasmic reticulum stress / glial limiting end-foot / HRI-mediated signaling / Cellular response to mitochondrial stress / response to manganese-induced endoplasmic reticulum stress / positive regulation of type B pancreatic cell apoptotic process / Response of EIF2AK1 (HRI) to heme deficiency / Recycling of eIF2:GDP / negative regulation of translational initiation in response to stress / PERK-mediated unfolded protein response / PERK regulates gene expression / response to kainic acid / eukaryotic translation initiation factor 2 complex / multi-eIF complex / regulation of translational initiation in response to stress / eukaryotic 43S preinitiation complex / translation factor activity, RNA binding / eukaryotic 48S preinitiation complex / ribosomal subunit / Formation of the ternary complex, and subsequently, the 43S complex / laminin receptor activity / Ribosomal scanning and start codon recognition / Translation initiation complex formation / Formation of a pool of free 40S subunits / ubiquitin ligase inhibitor activity / Response of EIF2AK4 (GCN2) to amino acid deficiency / positive regulation of signal transduction by p53 class mediator / GTP hydrolysis and joining of the 60S ribosomal subunit / 90S preribosome / L13a-mediated translational silencing of Ceruloplasmin expression / phagocytic cup / mitophagy / ribosomal small subunit export from nucleus / rough endoplasmic reticulum / laminin binding / translation regulator activity / gastrulation / MDM2/MDM4 family protein binding / translation initiation factor activity / cytosolic ribosome / stress granule assembly / class I DNA-(apurinic or apyrimidinic site) endonuclease activity / DNA-(apurinic or apyrimidinic site) lyase / cellular response to amino acid starvation / response to endoplasmic reticulum stress / ribosome assembly / positive regulation of apoptotic signaling pathway / maturation of SSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / maturation of SSU-rRNA / small-subunit processome / translational initiation / ABC-family proteins mediated transport / PKR-mediated signaling / spindle / cytoplasmic stress granule / rRNA processing / cellular response to UV / rhythmic process / positive regulation of canonical Wnt signaling pathway / regulation of translation / ribosome binding / cellular response to heat / virus receptor activity / ribosomal small subunit biogenesis / ribosomal small subunit assembly / cellular response to oxidative stress / small ribosomal subunit / small ribosomal subunit rRNA binding / cytosolic small ribosomal subunit / perikaryon / cytoplasmic translation / cell differentiation / tRNA binding / mitochondrial inner membrane / postsynaptic density / rRNA binding / structural constituent of ribosome / ribosome / translation / ribonucleoprotein complex / cell division / DNA repair / mRNA binding / apoptotic process / synapse / dendrite / centrosome / nucleolus / perinuclear region of cytoplasm / Golgi apparatus / mitochondrion / DNA binding / RNA binding / extracellular exosome / zinc ion binding / nucleus / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Hepatitis C virus (isolate 1) (C型肝炎ウイルス) / Hepatitis C virus (isolate 1) (C型肝炎ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
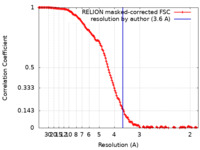
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||
 データ登録者 データ登録者 | Brown ZP / Abaeva IS / De S / Hellen CUT / Pestova TV / Frank J | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2022 ジャーナル: EMBO J / 年: 2022タイトル: Molecular architecture of 40S translation initiation complexes on the hepatitis C virus IRES. 著者: Zuben P Brown / Irina S Abaeva / Swastik De / Christopher U T Hellen / Tatyana V Pestova / Joachim Frank /  要旨: Hepatitis C virus mRNA contains an internal ribosome entry site (IRES) that mediates end-independent translation initiation, requiring a subset of eukaryotic initiation factors (eIFs). Biochemical ...Hepatitis C virus mRNA contains an internal ribosome entry site (IRES) that mediates end-independent translation initiation, requiring a subset of eukaryotic initiation factors (eIFs). Biochemical studies revealed that direct binding of the IRES to the 40S ribosomal subunit places the initiation codon into the P site, where it base pairs with eIF2-bound Met-tRNAiMet forming a 48S initiation complex. Subsequently, eIF5 and eIF5B mediate subunit joining, yielding an elongation-competent 80S ribosome. Initiation can also proceed without eIF2, in which case Met-tRNAiMet is recruited directly by eIF5B. However, the structures of initiation complexes assembled on the HCV IRES, the transitions between different states, and the accompanying conformational changes have remained unknown. To fill these gaps, we now obtained cryo-EM structures of IRES initiation complexes, at resolutions up to 3.5 Å, that cover all major stages from the initial ribosomal association, through eIF2-containing 48S initiation complexes, to eIF5B-containing complexes immediately prior to subunit joining. These structures provide insights into the dynamic network of 40S/IRES contacts, highlight the role of IRES domain II, and reveal conformational changes that occur during the transition from eIF2- to eIF5B-containing 48S complexes and prepare them for subunit joining. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25538.map.gz emd_25538.map.gz | 226.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25538-v30.xml emd-25538-v30.xml emd-25538.xml emd-25538.xml | 64.3 KB 64.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25538_fsc.xml emd_25538_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25538.png emd_25538.png | 38.2 KB | ||
| マスクデータ |  emd_25538_msk_1.map emd_25538_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25538.cif.gz emd-25538.cif.gz | 13.2 KB | ||
| その他 |  emd_25538_additional_1.map.gz emd_25538_additional_1.map.gz emd_25538_additional_2.map.gz emd_25538_additional_2.map.gz emd_25538_additional_3.map.gz emd_25538_additional_3.map.gz emd_25538_half_map_1.map.gz emd_25538_half_map_1.map.gz emd_25538_half_map_2.map.gz emd_25538_half_map_2.map.gz | 119.5 MB 132.8 MB 193.2 MB 193.3 MB 193.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25538 http://ftp.pdbj.org/pub/emdb/structures/EMD-25538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25538 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25538_validation.pdf.gz emd_25538_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25538_full_validation.pdf.gz emd_25538_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_25538_validation.xml.gz emd_25538_validation.xml.gz | 20.8 KB | 表示 | |
| CIF形式データ |  emd_25538_validation.cif.gz emd_25538_validation.cif.gz | 27.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25538 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25538 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25538 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25538 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7syrMC  7syiC  7syjC  7sykC  7sylC  7syoC  7sypC  7syqC  7sysC  7sytC  7syuC  7syvC  7sywC  7syxC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25538.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25538.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post processed map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25538_msk_1.map emd_25538_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Local resolution values
| ファイル | emd_25538_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution values | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Local resolution map filtered at local resolution
| ファイル | emd_25538_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution map filtered at local resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_25538_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map
| ファイル | emd_25538_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map
| ファイル | emd_25538_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : 40S ribosomal small subunit with HCV IRES
+超分子 #1: 40S ribosomal small subunit with HCV IRES
+分子 #1: 18S rRNA
+分子 #36: Met-tRNA-i-Met
+分子 #39: HCV IRES
+分子 #2: Eukaryotic translation initiation factor 1A, X-chromosomal
+分子 #3: uS2 (SA)
+分子 #4: eS1
+分子 #5: uS5
+分子 #6: uS3
+分子 #7: eS4 (S4 X isoform)
+分子 #8: uS7
+分子 #9: eS6
+分子 #10: eS7
+分子 #11: eS8
+分子 #12: uS4
+分子 #13: eS10
+分子 #14: uS17
+分子 #15: eS12
+分子 #16: uS15
+分子 #17: uS11
+分子 #18: uS19
+分子 #19: uS9
+分子 #20: eS17
+分子 #21: uS13
+分子 #22: eS19
+分子 #23: uS10
+分子 #24: eS21
+分子 #25: uS8
+分子 #26: uS12
+分子 #27: eS24
+分子 #28: eS25
+分子 #29: eS26
+分子 #30: eS27
+分子 #31: eS28
+分子 #32: uS14
+分子 #33: eS30
+分子 #34: eS31
+分子 #35: Receptor for Activated C Kinase 1 (RACK1)
+分子 #37: Eukaryotic translation initiation factor 2 subunit 1
+分子 #38: eL41
+分子 #40: ZINC ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.000075 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: OTHER / 詳細: H2/O2 mixture for 25 seconds at 25W power |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: 4 second blot time, force 3. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 4.0 sec. / 平均電子線量: 70.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 得られたモデル |  PDB-7syr: |
 ムービー
ムービー コントローラー
コントローラー

























































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)